lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01002303.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-610.30) is given below.
1. Sequence-
GACUUAAUUUUAUGCUAGGAUUCAAUUGUUAUUGUUUUAAUGUGAUUUAGGGAAUAACUU
GCAGCGUAAAAGGGAAUUUGCUGACUUAUAUGUUACCUGAAAGUGUUCAAGGUCUAAUGA
GUAAAAAAAACCUCAUGUUCUGCUUGUUGUUAAGGAUUUUCUUGUUUUUCUGUUUGUUUC
CCAUGCUUACAGUUAAGUUUUGGAACCUUUAUUCCCUACGACUAUGCUAAUGGAAAGGGG
AAUUUCAGUUAGUUUCACUUGUGAAUAGUCCUUUUUCAGAAAGUCAAGUUUUCCAUGGUU
GAAUUUAUUUCUAAGGGAGAGGUUGGAGAUUUAUGUUUAAAAGUAAAAGUUCAUGUUCAA
UUUGUUCCCAGCAAGUCUCUGUUUCUUGUUUGUGUUUGCAUUUGGCUUCAUUACAUUAGC
UCUCAUCAGUACUUGUUUAAGACUUCUAUGAUGUAAUUACUAAUACAGUUCAGCUUUAUC
CAUGAAUUUAUUGGUCCCUGUUUACCCUAUAAAUGAUAGACUUGAAGGGUAAAUUCCUGU
CAUGAUAGAGUUGAUAGUAUUAAAUAUGGGAUAUAACAUGUGAAGGGAAUUCAUUGAUUU
UGUAUUCUAGAAAUCUUCUUGUUUGAUUCCUGGUGAAUGAGUUCAUAGAUAGGUAUGUCU
AAAUAGGGUAUAGUGCACCCACUACUAAUUAAAAAAAUGAGUAAAGUUAUAUCUUAUGUU
GUUGCAUCUAUUAGUUUAAGAUCCCUGAAAGUUGGUCCUAAUACUCUGUUAAUCGAGUAU
AGUUCAAUACGUGUGGAUGGAUUAGAGUUUAUUAGUUAAGUGGUCUCAUGUUCCUUGCCA
UCGUGAAUUGAAAGAAAAAUGAUGAGCCCGUCUUUCUCCCUUUCUCUUCAGCUGUUCUAG
UAGGCAUUCAAGUUUCAACAAAUCAGUGUUUCUUUUCUUCCUUGUUCACAUAGUAUGCAC
UUGGUGAAAAUCUGAAUUUAGGGUGUUGAUCUUAGCAUUGACUUGCUUAAUGUUUGGUAA
UCUUAGUCACGUUGUCUUAUGCUUGGUCUCUUGUUUGUUUAGGUCUAAGUAAUGUACAAU
CUAUGCUAGUUAUAACUAUAUAUAUAAAGUCUAAUUCUUGAGAAUCUUUUCUUGUCCAAG
UUGUGAUCUUAUGGUUUCAUGAAGUAAAAGGUCAUAGUGUCCUCUACUAUUUGAGUACUG
UUAUGUUUUUUAGAGUAAAGCCUUGACCCUUGGAGAACUAGUUUAUAGUGACUAAUUAGG
UUAGACCUGCUAGAUGACUAAGUUAAAUGUUUUUGAUUGGCACCUGAUUGUUGGCCCUUG
UCUCGGUUUGCAAUUAAUCAGUAGCAUCUUUUAUUCUAAUUUUAUGUCCCCAUUUUCUGU
UUGAAGCCGACUUUUUAUUUAUAGGCUGAUUGUCUUGAUUUAUAUGUGCAAGGUUCUUAA
AUAAAUCUGGUUCUUCAUCUUAUGAGACUUGGUGCAUAAAUCGGUGUAUGUAAGGCCUUU
GAGUUUAUCUAAACUUCAAACCAGUGAUAGCUUGUGAAAACUUAUUAGCGAGUAAUUACU
CUUACAUGAAAUCUAGUUUCACGAAACCAUUUGAUGCAAGAACAUGUUGAUUUCUUGUGC
CCCUGCCCUUAGUUUCUUAAGUUUCACUAAUACUAUGCAUCAUGCAUGUUUGAUCCUGCU
GUUCAAGUCCUUUGUUUACUGGGAAAAUGACAUUUUCUACUUAAGUUGGUUUUGUUUCCA
UGAGCAGUAGGCAGCUGCAUACGUCGAUUCUAUUUUUUGUAAUGCAAUGGCAAUUUUUUU
GAGCAAGUUUAGCCAUUUUCUAAAAGAACCUUUUUGUUUAUAUGGCAAGGUGCAUAUCCU
GUUAGAAGUUCCCAUCACCUGGACUGUGCUGCGAUAUUUGAUGUUUAGGUAACAUAUGAA
UGAUUAAGGUUACGUCCCUGUUGUGCCACUCUAUAUUAGCUUGGAUUUGGAUCCAGUAUU
CUUUAGUCAAACCCCAUAUUAGGUAAAAUGUUUUUCUUGUUUGACUUUGUUCAGAAAAGU
GAUUUUAUAUGUCUUUGCAUGCAAUCUAUGAAAGCUCUUCUAAGUGGAUGAUGACAAUAU
UAGUUAGUUCACUAAUCAUUCUCAUUUUUCUUUGCAUGAACACGUCUCUUCCGAGUCACC
GCCGGAAUCAUCUUCCUUGCAUUGAUUGAGUCAAAGCUCCAUGCCAAGUCCAUUAUUGAC
UGAAGACAAGGAAAGCAAGACAAAAAGGGGCCAAAACCCAUAUAAGGAAAGGCAACAGCC
UAUUAGUCCCAGAAAGGCCAUCUUUUAUUUCUCAUGGGUGGUAAUAGGCCAUAAGGCCCA
AAAUUUUUCCUUUUACUUUAUUAUUCUCAUUUAUGGGCCGAAAGCACAAUAUUGUCAAAA
CUAGGACAGGUACGAAAAGAAUUGUGUCAGUUCAGUUCUCUUCGAGGCGAACAUAUAAGG
AGAACCGGGUCCAAGUGAUAGGCCAAUCUUGGGCAUAAACAAGGCCACCCACUAGGGGAU
GUAUUAUAUUCUUACCUGUUGUGUAUUGUUUAUGUGUUAUGUGAAAAGUGUUACUCUAAU
CAAGGUGAAUCAACUAGUUUCUACUUUGGUACUCCUAAAAUAAAGAAAGUCC
2. MFE structure-
(((((..((((((..(((((.(((...((((......)))).)))..(((((((((.(((...((((((.(((((.(((((((((((((.....(((((((....))).))))...))))))...((((((.....(((((((.....))..)))))......))))))((((..((.......))..))))........
.((((......))))...((((((((....((((((((((...((((...(((...)))..))))...))))))))))(((((.....))))))))))))).............((((.((((((((.((..((.((((....)))).))..)).)))))))).))))))))))))))))...(((((((((((((((..
.(((((((.((((((((((((....((((.....((((.(((((..((.((((((((...((((((...(((((((((((.((((..............(((((((((.(((.........))).))))))))).....)))))))))))))))..))))))(((((((((((((((.........(((((((((.....
......((((.((((.........)))).))))..))))))))).((((((....))))))....((((.......))))..((((.(((.....))).))))..)))))))))))))))))))))))))..))))).))))..)))).))))))).(((.(((((........))))))))(((((((((.((((..((
(.(((((.(((((.....))))).))).)).)))...)))))))..))))))..........((.(((((...((((((.......(((..((((((((((.((((......((((......((((((((.....((..((((((((((..(((....)))..)))))..........)))))....))....)))))))
).(((((........)))))..........((((((((..(((((((((((.((......((((((((.((((.......(((((((((((...............(((.......)))(((((....))))).((((((((((((((((.((.(((...))).)).)))))))))).((.(((........))).)).(
(((.(((((.......)))))..)))).)))))).)))))))..))))......))))..)))))))))).))).)))))))).............(((((.(((..(((((.((((((((((((((((((.(((....))).)))...........................((((.....))))..(((((.......
...(((.....((.((((((((...((((((((............((((((......((....(((((((.(((((..((((((((((((((.((..(((((((((....)))).)))))))(((((.((((((.((((.((.....((((((....)))).((((.((((((..((((((((((....)))).))...)
))).....)))))).)))).....))....)))))).))))))))))).....((((((...))))))......(((((((((((..............((((((...(((.....((.....))...)))...)))))))))))))))))....))))))))))))))........((((.(((((.((((((((((((
((((((.....(((((....((((((....)))))).....)))))..(((.((.((((.(((((.((....(((.((..((((.(((((..(((((((.(((((.....)))))..)))).)))..)).)))))))..)).))).....))...)))))..)))).)).)))...........((((((((...(((((
......))))).......)))))))))))))))))))))).........)))).)))))))))(((((((((.....)))...))))))(((((.(((((((((......)))))).))).)))))......))))).))...)))))....))......))))))........))))))))....)))))))).))...
..)))))))).......))))).))))))))...))..)))))..))).)))))...((......)).)))))))).((((((((..(((((((((.....)))))........))))...)))))))).....((((((((((........................)))).)))))))))).........))))...)
)))))))))((.((((.(((((((........))))))).))))..))))).......)))))).))))).)).))))))))))))...))))))))))))))).....(((((((.(((((((.(((........))).))))))).)))).)))))))))...))))))).)))))(((((((((((.........))
).))))))))..)))))..)))))).))))).
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-663.73] kcal/mol.
2. The frequency of mfe structure in ensemble 2.24507e-38.
3. The ensemble diversity 600.55.
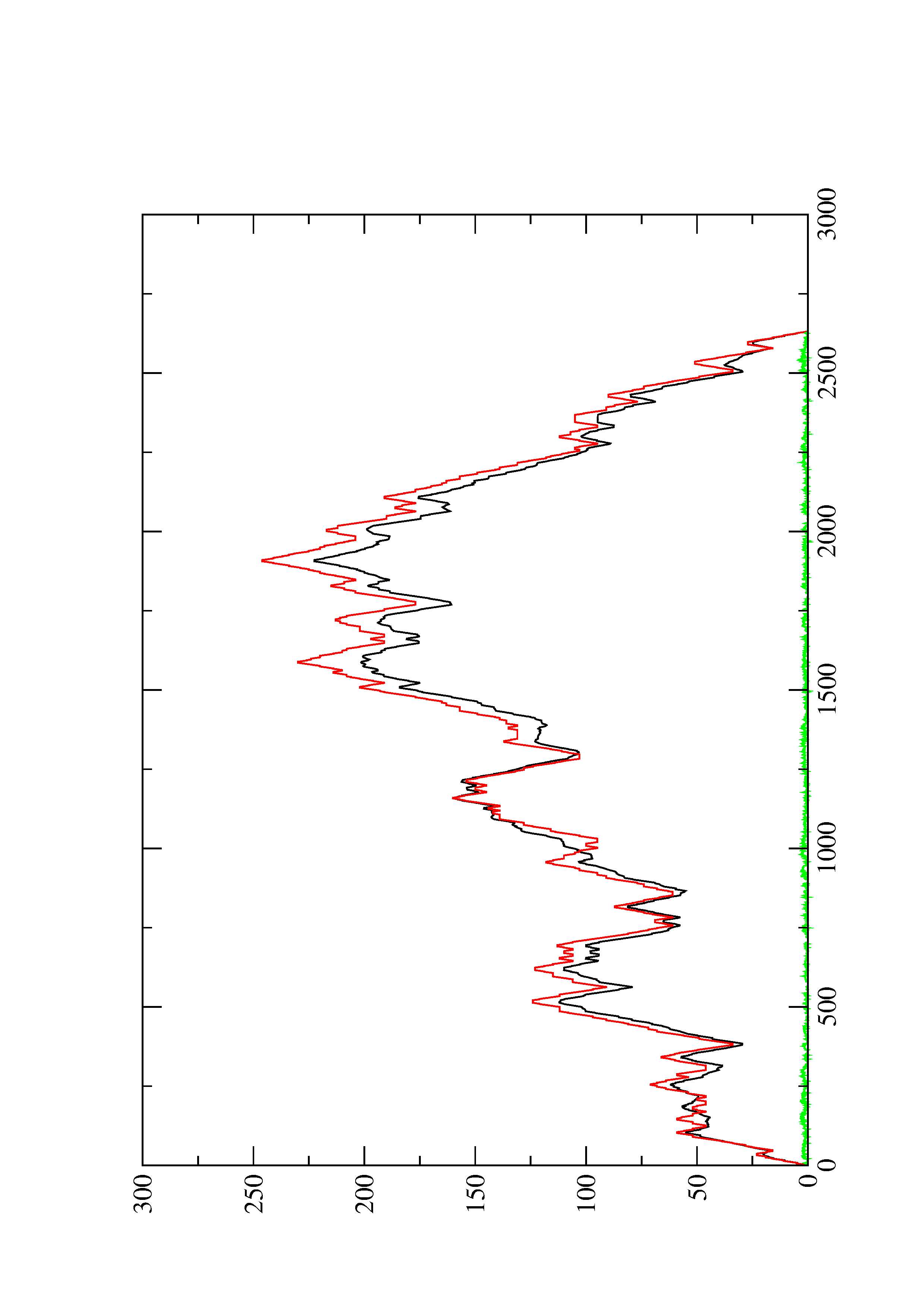
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.