lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01002587.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-634.10) is given below.
1. Sequence-
UUGAGUUUCAGUUUGUCCCCCUUUACUGUCUUUGUGGUACUUUUUGUCUGUUGAGUUUAA
AGAUCAGAUUUUUAUGUUGUCUUUUUAUGAUGGGUAUUCUUGAAUUUGCUGGGCUAUUCC
AUUUUAUGUUGUCUUGUUAUGUUGGAGUUCUUGAAUUUGCUGAGGUAUUCCAUAUUUAUG
UUGUCUUGUAAUGAUGGAAUUCUUGAAUUUCUUGUGUUAUUCUAUUUUGGUGUUGUCUUG
UUAUGUUGGAACUCUUAAAUUUGCUGAGUUAAUUCCAUCUUGAUGUUGUCUUGUUGUGUU
GGAAUUCUUGAAUUUGUUGACUCAAUAGCUCAAUGGGUAUUUACUACGUUGAAUUUCUGA
UUGCCCCCUUACUGUUUUUCUGAUAUUUGUAUUUUCCUAUUGAGUCUAAAGAUUCCAUUU
UUAUGUUGUCUUGUUAUGCUGUUGUUCUUGCAUUUGCUGUGUUAUUCCAUUUUGAUGUUG
UUGUUAUGUUGGAAUUCUUGAAUUUGUUGAGUCAAUAACUCAACGGGUAUCGACUAUGUU
GAAUUUCAGAUUGCUCCCUUUACUGUUUUUGUGGUACUUUCUGCUCCCUUUAUUGUUUUU
GUGGUACUUUCGGCUGUUGAGUUUAAAGAUCCCAUUUUUAUGUUGUUGUCUUGUUAUAUG
CUACACCAACAUACCCAGUAUAUUUCUACAAAGUGGAGUUUGGGAGGGUAGCCUAUGCGC
AGGCUGAUAGACCCUUGUGGUCGGGCUCUUCCCGGAAUCCCACGCAUACGGGGAGCUUUA
GUACACCGGACUGUCCUUUUGUCUUUUUAUGCUAGAGUUCUUGAAUUUGCUGAGUCAGUA
ACUCAAUGAGUAUUUACUAUGUUGGAUUUCAGUUUGCCCCCUUUACUGUUUUCUGAUACC
CUCUUAUUGAGUUUAAAAGUCUCAUCUUAAUGCUGCAAUUCUUGAAUUUCUCAGUCAGUU
ACUCAAUGGGUAUUUACUAUGUUGAACUCCACUUUUGUUUUUUGAUAACCGUCGUGUUCG
GACCAGCUGCACUCACCUCGACUGAUUUCACGAGAUGCGUGGCACUCCCACUAGCAACAG
GUGCUAUGUAAUAUUAUCCACCAAAACUAGGAUAGAUGGGAAGAGAUCAGCACCACCUAG
UGCUGCACGUCACUAUGCCCCGUACUGUUUUCUGAUAUUUCUAUCGAGUUUAAAGGUUAC
AUUUUUAUGUUGCAAUUCCUGAAUUUUUUUCGGUUAAUAUCUCAAUGGGUAUUUCUAUGU
UGGACUCCACUUUGUCCCUUACCGUUUUCUGAUAUUCCUUUUGAGUUUGAACGUCCCAUU
UUUAUAGGAAUUCUUGAAUUGCUCAGUUAAUAAGUCAAUAUCAAUCAACUACUUCUCAAU
GGGUUCAGUUAUAUGAAUCAUAUGUUACUAUUUCACUCGGUCCCAUAUCAAUUCUUGAAU
UGCUGUGAGCAAGAAGUUAGCAAAUGGAGUUUUUGACUUUUGAGUUUAAAGGACCCAUGU
UUAUGUUGCCAUACUAUGGUGGAACUGUUAUUGCUCAGUCAAUAACUCAAUUCAAUGUCA
AUCAACUAGUUCUCAAUAUCUAACAAAGUUGGAAGUCUUUUGUUCCUAUUUCAUCAGGGC
CUAAAACAAUUCUUGUAUUGCUAUGUGUAAGAAACUAGCAAAUGGAGCUCUGAAUUUUGA
GUUUAAAGGUUCCAUUUUUAUGUUGCCUUGUCAUGCGGGAGUUCUUGAAUUGCUCAAUCA
AUUACUCAAUUCUAUAUCCAUCAUCUACUUCACAAGACCAAGCAUAGUUGGGGUCGGCUA
UAUGAGUUUUUUGGUACUAUUUCACUCGAGCCCAAAUCAAUAUUUGAAUUGCUAUGUGUA
AGAAAUUAGCAUAUGGAGUUUUGACUAUUGACCCCUCCGUUGGAGACACAAAGGGAAAAA
GAAAGUUGUAGUCUCUUGAGCUUGAUUAGAUUUUGACUUUUGAUCUGUGUGAAUAUAUAU
UAGUAUACGAUUUUGAGGGAGUAUACAAACAAAACUACUAUGCCUCAGUCCCAAACAAGU
UGAGGUCCGCUAUAUGAAUCUUUACUAUUCCUUGUAAGCUCAACUCAUGUAAAAUUUAUA
AGAGGUAGUGCACGAGAUCGGUCAUUUGUGACACUUUUCCAAUUUGAGAAAAGUGGAAAA
ACUAAAAGAAAAAGAAGAUGUAACUGCUAGGAAAGAAAAUUAAGAUGUACUUUUUCUUGC
UAAUUCAUUCACCUAAUGGUUGAAGUCCAUAAUCUUAAUUGUAGAGUAAAGAUAUCUCAA
UCCUCUGCAUAUCAUAACAGUCCCUCUUAAUCUUCUUUGUCGUUAGCAAAUGAACUUCAC
AUUGAUAUAUGUAGUAAUAUUUUGCCUUUGAUUGUUGUAUAGUUCCCUGUUAAGAUUUUA
ACCACGGUGAGUGUGGUACCACAUCAAUCUUGUAUGUUUGAAAGCUAAGUGAGCAAUUCA
UAUAUGUGAAUAAACUGAAAUAGUUUAUUAUUAGUUCAAUCAACUUAUACUGUUGUUCAG
CAUAACAGUUAGUGCCAUAUUGUAUAAUUCUUAAAACAUGUUAUUCCUCAACAAUCAAGG
CAGAAGCCCCCUACUUCAGCUGGCAUAUGUAACUUAACUUGGAUAGUCGAACUACAUAUC
UUACUUAUCACACAACUACCAGUCAUCUUUCAAAAUUGUUUCCAACAGUGUUUCGGAACC
CCUUCUUGCAGUGGGCAAAUACCAGUUAUUUAUGAUUUAUGAAUGCCAAAUUCAUUUCCA
UCUCAGGUUGAUGCAAUCUCUUCUG
2. MFE structure-
..........((((((((......(((((......(((.((......(((((((((((((((((..((((..((.(((((.......(((((((((...............((((((.(((..(((.((((...(((((.((((((((....((((((...((((.(((((((..(((((......)))))..)))))))
))))))))))..................(((.((((((((...(((((((((((...........)))))(((((((...(((((......)))))...)))))))...((((((...((((((((((.(((((((((....))).))))))....................((((.....))))...........))))
))))))...))))))......((((((.....(((((((...((.....(((((.(((((...((((((...(((((....)))))...))))))..(((((((((((((((.....))))))))))).)))).((((((((((..((((((((((.(((..((.....(((((((((..((((((..............
((((..((((.((((....)))).))))...))))...(((((((.(((...((.....)).))).)))))))(((((...(((((........))))))))))((((((((((......))))).....))))).(((((((((.....)))))....))))(((....(((((((((((((....((((.........
)))).....))))))))))))).....))).))).))).(((((((((((.........(((((((...((((...........))))...)))))))....))))))))))).((((((..(((((((((...............(((((.((.((((((((((((.(((....)))..)))))..(((((((((.((.
.(((....(((.......)))......((((((.(((((....(((((.(((.(((((((((((((.............)))))))))))....(((((..........)))))((((((((...(((((.......((((((.(((........)))...))))))....))))).))))))))(((((((((((....
...(((((((.((.....(((((.....)))))..(((((((....)))))))........((((........))))......(((((.((((((((((((.(((((.(((..(((.((....)).))).))).....((((.(((((((.(((((((.(((((((((.............((((((......))))))(
((((..(((.........)))..))))).(((((..(((((.(((((.(((((..((.(((((((((((((((((((((..((((.(((((((((((((..((((....)))).))))).(((((((((((((..((...(((......)))...)).)))).))).)))))).....((((((...)))))).))))))
))...........((((((((((..((((...)))).((((((.((.......)))))))).))).))))))))))).))).))))))))))))))....)))))))))))..))))).)))))..)))))((((((...(((((....((((...(((....(((........)))..........(((((.((((...
....(((........))).......)))).))))).((((((.(((((...((((((((.........))))))))...))))).))))))...((((...))))........)))...))))....)))))...)))))))))))).))).))))))))))).))).)))).(((((....))))).))))).))))))
)))).))...)))))......(((((((...........)))))))..)).)))))))...........)))).)))))))...)).))).))))).....))))))))))).((((...((((....))))...)))))))...)).)))))))))......................))))))))).)))))....))
))))))).))))))...........((((((((...(((((((((((............(((((((....(((......))))))))))....(((((((((..........((((((((.....))))).)))....((((......)))).((((....))))....)))))))))((((....))))...)))))))
)))).)))))))).)))))))))((((..........)))).))..))).)))))))((((....))))((((((((...))))))))(((.((((.....)))).))))))..))))))))))))))).)))))))....)))))))......)))))).......))))))..))))))))..)))....))))))))
)))))....)))).)))..))))))))).............))))))))).)))))))..))))))))))..))))).....))))))......)).))).......))))))))))))).............(((..((((((.....))))))....)))..((((((...))))))......
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-680.15] kcal/mol.
2. The frequency of mfe structure in ensemble 3.52417e-33.
3. The ensemble diversity 876.48.
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
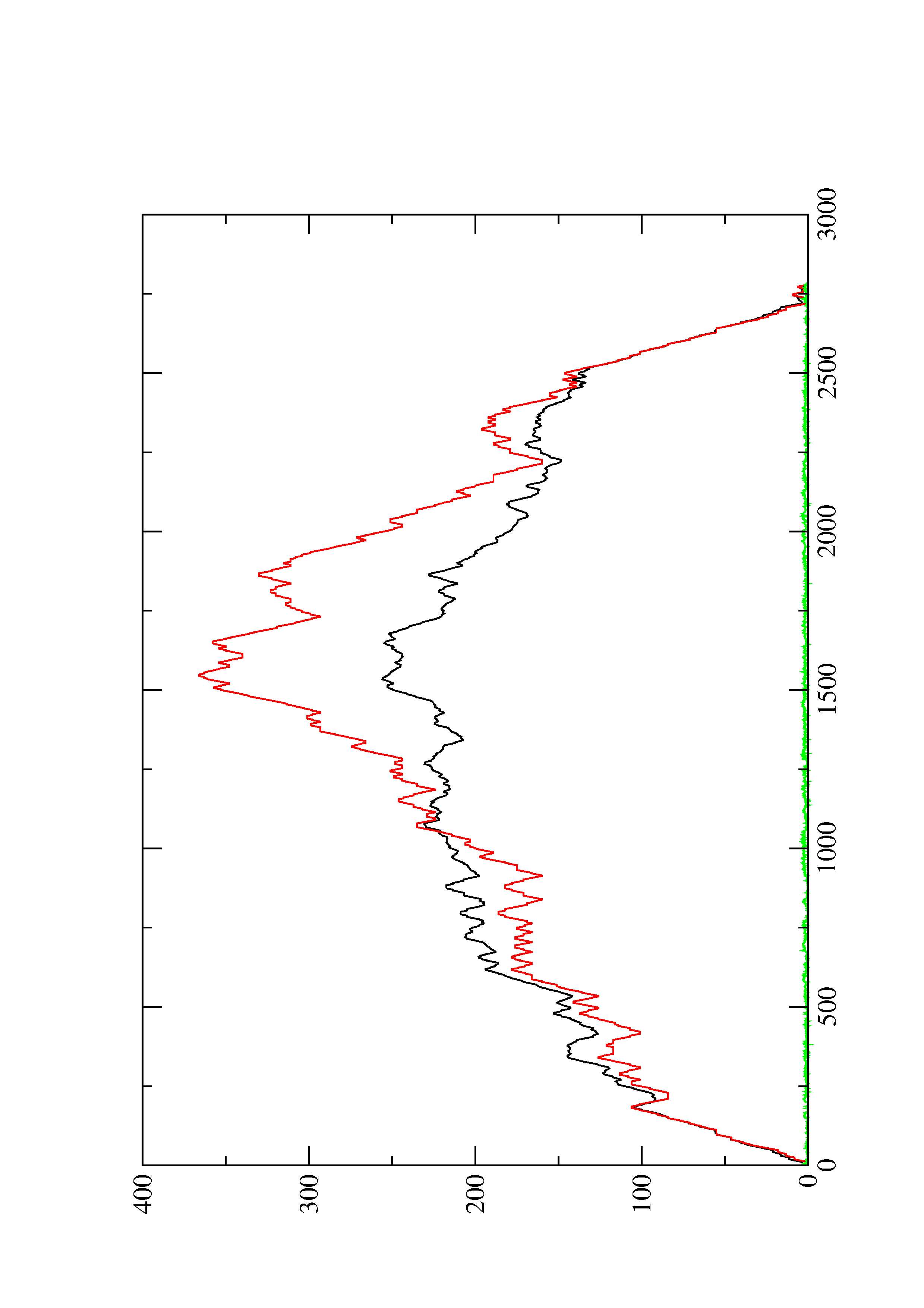
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.