lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01003018.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-570.60) is given below.
1. Sequence-
UAUCAUUCAGAGUAUUUUGAAGUUUUUUUUUAAAUUUUGAAAUAGUUUGACUCUAAUUUA
UUUAAAUUUUAUUUUGCCAAACAUACUUUCCUUCAAUUAAAGAGGGAAGUAAACCUUGUA
UUGUUUCUUGAUAUGAGGUCAUGGGAAGGUGUGGUAGUCAUCUUGUUAGUUUUCUAGUCC
UUUUUCCCUAUUUUGUGGUUAAAAUCUUCAUUCGAGAAAAAAUGAUCACAAGUGGGAGAU
AUAGUAACAUUUUGAAACAUGUGCUAGAGGUUGUGCGAGCUAAAUAAAGUAUGCUGGUGA
AAUUUCACCAAAUUUUGGCAGUGGGCUUGUCUAAUAGAUCGUUCAAUGAUCCGUAGUCCA
GUCGGACAGACCAUGGAAUGGUCAGUUAAAUCAAACCAGUGAUGCACAUGCUCUGCCUAC
GUUAGACGAAUAAUGGGACAAACUGUGGUCUCGUUCGAUAGUCGGUCGUUCCUUUCGUGG
UAUGGACCUAAAAUCCUUAAGAGUUUUUCGUAACGCAUAGAUCAACCCCAUGAAUUGCGU
AACGGAUUGUGGAAUGGGUGUGGGCUUCGUGGAAUAAACCAUUGACCCUUCAACCAUCAA
CUUGGUUGAUGGACCGUUGACCAUGGAAGGUGACCAUUUUUUUGUUUAAUUGGGUUUUUC
CAAUGACCUUUUAGAAAAUCUUAAUCCAUUAAUUGGUAUUUCCCAUGUCUUUAGCUGGUG
GAAAAUAUAAAUACCCAAACUCUAUUUAUUUAUUUCAUUAAUCCAUAAGAUAUUAACGUC
AGAUUCUCCAAGGCUUUCCUCCCAAGAAGAAAGCCUAAGGUUUACAGCAAGGGGUUAUUG
UGCAGCACUUUCUCCACAAAUUUCUUUGUAGAUUCUCUCUAAUUCAUGUAUGUGGCAUUA
UGAACAAGGUGUCAUUCAGUCUUGUACCCAACUACCCAAUAUUUUGUGUCAUGGUCAUGA
UCUAGAGAUUUUAGGGUGAAUUUUCAUAAUUGUGUUCAUGUUAAAAAUGGUGUAUUUUAU
AUACUCUUGAUCAUAAAUCAUCUUAAUAUAUGUUUUAUAAUAGAAUCUCAUAAAAUUACU
AAUAACUAAGAAGCAUGAUAUGGUUCAUAAGUUCAAGUUCCUAAUUUGAGUCUAAAUAAA
UUCUUUUUCAUGUUUUGGGGAUAAGGAUACCAUAAUAAAGGGUUAAGUUUCAUAGUAUGG
UUAGUGUAGGGUUAGAAAUCACGAUUCAUGGUCAUAGUUAGCUUUAUGAAUUAUAAAUUA
AGGAUUUUGGAUUUUAGAAUCUAAGUCAAGUUCAUGGGUUAGUUUAAUUGAAAUAACGUU
AAAAUAUUAGAAUGAGAUAGCAAUACAACAAAGUCAGUAUGUUUAGUAAGUUCAUGAGUU
CAUGGGCCCUUAUGUAGUCAAGGCAGACAGUAUUAGUUCAUGUUCAGUUCAUGGUCAGUU
UUGGGAGUAGUAUUAGCACCGAACAGGUUUGUGUGACUUAGUCCUUUUCCAUAACCUUGU
GUCAUCAAACACAGGUAAGUUCAUGGCCAAAAGCAACAUUCAUAGUUUAUGGAUUUGUGG
AUCCAUUAGGAGAUAAUUCAUGGCCCUUACGUUGGUAGGUUUGGAACCCUACCCAGCAUA
GGAAUAUAUAUUGGAGCUCUAGCCUUGUGCUAGAGGAAUGUCGGUUAUAUAACCUCUCAG
GUCAGUCAAGCGUCAGUGAGUCAGUAUAGUCAAAAAAAAGGUCAGAAAGGAGAAUUAAGU
AUAAACAGAAAACUUUGAGUAAUUAUUGUCAUAUUGCAUUUGUUUAUUUAUUGAGUCAGC
AGUUGACAAUUGAGAUGGGUAUCAUGUGUUCAUGCAUUGCAUAGAUAAUUUCCUUUUUGU
GAUAGCAUGUUUGCUAACCUCCUACAUAUUCGGUAUAUUCAAUGUAUUGACCUGUGCUUU
CUUGUGUUGUACGUCUUUUACAUAUGUAGAUUCUGCCAGUCAGCCAGACGUUUGCGGUUG
ACUUGGAUCCUCAUUUUUAGAGGACUAGAAGUAAAGUUGUGAGUUCCCACCUGAUUCGGG
AACCUGCUGGAGUAGUUUUAUUGUGGUAUAUUUUAGAUCUUAGAUUUUUGAAAUUGUACU
AUUAAUAUGAUUUUGGCCAGCUGGGGGCAUUGUCCUAGUUUAUUAUUACUUGGCUUAGUA
GUGUCUUUUAGGCAUAAAUUGUUUUAGCUUUGGGAUAUUGUUUUUUCAUUAUUUAUUCAU
ACAUCCUUUGAUUGAUGUGAUGUUCACCAUUU
2. MFE structure-
......(((((.(((((((((((((......))))))))))))).)))))....................(((((.(((....((((((((((........)))))))))).(((((((((((.....)))))))))))..))).)))))(((((...((((..(((((((...........((((((...(((((((((
(...((((.....)))).....)))))))))).))))))..(((((.(((........))))))))((((.((((.(((.((((((........((((((....)))))).((((((((.(((((((((.((..(((((...((...((((((.(((((.....(((.((((((...)))))).)))...........((
(((......(((((((((......(((....((((((((........)))))))).....)))(((((..((((((((((((((((((((...(((.(((......(((((.(((((....(((......)))..))))).)))))))).))).)))))..(((.((.((((......)))).))))).....(((((((
(...)))))))).))))..)))))))))))))))).............((((((....))))))((((..(((((((..((((((((.(((..((((((((((((((((((....((((((((...(((((((.........)))))))...))))))))......))))))).......(((((((...((((((((.(
(....)).))))))))..((((((...((((((..(((.(((((.((.....((.(((((....))))).))...((((((..(((((..((((((((.....((((((((.....)).))))))....................))))))))..)))))..))))))((((((.((((((...((....)).)))))).
)))))).)).)))))...)))..))))))....)))))).......((((.....))))..))))))).....................(((((((((...((......((((((((((...)))))))).)).........))...))))))))).))))....)))))))...))).)))))))))))).)))...))
)))).))))))).....)))))(((((((((............))))))))).........))))).))))))...))))))))).)))))))))..((((.(((....))).))))))))))))......((((((.(((((((...((((((((((((((((((...(((((((....)))))))((((........)
))).(((((.((((((.(((.((...(((..(((((((((.((((((.((.((((((((((((....)))).))).)).))).)))))))).)))(((((((......)))))))........))))))..)))..............(((((((....)))))))(((((.......((..(((((((....((((((.
.......)))))).((((((((...((((((((((.(((((((.....)))))))((((((.((((....))))....((((.((.(((((((..((..((((...................(((((((((((............((((....)))).......(((((((.(((((...((((((.....)))).))))
)))))))))).........((((.(((((.........))))))))).))))))))))))))).))))))))))).)))).....))))))......))))))))))...))))))))..(((...((.((.((((((((...(((..(((((....((((((((..........)))))))).)))))..)))...)))
))))))).))...)))......((((((.........))))))..(((((...((((.(((((((((((((((((((.........))))))).))))))).))))).))))....))))))))))))..)))))))..........)).))).)))))).))))).))))))))..)))).....))))))...)))))
)).))))))))))))))).)))).)))).)))))))..))))...)))))..
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-612.85] kcal/mol.
2. The frequency of mfe structure in ensemble 1.68437e-30.
3. The ensemble diversity 622.30.
4. You may look at the dot plot containing the base pair probabilities [below]

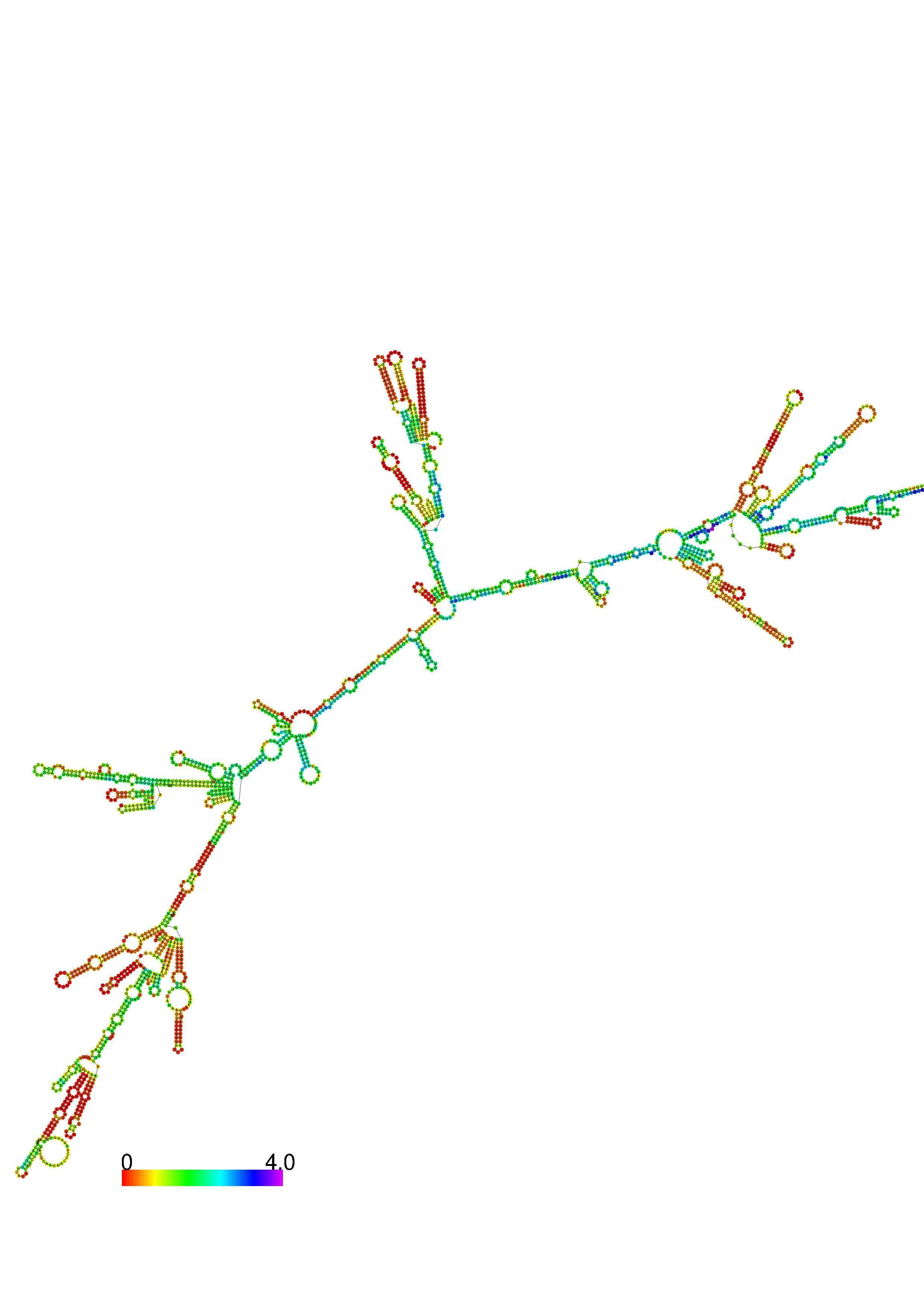
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
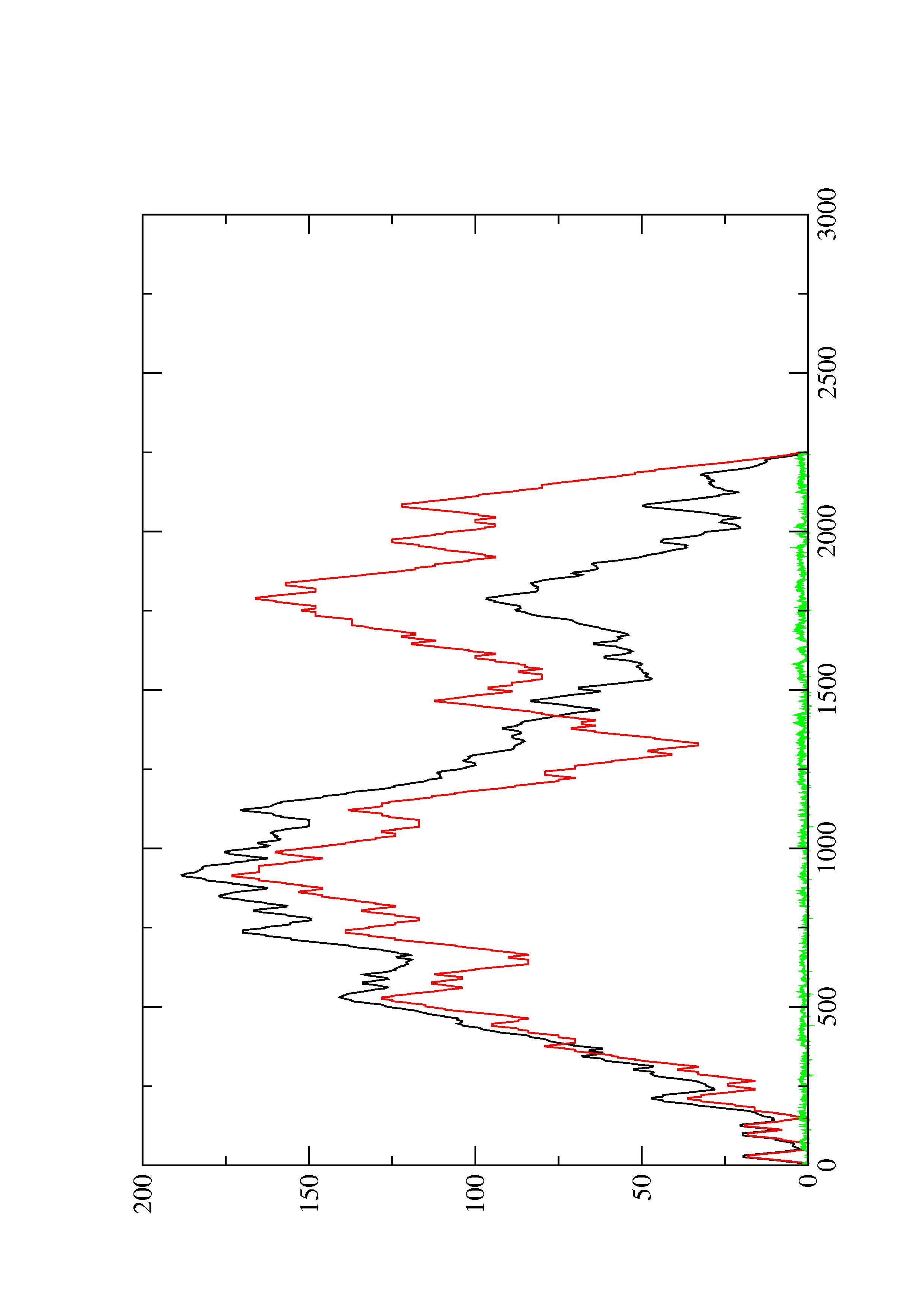
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.