lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01005619.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-697.60) is given below.
1. Sequence-
AAACUGGAAAAUUGUGUGCUUUUGAACGCAUUUUCUGAAUUUUCCAUAAUUUUCUUGGAA
UUAGAAAAUUUAGAAAAUUAGGAUGGAAAACUGAAAAUAAUUUUCUGUUAAUGAACGCCA
CCCUAAGAUAGUCUAAAAUCAAUAGAAAUCCACCCCAAGCUUUUAGGAAUCUCAGUUCCU
UCCCCAAAAUACGUGAGAAACAUUCUAAUUCGUACACCAUUUUAACACUUCCUCUGUGAA
UUGGAAAUCUUGAUUAUAAGGUGAUUGUUUGUUGUUAUUUUCUUUUCUGUUAAACAGUUU
UAAGUAUGAAAUCAUGCAUGUUAGGGGGAAAUAAGAUUGUUUAGAUUGUUUUGGCUCUCA
UAAUUUUAUCAUCUAGAUGCUUUGGUUAUGACAACGUGAUAAAACCCUCUAUCUUGUUUC
UUCUUUAAGCUAGCAGUAAGUAACUAAAGAGUUUCUUAGCCAUAAGUUAUUUAUUUGCUC
ACAUAGUAAUGUAAGUUUGGUUAAAAUCUGACUAAUCUUGGUUUAGUACACUGACCUGCU
UAAUAUUUGCUUGCUUCAACCAUGUUACUUUAUUGUUUAAAGGUAGAGUCUUUCGUGAUU
AGGUCCUUUGCUUGAUAAAUAUGGCUCAUUAUUGAUGAUAAUGAGUGUCAAGUUUUUUGU
AUGGUCUUAGGAUGUCAAUUGGAUUUCACAUAUCUAAAUGAGUAUGCUGCUAAUAACCUC
UGAUAGGCUUUGCUUCCUCCAUCUGGUUCUGGUAGCAACAACAGGAAUCUUUGUCCCUUU
UCUUUUUUAGGGGUUUCAGGUCAUCCUAAUAAGUUAAGAGUUUGUCCAAGCCUGUUUAAG
UGAAGUUUUAUUGAUCUAUGAAAGCUGGCUCAGCUGAUUAUUUUCUUAAUCUUAAAAUCU
GGUGUUUCAGUCUUUAAGUGAGAGCAUGUUUUAUUUAAGCAAGAAUGAGGAGUGAUUGUC
AGUCUCUGUGAAUUCUCUUGGCUUGUGAGGUCGUGGUGCAUAUUCAUGAAAGAUGCUUCA
AUGAUACAUCUUGUCCUCUAUCUGUCUAGCUUACUAACUGGUUCUUGUCUCACAAGGCAU
AUAUAAGGUUUAAGUGCUGUGAAGGUGCUGAUAUUGCUUCAUAUAGUUUAGUAUAGGGGG
UAGCUAGCAUAUAACUUGUCCUUGCCUUGUUUCUCCAUGUUUAGCUCUUUUGAGUUGGAU
UUUAUAUAUGUAUUAGGUGUUUUAUUCAAAGUCUGGCAGUCUAUUAAGCUUGGUAGAGUA
GGAUUCUCUGGUCCAUGUAUGGUUGCACAAUCAAUCUUUGGCAUAUCAUGUUGCAUAAAG
UUUUAGUAACUAAAAUCUGAGUUUUUUAUAGCAUGUUCUCCUCUAUUUGCUUUUGUUAAC
CUUUAUUUUCUUAACUACUCGGUCCACAUGAGUUUUGAAUGAAACGGGAUUAGGUCUUGA
UGGUUUCUUUCCCCUGUUUGUGAAACAGUAUGCUGGUUUCGGCUUGUAAUAGGUCUUGAU
GGUUUCUUUCCCGUUUGUGAAACAGUACCCUGGUUUCUGCUUGUAAUCAUUUAGACUUAA
GAGAACUAGGCAGCCAGGUUUAAAACCAUAUAGACAGCAUGCUUAGGGGCUUAAUUGGUC
UAAAUCUUUGUCCUAUGUAUGAAUAAUCAAUUGUCUUUAAAAUGAAUCUGGGGUCUAUGU
GCUCUCUAUUUUAUUCAAACCCAUUGACAUGAUUAAGUUGGUUCAAUUAUAGUCUGUUAG
GUUGACUACCAAAUUCCUAAUCUUUAUAUCAAUAAAUGAAGUCUAUUUAUGGUCUGUUUG
UUGUUUGCUAUUUAAUUCAAAACUAGAUGAUUUUUUUAGUUCCUGGGGCUUUCUGGGUGA
UUUCUUUUGAAAAUACCAUGCAUAUUCAAUUAGAAACCGAGUAUGUUUGAUACAAGAUCA
CAUAGCUUGUGUAUCUAAAGUUGUUUGUCUCAACCAUGAUACUCCUUGUUAUAAAAGGUG
AAGUCCUGUUGUGGUUGAUAUACUGCUAACUGUUCUUUAUCAUUGUUUCAGAUAGUUUUA
AAUGACUAAUAGAGUCCCUACCUGAUAGAAUCAUUCAACAUCUAUUUAGAUUUUAUUCCA
CCUCAUUCCUGUAUGAAUCAGUUGUUAAAAUUAAGAUUUUAGAUAAAACCUUGUUCACUU
GGGCACUAUAUACAUGUUCCCUGUCUAAAGGUGAGAAUUUGUUCCCUAAUGUUGAACCAG
UAUAAUGGUGAUCCGUUAACCUAACCCUGCCUGCUAUGUAACCUGCAUGAAGGAUUACAC
CGACCAGGGUUCUAAUAUGCAUACUUAGAAGAUGAUUAGUUUGGCCAGAAUUUGUUUGUU
UAACUGUAUUCAUGUCUCAUAGAAUGUCUCUGGUCUCCUCAAACUUCAUUCUUUACUGCU
UGUUGCUGAUCUUUUAAAGCAUCGAGUGUUAUGAUCUGUUUUUUGUUUGAAUGCAUAGCU
UUGGUUGAAAUAUGUUCAUACUAAUAAAGUUUACAGAUGCGGAUCAAAAAUAUUAAUGUG
AUUAUUUUUAUAUUAAGUUGGUAUCUAAUCUUUAUCAUUUUAUUGUUGCAUGUGAUACCC
AUUACGGACUCUCUCCCUCUCCCUCUUCGCAUUUGAGUCAAACCUCCAUGCCAAGCCUAG
AUGGGCUGAACACAAGGAGCUGGAAGAAAAUUAAAUGGGUUGAGACCCAUUAGUAUUGGC
AGCAGCAGUGAAGUAAAAAAAUGGGCAGGAACCGAUUGAUUUAUUUAGUAGCAGCAGCAA
AGAAGAAGCUUAGUAAGCCGAAACCCGUUAAGUAGUUAUAGUUUUGAUUGAAAAGGCCCU
UUUCUGUUGAUUUUUGUUUGGAAAAAUUACCGAACAUACUCAAGAUAAGAUCAAUACUUA
CAAUAAAUGACAACACUAGUAAAUCUAUAUCGAUUAUAAAAAUAUGUGAAGGCUUUCAGA
GAAGUGAUAUUGAACUUUUUGUCCAUU
2. MFE structure-
....((((.....((((........))))(((((((((((((((..(((((((...))))))))))))))))))))))...(((((((((..........)))))))))..((((.(((...(((((((((((((((..((((.........((((.(((....((((((....)))))).........((((..((((.
..))))....))))..(((.((((.((.......)))))).)))..((((((....))))))(((((((((...................)))))))))....(((((....)))))..))).))))........))))))))))))))))))).((.((((.((((((.((((((((((((.((....(((((...(((
(........)))))))))....))..)))).((((((((((((((.....(((....)))....))))))))).))))).....(((((..(((((..(((((....(((((((....))).)))).....))))).)))))....)))))((((.......(((((.((((((((..(((((...((((......))))
....)))))(((((((.......((((((((((...))))))))))))))))).....(((((((((((.........((((((.......)))))).((((((((((((((..((((...((((((........)))..))).)))).)))))))).(((((((((........(((((((...(((((((((((((((
....))).((((((((((((....((((....(((((((((((((.(((((......)))))(((((...)))))...(((.((((((..((((((..((((....))))..)))))))))))).))))))))))))))))(((.(((.((....)).))).))).)).))...))))))))))))((((.((.((((..
...(((((((......))).))))..)))).)).))))..........((((((((((((((((((((((.((((((((......(((..(((((.........((((((...(((((((.((((......)))).))))))).))))))...))))).)))))))))))........(((((((((....)))))))))
.........((((...((((((((.((((((.((((((.............((.(((.(((((((((.(((..((((((((((((.((((.(((..((((((..(((((..((((((..((((((....(((((((((.((((((..(((((..((((..........((((.(((..(((...................
)))..))).))))...((((((...(((((((((..(((.(((((........((((((...))))))...((((....))))......(((((((.(((((((............(((((((((....)).))))).))....))))))).)))))))((((((((((((..(((((((((.........(((((((((
((.(((((.....(((......))).....))))).))))).........))))))......))))))))).))))).))..)))))..........((((...(((((.((((((((((...))))).))))))))))))))))))).)))...)))))))))..........(((((((((((...((((..((((..
..((((.............))))..(((((((((.((((.....(((((((((((((((.((((....)))).))))..((((((((...........)))))))).(((((.((((.((..((((..(((((................((((((((((((((((((.......))))...)))...)))))))))))))
))).))))..)).)))))))))......))))(((((......)))))...)))))))...)))).))))))))).)))).)))).))))))))))).....))))))......))))..))))).)))))).))))))))).))))))((.(((((((((((........(((.(((((((......)))...)))).)
))..))))).)).)))).))..((((((....))))))....(((((((..((...((((((((......))).))))).))..)))))))....))))))..........))))).......))))))..))).)))).))))))))..))))..))).)))..))).))).)))))((((((..((.((...((.(((
((.((((..........)))).)))))))...))..))......)))))).))).))))))))).))))))))....))))..((((((.....((((((.(((........(((((((((......)))))))))))).))))))...)))))).(((((((((((((.(((.((((((.....))..(((.(((..((
((......((((..(((......))).))))..(((((....)))))........))))..))))))......(((((((....))))))).))))..))))))))))))))))........))))))))))).(((((.....))))).((....))........)).))))))))).))))))).))))).(((((..
....))))))))))))....))))))))).....(((((((........)))))))))))))...))))))).))))..))))))))))))).....)))).)))))....))).)))))).)))).)).))).))))(((((((........))))))).))))..
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-751.33] kcal/mol.
2. The frequency of mfe structure in ensemble 1.38732e-38.
3. The ensemble diversity 851.16.
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
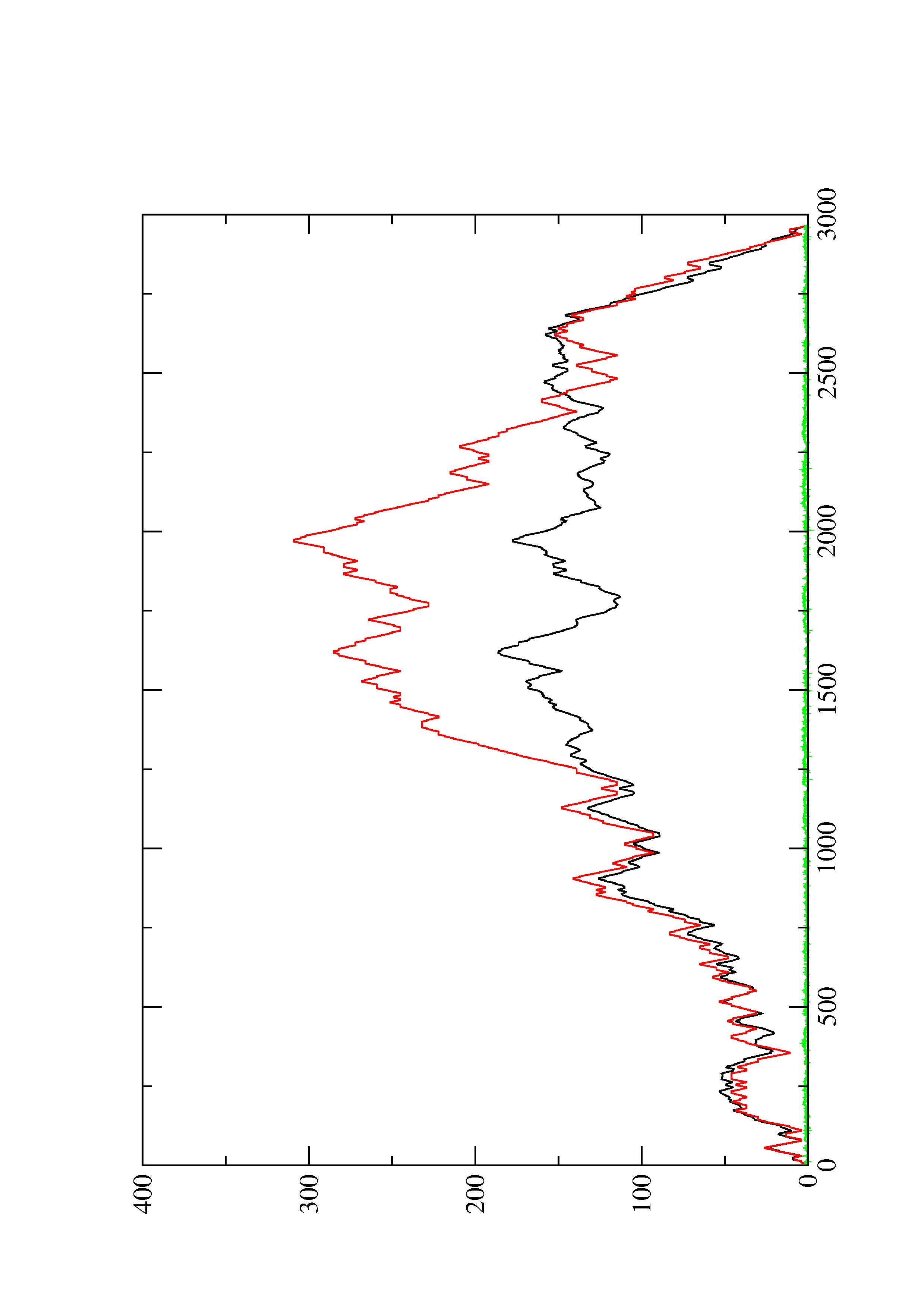
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.