lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01016544.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-670.90) is given below.
1. Sequence-
CGGAGAAUUGACUGAUUUUAGAUUUGAGGUUUGGUUGUUUUUGGUUAAAUUGGAUUUGAU
UUUUGUAUCAGAAUUAUAUUAGGGCUUUUAAUUCAAAGAACCGGAGAACUUCUUGAUUUU
AGAUGGUGAGGUUCUGAUUUUUUAAGGACUUGUUCAGUUAACUGGAUUCGUGUUUUGCAG
UAGGAGUCAAUGUUAGGGCUUUCAAAUCGGAUGAGUGUUUGAGCUUAUGCAUUAAAUUGG
AACUGACUUUGUUGUCUGAAGUCAAAUUAGGGCUUAUUUAAUUGGAGUUGUUUGUGUUUA
UGUUUUGAGAAGAUAUUUGUUGGGGAUUAUGUCUGUUUUUAGUGUUUAUUGAUAUUCAAA
UUGGGUUGUAUUUUGAAAGGGAUGUUUUUUCAGAAGAAGUGCUUGAGCUAAUGUUUCAAG
CUGAAAUCUUUUGAAAAUUCCCGAACUUGUUGGACUUGGGAAGUUAGAAGUGCUAAUAUA
UUGUUGAUCAUGUUUCUUUCAUCUGAUAUUUUCUUAAUAGUGUCUGUGUAUUAAGUUGAU
GAAGGGACACAUGGUGCACAUCAUAUUAUUGAAAUGCUUAAAGAUAAGAACCACAUGUUU
ACUUCGUUUUUUAUGAUUGAUUCUUGAUCUGUGAAUGUUGGUUGUUAUGGGGUUGUUUCC
UGGUGGAUAAAUCUCAUUGGUUUGAUUGUGUCGACUUUAUCAUCUGUGCAAUUGUUGGUA
UAUUAGUGAGUUGGUACAGAGAAUUUUUUGAGGACAGUAAUUUGGCAACUUCUGGUUGUA
CUUGGCAGAAGUAGCAUAACAAGUUUUGAGCUUUUUAUCAGAUAUUUUGAGGAAGAACUA
AACUUUUGGAGUGCAAAUUAGUAAUAUACGUCAAAAUUUUCUCAAAUGGAUCAGUGUCAG
UAUGAAAAGAUAAAGGUUGAUUUAGAAUUUUCAGUUGGGUUUUGAAGCCUCAAUAUUUUA
UAGAGCUUCAGAUUUUGCUUAUGGUGUCUCACAUAUGAGGAUGGAUGCUCCUUUGGUUUG
GGCUGAUACUUAUGAAAACAUAGCCGCGUUUCAUCAGGCUCCAAGUGAGUUGCACUUGUA
AUACCAAAGAGUUGGGUGGAAACCAAAGUUCUGUCUUAGCUGCAUGGAUGAGGGGACUUA
AUAUCUAAUGGAGAUGCUUACAGAUGUUAUGAUCUAUUUUUUGCACAUCCUGAACUAAUG
UGAUGUGAAAAUUUUCAUCAGGUUAUAUGAGUGACCUAUCCAUCACUUGAAGUGAAUGUG
UAUCUAUUGGUUGUGACAAUAUCUGAUGGGUAAUGUAGUUGAAGUUCCAACCAGUUUAUU
UGGAUUAGGUGCAACUAGGUUGUUCAUUGGAUCUGGAUAAAUUCAUCAGGAGAUGCUUUU
AUUUGGCUAAUAAAAGCCUUUUCAUCUUUGAUAGUACUGGAUUCUCUUGUUCUUUCUGUU
UUCCAUGUUUUGCGAAGUUAUGAGUGCGUGGCAAUGUACACAAUGAUGAUAUUGAGAUAU
UCAUGUAGUGUUGAAUACUAAAGAUGUACAUCAAAUGGUUUCUGUGUAUUCAUUAUGGGA
ACUGCAGUUUAUAAGGUUCCCUUCCUGAAUUGAAUUUUUCCAUAAAUAAUUAAUGGGUAU
AUGAUCAAAGAAAGAUUCAACAGGAUUAGGAAUGAUCACAUCAGAUAGAGGAUGGAAAAA
UGAGAGGAUUGUGGAGAAUGGUUUGUUCAUAAUCUAUGUAAAUCAGAUGGUUACCAGUUC
AUUUGUGUGACAUUGCGAUGAUUGGGGAUGUACUAAAAAGAGAUGUGAUGGACAUAAACU
ACCGGCUGACAGACGCAUGAUCAAAUGCAAAUGGUACAUAAUGUAAAAUAUAAUACGCAA
UACCUACUAGUUGUGUUUUAUCUUUAGUUUCUGUUGUACUUGUAUUAGAUCUGGUGCUUG
CGAGUUGCUUGAAUGAAGCUAUUUUUGUGUCUUUGGUUGGUUAGAGAUCGUAUCUCAGUG
UUGGGUUAACUAUGAGAAAUAAGAACUUUUUGUCUGGAAAGCUCCUAUUGCUCUUGUUGG
AACUUAUUUAGACGUAACUUUUGGGAAAAAAAUAUUUACUAGUAGUGGGUGAAAUGGACC
AGAAUAAAGAGGGCCUUGUACAUAGCAUUCACUAGUUCCCAUCUGACUAGGGAUUUCAUG
CUUAGCUGACUGUUGAUUGUUGCAUCUUUUCCAAAUUUUUAGCUGAGAUUGAUAAUUAUA
CUGUUUUAGCGUUUUAUGGGAAGUUUUGGUUCUAGCUUAGUUUGUGUUUUUGUCAUACUU
UAGUUGCUUGGCCUUGUAUCUAAACUGAUGCUCAGUUUUACAAAAGGAUCUGGCAGUUAU
AUGUGUCAGUAGUUCCUGAUUUAUUCGGGAUGUAUGUUAAAUUGAUCAGAUGAUGUAAGC
CUAAAAUAAUACAAUGUUACUGAACGAGCAUUUUAACUGUUUGGGUGGGGGCACGUGAUG
UUGACCCAUGUAUGCCUUUAUUCUCUUAUGUACUUGUAGGGGUUGUAUAGACGUGUUAAU
UCAUAUUUUGGUCUUUAAGGGAAGCUUCUACAUGUAUUCUUGUGUCAGCACUGCUAUCAU
UUUUUGUAAUUUGACAAUUGGUUCUAUAUUCUUAUAGAAACUAAACAUGCCUAAGGAGAG
GAGAGAUAGAUC
2. MFE structure-
.((((((((.((((((((...(((((((((((((((.((((...(((((....(((((((.......(((((((((((((((.((((((((((((((((((((((.((((((.((((((((((((((..(((((((((((.(((((.(((((((((((((((..(((((.((........)).)))))...))))..)))
........)))))))).)))))((....))...))))))))))).))).....)))))))))))(((((((.....))))))))))))).)))).))))....)))))((((((((((.((..(((.(((..((...(((((((((....)))))).)))...))..))).)))..)).))))))))))((((((((...
((((((((....)).)))))).....))))))))...(((((((.((....))..)))))))))))))))).)))))))).(((..(((((((((((((((((.........(((((((.........)))))))..)))))))))).)))))))..))).((.(((((((.....(((((((((((.(((((.((((((
((.(((....(((....)))....)))...))))))))))))).))))((((((...(((....)))..))))))...(((((..((((((((((((.......(((((........))))).....)))))))))))).))))))))))))..))))))).))((.(((((((...........))))))).)).....
..))))))).......)))))))..))))))))).))))))))((((((.(((.............))).))))))....)))))))))))))))..((((((..(((((((((((((...((((.(((((((.((((((.((((..((((.....(((((..((((..((((...((((.((((.((.(((.((((((.
......((.((........)))).....)))))).......((((...........)))).((((((.....)))))).....((((....))))))))).))))))))...)))).))))..)))))...))))..)))).))))))))))))).........((((((((((.......(((((((.....(((...(
(((((((((....))))..(((((((....)))))))...))))))....)))..)))))))......))))))))))..((((((((((((((.((((((.....((((((((.....)))).)).))..)))))).))))))))))))))(((((((((..((((....))))..)))))))))))))....))))))
)..))))))....)))))))))))))).....(((((.((((((.((((((((((((..((((((((((....))))))...((((..((......))..))))((((((((((.(((..((((....((((........(((((((..(((((..(((((((...........))))))).(((((..(((((((((((
((((.((.........)).))))......)))))))))))))))).....)))))..))).)))).....)))).....((((((((((((((((.((....)).))))))))).........((((((((((...((((.((((.(((((((((......(((((....(((.....((((.(((((((((((((((((
(..(((((.(((.(((..((((.((((((...((((((.....((((..((((.(((((((........))))))).)))).))))........)))))))))))).))))..))).))).((((.(((((.((((.(((.(((((((...(((((((.(((((((.((...(((((..((((...))))..)))))...
..)).))))..((((((((((.(((.............))).))).)))))))...(((.((((..((......))..)))).)))......))).)))))))))))))).))).)))).)).)))))))((((((((((((..(((..((((....(((((((((.....))))).....))))...))))........
.((((((((.............)))))))))))..)))))))...)))))..)))))))))))))))))..))))))))))..........((((......(((((((...))))))).....))))(((((((((((..(((((.((.....((((((.....)))))))))))))..))))).))))))...)))..)
)))).......))))))))))))).))))....))))))))))((((((((((((((.........)))))..))))))))))))))))((((.((((((((((....((((..................))))........)))))))))).)))))))))))))))))))))..)))).))))))))...)))).(((
((((((((....))))).))))))..........)))))).)))))......
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-721.33] kcal/mol.
2. The frequency of mfe structure in ensemble 2.9082e-36.
3. The ensemble diversity 855.91.
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
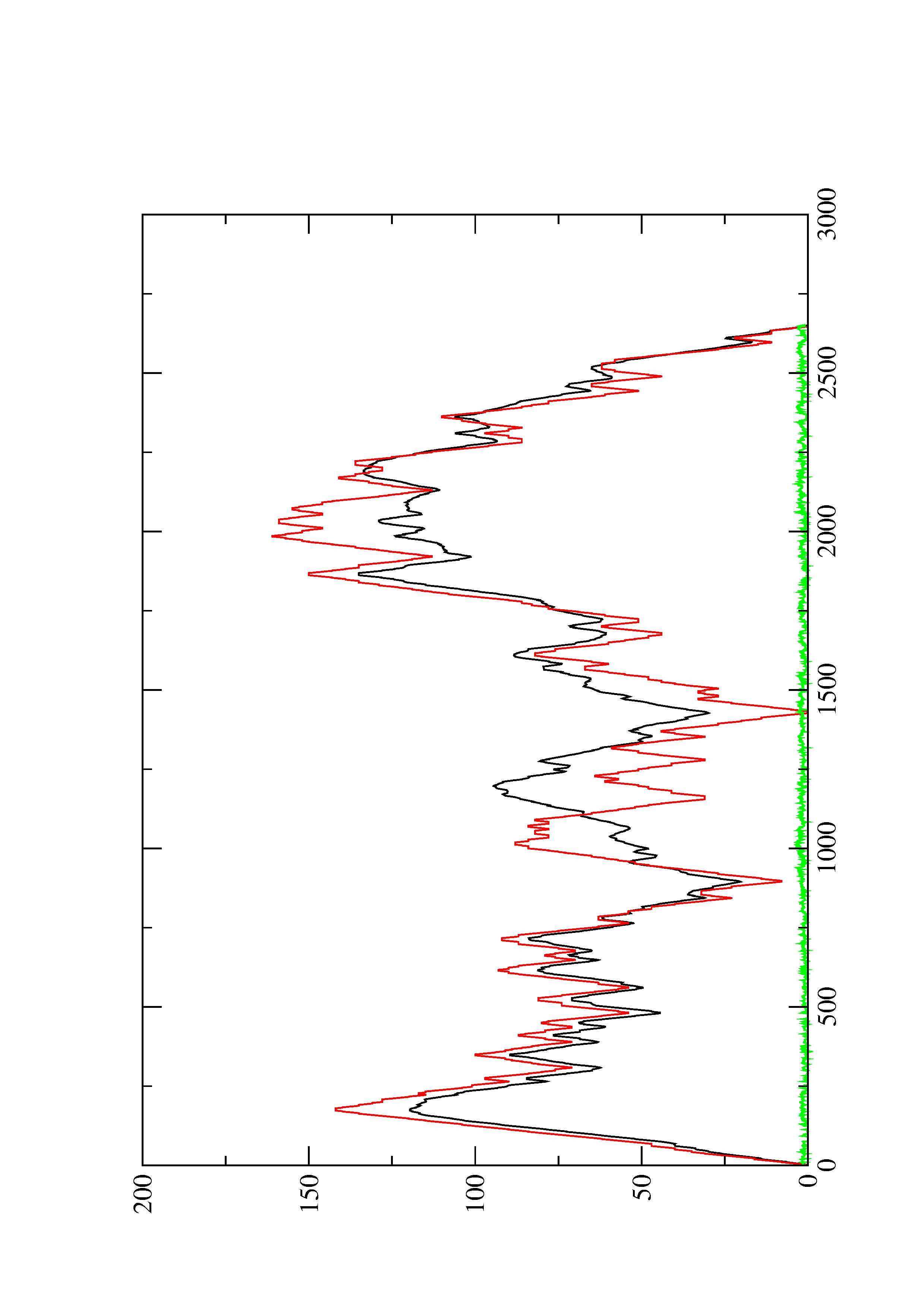
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.