lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01017183.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-594.10) is given below.
1. Sequence-
UUUACUUUCAAUUCCAAAGAGCACUGUACCAUUUUCGUUUCACCUACCCUAUUUGAGUAU
UUGUGAACCCUAGCAGCUCGAUUCCCCUUGGCUUUUACUCGAUUCUUCUUUAAGCUUCAA
GAAUCUUCUAGUGAUAUCAUUCUUCCGUCCUUGCUUUCCUACGUGGGUGAACCCCUUUCU
AGUGUUUUGUGGACAUUGUUCCUUCGUACUAUCCCGCGUUGGGAUUGCGUCCCUCUAUCC
CUGAGGUAUUUCAUUCUUGAUGUUCUUCUCAUACAGUCGUACUCUUAUGGAAUCUAAUGA
UUGGCUUAUGAUGGCUUUUAUUUCGUUUUCCUCAUUCAAAAUUGAAAUGUAAAGCCCCGA
AAGACCGAUAUCUUUUGGUUUCGAAUGAAGGGCUGUGAUUUUCCCUUUCUAGGUAAUUUU
UGUGUUUUCCCUUUAUUUCUGUAACUUGUUGAUCAAGUCGAGUUCCAUAUAUAAUCCCUU
UCCCUCUAGGGUUUUUGAGAGCUAAAAAAAAACGACAAGUUGACAGAAAAACAGUGGAUA
AUGGUCGAUAUAGAGUUUUUUGGAGUGAAUUUUAAGGUUUAUAAUUUUAGGAUUCCUCCG
AGUUUUAGAUUAUCUUUUCAUAUUAUAUUGCUAGUUCUUAAAUAAAAAAAUAGUUUUAUU
GUUAAUAUAAUGGUUUAGAGAAUUGUUAUAGGCUGUAAUCCUAGUUUUUAGCUACUUAGG
UUCAAGGUUUUAGUCUAAAAUUAUUAUAGCAGAGGUAAAAUUAGAGUGAGCUACAGUCCU
UGGAGUGUUUCACUCCUUUUAGGGAUUUAUAGGCGGUACUGAGCUUGAGUGUAUUUUAUU
AGUUCUAUUUCAGGUUUUCCAGGAUGCUAUUUGGCUCUACUUUGGUUCCUUCUUUCUAAA
CGGUAAUCUACUAUCCUCUUGUUGGUAUCUUGCUCUCUUAUGCUACCUACUUGCUGGUUU
AGGAUGCUUCGGUAAUUAUUAGUUGCUUAUUCGAUAUGCUAGCUAAUUAUGAUAUUACUU
GUCUAGUUGAUUACUUGUUAUUAAUUCUAGAAAUGAACCUGUUUCUCUAGUAAUUUUUGC
CUAUUUACUGCCCGAUUACUGCUAGAUGCUUAUUUGAGGGUAAGUCAUUUGUAUUAUGUC
CCUUUUGACAACUGUUUGAUACAGUGUUAGUUUCCUGUUUUAAAAAUCCAUGUCGUAUUG
UCACUGGUUAAUUGUUCACUACUUUAGUUGAUCAUAUUAAUUCAUCUAGAGUAGAUAUGU
GGUUUGUUACUUUAUCUUAUCAUCAGGAUAGACUAAACUAGUAGUCAUCAUGGAAUGAUU
UGAUUUUGCCUUGACCCUGGGUUACAGGGGGUUCUGAUGGUGGUUUUGAUUUGAUUUUGU
CAUUUGUCGUAUCUGAAUAGAGUUUAUCCUUGGUCAUUUUAGUUAUGUUCUAUGGUAGUU
UGAAAAAGGUCUGUAUGCAUGCUUAAAACAAAGUGGUUUGAGUUUGUUUACUAGAUUAGU
GGUUGCUGCUUCUUUUGAAGAUUAUGAACUAUAAAAGUUAUUUGUCAUGCAUUGAUUGAC
UUGACUAUGUUCUGUGUUGUUUCAUUCUAUCUAUGAGUCUCCCUUAUAGUUUUUGAUGUU
GGUCAUUUGAAUCUCUCUGUUGAUGCUAGUCACUGUGAUGAUCUUGGUCUAAAUCCAUGC
AUUCUUGAUUUUGUUAGUUAACAUGAUCAUUUCCAGCAAUCUUUGGUGUUAAUUGGUUUC
UAUGCUAUCUUACUUGUGGCUAUAUAGUCUAUUUUGUGUGAUCACCUAGUAUGUCAACUA
AAGUUGUAGAAUGAGUAUGAAUGAAUAGUCUUUGCCUUAUGUAUGUAUGCCUAUGUUUUC
UAUUUUUUAGAGAUUUGGGAUUGUGGAUGUCUAAGCCAGACACAUCCACCCCUUUAAAUG
AUAUUCCCAGUAUGGUUGUUUUUAUGCCCAGUUUCUUCCCUCUAAAAUGCAUAAUCAUCG
GUUAAUAUUAAUUAAUAGAGUUUAAGUUCUACCAGUUUGGUGGAAUAUAUAUUUUCUACU
CUAAGCAUGAUGUGGAUGUGAAAUCUCAAUGUUCGUUUUCUUUACUGUGGUUUUCUAGAA
CUAAUUCUAGCCUAGCAUGUUCUAGCUUAUGAAUGGCCUAUACUUGCCCUGUUUCGAUUA
AGUUUUCCUCUCUUAAAUGAUAUGUAUGGCCUGUGUGAAUAUUAUAUGUAUCAGAUAGAU
ACAAUUAGGGGUAUACUAAAGGUUAAAAUAUUAUCAUUCCUUUAUAGGUUGGAAAAAUGA
AUAGGUUGAUGUGUAUUUUUGUGUUUAUGUCCUUUGGGCCAGAUUGUAACCCGAGUGUUG
ACUCUCCCAUAAUUAUUUUUUUAAGUACGGGCCUAUCUCUAGGCUGCCCGAUUUCUUUGU
UUGGCCGAAGUUCUUUUAUUGUCAUAUGCCCUUGGGAUUGGCCCAUGACUGAGUAGUUUC
ACUUUAUAAUUAUUUGUCCAUAAUUAUGUGCUAAUUUGUCUGUAAUGUUUAUUUAGUACC
ACUUUCUAAUCCCUUUUCUUGUUUCUAUGUUCGAACCUUUGUUAAGAUCCUCUUACAUGA
UCUUGUGGGCUCUGGACCAUC
2. MFE structure-
............((((..((((.((.............(((((.(((.........)))...))))).....((((((((((((.....(((((.................)))))....)))))....((((....))))...........((..(((((((((((............((((((((...))))))))..
............)))))).)))))..))...((((.......))))...(((((((..((......))((((((((((((................)))))))...))))).(((((((((((((((((........)))).))))))).))))))((((((((......))))))))....))))))))))))))....
...(((....)))..........(((.(((((...((((((((((((((((.........(((((((........((((........))))....)).))))).........))))))))).)))))))...)).))).)))(((((((((((((...(((((((.(((((((((((((...))))))))))))))))))
))..........(((((...((((((((((..........(((((((......)))))))..))))))))))....)))))...((((.((((((......)))))).)))).(((((((.(((((((...((((..((((((((...((((((((((((((((((.((((((.(((((((((((((...))))))....
)))))))....((((...(((((((((((...................))))))))...)))..))))...))))))......(((((....((((((((((((((((.((((.......((.((((....((........)))))).))..((((((...(((((....(((((((...)))))))))))).....)))
))).......((((.....)))))))).)))))).))))........)))))).))))).....)))))))....))))))).....))))(((((.(((((((((((((((((....)))))).)))........((((......))))(((((.....))))).(((((((((((......(((((((((((......
....(((........))).((((((((.(((..........))).)))))))))))))).)))))..................)))))..)))))).)))))))).)))..))))))))))))))..))))))).)))))))...((((((((..((.((((((..((((........))))..........((((((((
(..((((....((.((..(((((....(((...........)))...((((((((.(((.((((((..((...))..)))))).))).))).)))))...((((((((.(((....)))((((((((((((..(((((((......((((((.((((.....(((((...((.(((......))).....(((((((...
..)))))))....((((....((((...((.....))...))))....)))).......))...)))))..)))).))))))...)))))))..)))))..))))))).....)))))))).(((((((((((((((...(((((.....((((.((((.(((((((......))))))).))))..))))(((((..((
(((..(((((..((.(((......))).)))))))..............((((...((((((.....))))))..))))...((((((((((.....)))))..)))))..)))))..)))))......((((((......)))))).....................)))))...)))))))))))))))....(((((
(..(((((((((((...))))))))).......))..)))))).((((((((((((((((((((((((...................((((((....))))))....(((((((.(((.((((((((((((((.(((........)))..........((((.........))))((((((((((.(((((((((((...
.(((..((((((.....))))))..)))...)))))...))))))..))).)))))))..))))))))))))))..)))..)))))))....................(((...((((((((.......((((((.((((((...........(((((....)))))..((((((....))))))(((.(((......))
).)))...............))))...)).)))))).)))))))).)))))))..))))))).............)))))).))))))))))))..)).))))))..))))))))).)))))).)).)))))))).......))))))).))).))).....(((((((.........))))))).))))))))))....
.
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-646.04] kcal/mol.
2. The frequency of mfe structure in ensemble 2.5009e-37.
3. The ensemble diversity 839.80.
4. You may look at the dot plot containing the base pair probabilities [below]

III. Graphical output
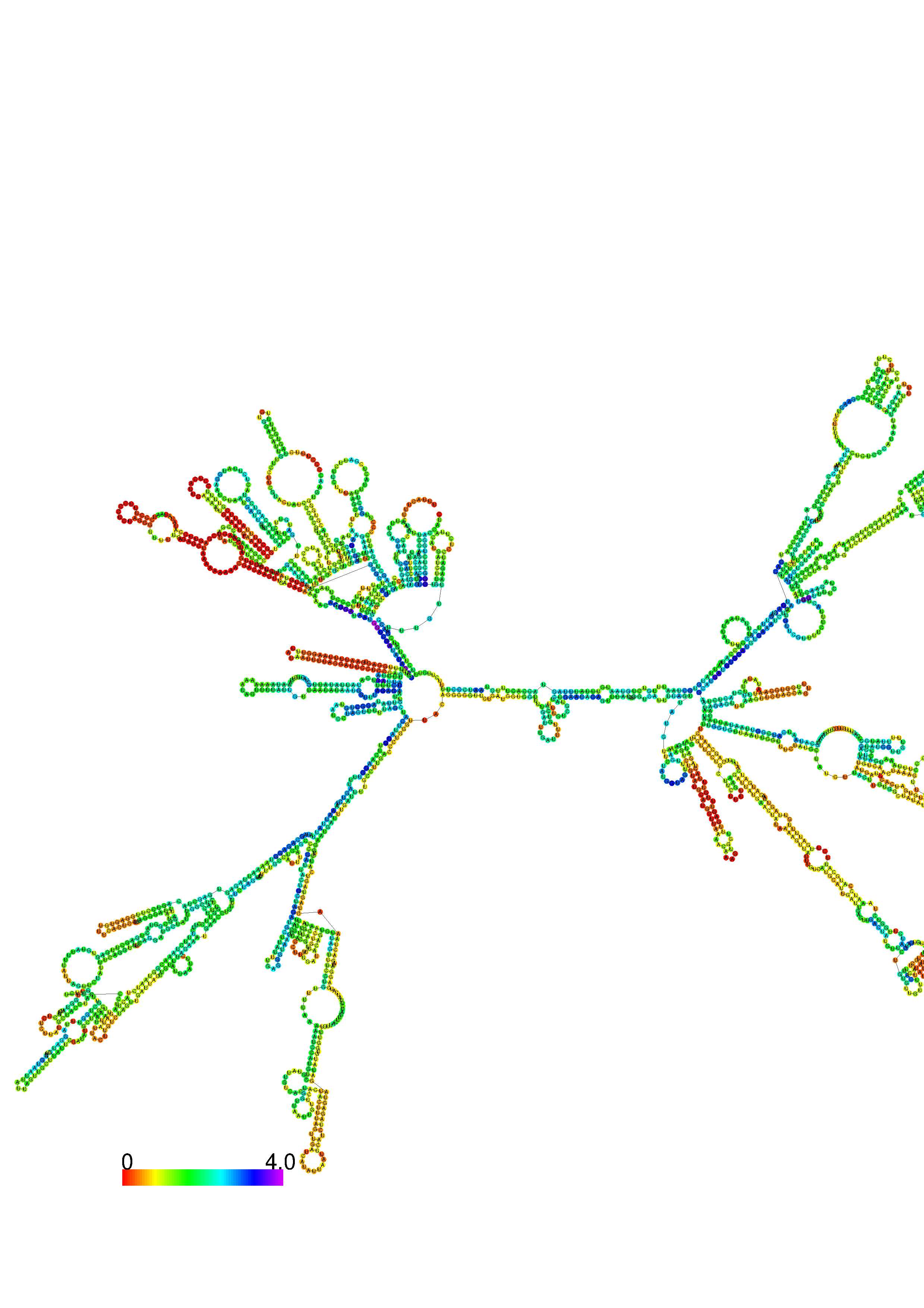
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
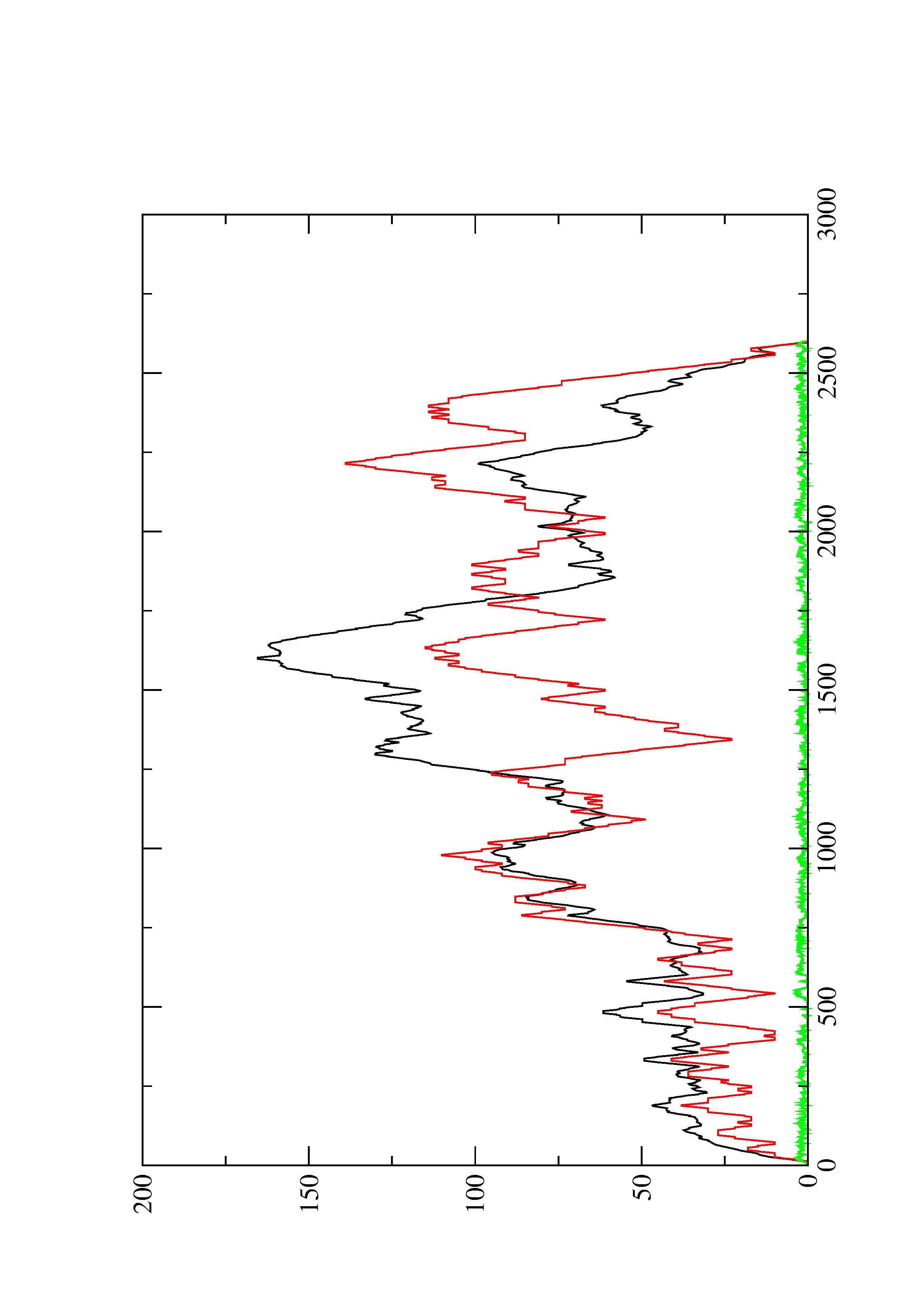
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.