lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01017590.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-718.40) is given below.
1. Sequence-
AUACAAAUACCAUUGAUUGAAAAGAAAAAAUAGAAAGAAGGAAGGAUACAAAAGAAUGGA
GGCUAAUAGUAGAAUGGAAAGCUUUAAACCCUAAAACAAUACAAAGAGCAUUUAUAUAUG
CUAGACUAAACUAAAUAAGAAAAAGACUAUACUAUCCUCACAAAGCAUAAUAACUAUUUU
AACAUUCCCCUCGUUAUUUCACCCCUAAAAUUAAUCUGAUGCAUGUCAAGUUGACUCUGG
AAGUUUUCUAGCAAUUCAUGUUUGCUGAAAUCUAUUCUAUAACUCACCAUGUUUGAAAUA
UGCAUGAUUCCUUAUGUUUCUUCACCUAUUUAUAAAUUGGUUGGGUUCCUCAAAUAAUGA
ACAUGAACUUCUGCCCAGCAAAUACUCUCUUUUCCCAGUCUUGACCUUAUCAUUAGAGAA
AUGACCAUUAAAAUGCUUUCCUGAUGUAUCUGUGGACAUAUGUCAUGAAUGUUUAUGGUC
CACGUCUCUUCCCUUAGGGAUUCUCUUUCUGUUUAUUAAGAGUAACCAUGCUAUUUCUAA
UUUCAGUGAGGUGUUUUAAAAUCAGAAUUACUCAAUCUUGAUCAACAUGACAAGUUCUAG
AUUCCCUUGUGAAAUUUAGUGUUUCAUCAUUUUGCAAAAUGGUUGGAUGUUGGAUUACCU
UGAAUUUACCUCCCGGCUAGUUUAUUGAAAGCAGUAAAAAGAAAUAUGAUUUUGUGUACA
GAUCCCUUUGGUUUCAAAUCUAGUCAUUUAUAUGAUUGUCUGGACCAAAUAUCACUGGUU
GUGACUUGGAUGAGUCCACAUUCUCAUGAAGAGUUGUAAGGAACAUCUUGCAAUAUGCUU
AGAUUUGUUUGGAGGUUAUGUGGUUUGUGUGUUGUCGGUUGCUUACUGUUCUUAAGGGCC
GUAGCAUGUGUACUUAAGUGAAAUAGGUUUUUUGCUUCAUUUGAUUUUCUUGAUUUGUUU
GUUUAAAACAUGUUUCUUGGUUCUGUUGGUUUCCAAAAAUGAUCUUUGUAUUAUUUAAGG
UUGAUUUUAUGUGCAUUGAGGUGUUGUUCACAGUUGUUUAUUGUUUCUUUUGAUGUAUAA
ACCCCUUCCUAGGCUGAAUUGUUUGGUUCUAAACCAAAGGUGAAGGAGCAUGUCUAAGGU
CUUGCUAGUUUAAUAACUUUGGUUUGCCAAAGUUAGUGCCUUUAAAGUAGGUUUGUUGAU
CUUUGUGGUCUUCAUGAAUUCAAGGGGCCUAAAUUGAGUUGCCUGUUGAAACAUAGUUUA
GCUUAAUGUUUUGUUGUGGCUGGUUUGUGUUGUACCUUAAGUGAAGCUGAUGUUCUUGGU
AUAAGGUUGAUGAAUCAUAGGAGUAAUUGUUUAGUCCCUGUGAGUUAUCUUGGCUUGUGG
AACUUUGGUAAAGAUUCAUAAAUGUAGCUUCAAUAACCUAUUCUAGGUGAAUGGUUGAUG
UUUGCACACUAAUAGGUCAUUAACAUGUGGAAUGUGUUCUUUGUCGUCUAUUCAUCACUU
AGCAAGUUCAUGUACGUAUUCCACCUACAAUUACCAAUUUCAAAAAAAACAUAUUCAGCA
CCUAGACUUAGUCCCAUCUGUUUUCUUAUUUCAUUAAAUAAAUAGGACUAAAAAUUGGAA
GUGUUACUCUCUAAUGCCCUGAAUAACCUGAAAACAAGUAUGAAUUAAGCUGGAAUUUAG
CUUCAGCUGUUCUAAGGUUUAAUUAGGUAUCUCAUGUUAAAACUGGAAAGGUACUUGCUU
GAGACUUGGUUAUUUUAACUCCUUUUUACCGUAUAUGAAGUAAAGAUUUUUACAACCAGC
AGAAUGUUGAUUGUUGUUGUUUUAUUUGUUAUUAAGCAUCAUAAUGCAUUCAUAAUUAAA
ACCUGAACCUUGCAUUAACUUAGUGUAGCAACCUGUUAGGUGUGGAUUAUUUUAGACCAU
UAAUUUUCAUACUAAUCUUGGAAGUUUUUAUGAAGCACUAGGUUCUUUUGCUUGCUGCUG
AAUAUGUAAGCUUGCAUACUUUAGGGAAAGUCUCUUCCCUUUAUGCUCACUUUGGUUGGU
UUGUUAGUCAGUGAUGUGUUGAAGUAUUGCUCAUGGUUGCUAUUUUAGUAUGUAAUUAUG
UAUGGGUUAAAAUGUGGAGGAUUAGGUGUUGAGGGUGUAGGGGAACGUUUAGGGCCAGUC
UGACCCAUAUUGUUUCAUAAUGUCAAAGCAACUGCUAGAUUGGUUGAAUACCUAUAGCAU
GUUUAGAUCAUAGCUAGAAUUGGUUGCAUGAUAACCAGUACGCUCGAACUUUGUCUAGUU
UGUCCCUCUCGAGUAGUGUUAACAGCCUCAAUUUUUCAGUCUUUACUCUAGCAUGUUGAA
AUUAAAUGCCCUUCUCGUGACUAGCUCUCAGGUUUAUACUUUACUGUCCAUGAAUAAGUG
AAAACUCUUAAACUCUUCUUUGCUAUGCCUUGAGAUGUGUCAUGGAGUUCCUGGUUACAA
CCUCUUUACGUAAAAUAUAUGAUAACUUUGAAAUAAUUUUUUCCCAUGUUUGCUUCUGUU
UCGUCUUGAAGUUAUUGGUUCUUUGAACGAUUUAAAUUCAAUUUAGGAUUUUAUUGCAUU
UGUUGGAUACUAAUUCUGUUCUCUUCUUUGCAUGUAAAACCUCCCAUCCGAGUUCCACCC
GGGCUCUCUCUAUUUUUUAUCCUCGGGAUAAAGCUCAAUCGAUUCAAACCUCAUGCCAAG
UCCUUUGGACUGAAGAUGAAGAAAUAAAAGUAGGCUACAGCCCAUUGACGAUGAGAAGCU
GCAGUCUUCGUAGGCUACUUUUACUCAAUUCUUUUUUGAUUUGGGAAAAAGCCCAUAAUU
UAUGUACGAGUUUGUAAUGGACCAUAGGCCAGAUUUAGGGCAGGUGGGCAACAAGCCCAU
AUUUGAUCUAUUUAAUUGAUGCAAUGAGCCAUUAAGGGCCAAUCUGUUAGAUUUAGGAUA
GAAGGAAGAUAAAAUGGGUUGAAACCCAUGAUAACAAGGUACGGUUCUCUCUUAAGCAUA
UAGAAUGGAGAACAGCUCCCAAUUAAUUUAUGUUCGGG
2. MFE structure-
..........(((((((((((.(((....((((((((((((((((((....((((((.((((((.(((.(((((((...(((((((.........(((((((((..(((((......)))))............................((((.((.((((((((((...(((((((((((((.....((((.......
.(((((..((((........(((((...(((((....(((((..((((.(((((.......)))))))))....))))))))))...)))))...((((..(((.((((...(((((((((((((((.........(((((((((((..(((((....((((((((((..(((...(((((.(((.(((......((...
(((((((((.....(((((...((((((......((.(((.((((((((........)))))))).))).))..))))))....)))))......(((((.((((((((((((.((((((((((....))))..............((((..(((((......)))))..))))..)))))).)))).)).)))....))
).))))).(((((((.....))))))).((..((((((..(((((....(((((..(((.((((((..((....))..)))))).....(((((....((((((((((.(((((...((((((((((((((...(((((((((((((....))))).(((..(((((........)))))..)))....(((((......
..))))).....((((((((......)))))))).....)))))))).))))))).............((((((.(((((.((((.......)))))))))))))))......(((((((((.(((.....))).))))))))).......))))))).....)))))))))))))))....)))))))).)))))...)
))))))))))..)).))))))))).....))......)))))))))))..)))..))))).)))))..)))))......)))))....((((((......)))))).....))))))))))))))))))))).....)))).)))..))))..(((((((((....)))))))))..((((.......)))).((..(((
(.....))))..))..))))..))))).......)))).....)))))))..)))))).......)))))))).)).)).)))))))))))))........)))))))..))))).)).)))......(((((.((((((((.(((....))).)).)))))).)))))((((((((((((((..(((...(((((..((
(((((.(((((..........((((((((..(((...((((((.......(((.(((.(((.....(((((((((((....((....(((.........)))......))...)))))))))))....))).))).))).......))))))..))).)))))))).(((((((.((.(((((............)))))
.)).)))))))......))))).....(((((.(((((((.((((.(((((................(((((((.......)))))))...(((((..((((.((((((((((..((....))..))..)))))))).))))..))))).........((((.(((((((((((((((.(((((....)))))...((((
(...)))))...(((...((((((...(((((.((((((....)))).)).)))))))))))...))).((((((..(((....(((((((((.....(((.((((....)))).)))....(((..((.(((((((..((((((((.(((..((((.(((((......))))).))))..)))...))))))(((((..
..((((((......)))))).)))))...))..))))))).))..))).(((((((........)))))))...)))))))))...)))))))))))))))))))..)))))...))))..))))))))).))))))).)))))))))))).....)))))...)))....))))))))).)))))((((.(((((.((.
.((...........(((((((.......))).))))..((((((((.....)))))))).....((((((......))))))..))..)).)))))))))...)))))).))))))..))))))...((((((((..(((...........))).)))).))))......(((((.(((((..............)))))
.))))).......)))))))).))))((((((((.((.(((((((...(((..(((((((.(((..((((((((...((((........((((......))))....((..((((((...((((((((((.(((...((((...))))....))).)))))...(((.((((((((((......(((((.......))))
).......)))).))))))))).......((((((.....))))))(((.(((((((..(((..(((((...((....................))...)))))..)))..))))))).)))......(((.(((....))).))))))))..)))))).))(((((....))))).(((((.(((((.((......)).
))))).)))))..)))).)))))))).))).)))))))))))))).)))))..))))))))..((((((.....))))))......))).)))))))))))...((.(((......))))).((((((........))))))..(((.(((((((((((....))))))........((....(((((((((........
.)))...))))))....)).......))))).)))...
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-772.83] kcal/mol.
2. The frequency of mfe structure in ensemble 4.45276e-39.
3. The ensemble diversity 1029.69.
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
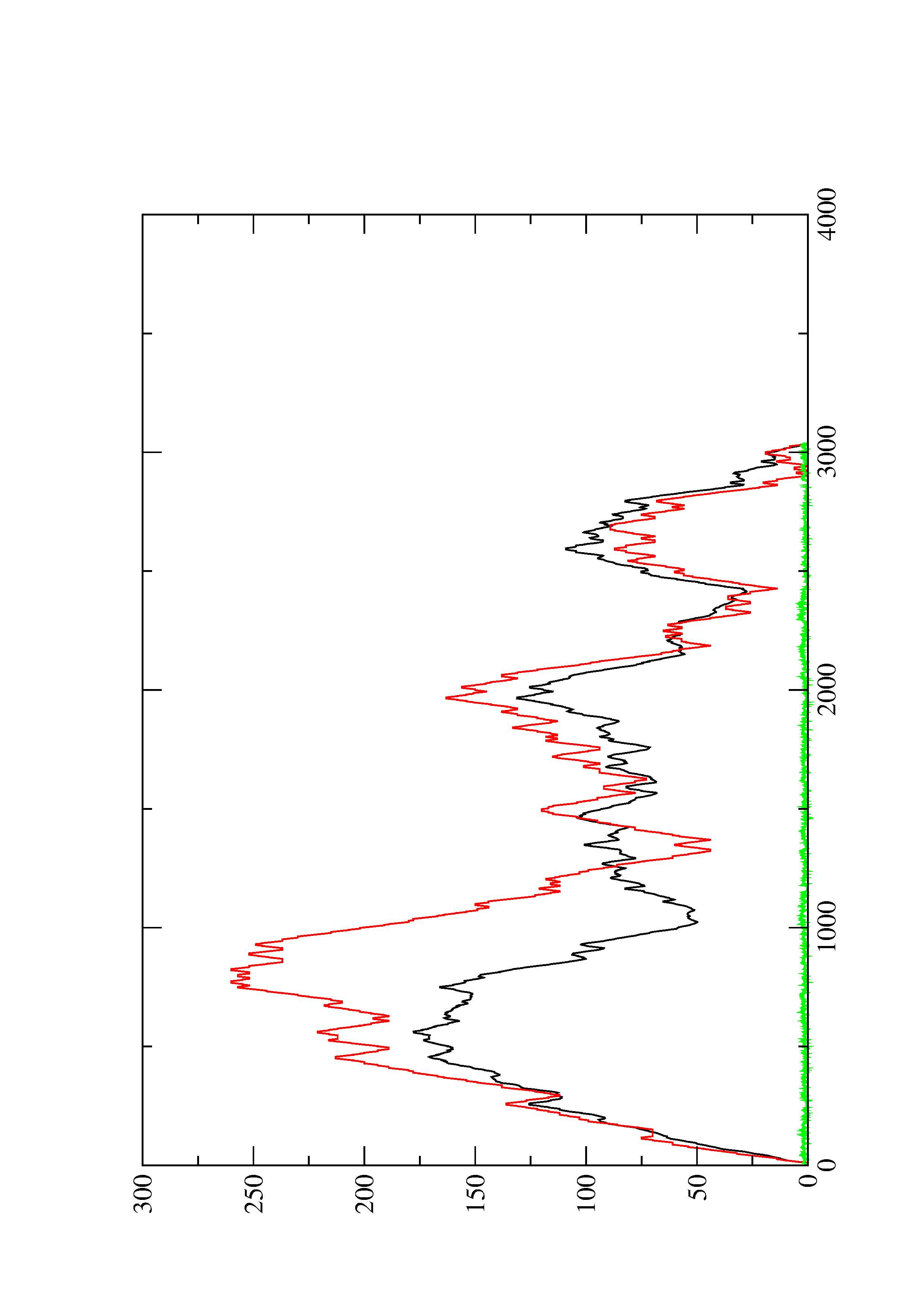
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.