lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01019191.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-921.10) is given below.
1. Sequence-
AAUUGGAUUAUGAAUAAAUUUAUGAUAUUUAUUAGUAUCAUGCGUAUUUUUUUACUGAUG
UGAAGUCUCACGCACAUGAUGUUAUUGAUCGCCCUGUGGAUGACAUUGUUAUCUUAAGAU
UUUGGUUCAAGUUGUGUCUUUUGAAGUUCAUUUCAAAAUGUUGGAUUAGGCUGUAUCACA
GAGGCAAAGACAUCCAUUUUUCAUUGGAUGAAAGCAUUAUAUGGUUCCCUAUUUAACCAA
GUCUUGCCAAGCAAGAUGAAACAAGAUCUGAUCUUGAAAGUACUAUCGAUUAGUUACUUG
CAAUUUUUUUUCCGUUCAGAAAAAUGUGGAUUUGUUAUUGUUUGCGGACACUAUCUAAAU
UGCAAGUUAGCUUGUUCCUUGAUGCAUUUGGUUACUCUUGUUUCCCUUUAGCAUGAUUCC
CCGUGUAGGUAGUACCUUGGUGGUUGCUUCCAUUUUUUGUUUUUGAUUUUUGCAUUACGG
UUUAUUCCAUCUCAUUUUUUGAGAACAUGGAUACCAAUGCUCCUUUUGGCUUGCAUGCCA
UACAACUAGAUCUACAUAUUUUUUUUUGUAUUUCAUGUUAAACUUUGUUCAAUUUCUACU
GUAGAAUUGAAACCUACAAUGUCACUGCAGUAUCAAGGAAAGCAGGUUGAGGAGCUUAUC
UACAACCUGGCAUACAUAGAAUUUCAAAUUUAUUUUCCGUACUUCAGAUACUCGUAUUCU
CUGUCCAAGCCAGUAAAGGUACUUUAUUACCUCAUCUGCGAGAGAUGUACAAAUGUGCAG
UUUGAUAUCUUGUGCAUUCUUUUCUAGAUGGGGGAAUUCUUAUUUGAGCCUAUAAGGGGU
GAGGCUUCAUUGAUUUUUGAUUCUUUCUAAUCUUUAGUCUUUUGGAAAGAUGACUAAUGG
UUUUGCUGCUGCGAUCCCUUGUUUACGUUCUUUUUGAGGCAAACUAGGUGUAUCUUGGCU
UUUUUGCAAGUGGGAAUUUUUUAAGUUGAUCAAUGAAAGAAGGAGCUAGUGUUAAUUUUC
AGCCUGUUGUUGACUGGUUUUUAUUAUGUUCUGAUAACAACAUUGAAACAGAGAGCAGGA
CUGUAAUAACCCUACUGGGUUUUACUGCAAAGCUGUCAUUCCUUGAAUUGUUACUCUGUA
AGUCGUGUAGAAAUUUUUUGGGCCUUGAAUCUGUGCAACUGGGAGGUCUUGACAUCCAUA
UUUCUUUUGUUCAAUUUUAAUGGUUGAAUUGAUUCGUGAAAUUUUAAUUGCCAACUGUUC
UGUCUUGUAUGGAUAUUUAUUUUGAUAGAGGGGGGUCAUUGGUGGUGAAGACAAAUUAAU
UUAGUAGUGUUAUUUUUAACCAUACCCCAACCAACUCUCUCUGCUUGCUUCAGACAAGAA
AGAGCAGGUGAGCCUGGUUGCAAAUGCCUGCAAGAAGGGGCUGGAAUAGAAGAGUACAGG
CUUGUAAACACGCAUGUUCACAUUUUCCCCUAAGAUCUUUCUUAUGGGGUGUUAGGCUGG
UACAUUAUGUGCAACCAGUCCAGACUAUGAGAGAUUGAUGUAAGGGUUUAUAUCUUGCUU
AAAGUUGUUGGUGUUUUACUAGAAUGUUGAAGCUAUGAAAUGGGAGAUGGAUGUUCCAUC
GUUGUGCUUGGAAGACCAUGCAAAUGUUGCAUAAGGCAAAAGUUUUGUUUUUUAAUAAAU
AGCAGCUAAAAUUGGGUACAAUUUCAUGCUUUUGUUUUUUGAUAGUCAUCUCUCCUUGAU
UUUAUGGAGGUUCUUCCUCGCAUGUUUUGGUCACCACCUGGGUGUCUGCAGAACUUUGCA
AUCAGACAGAAGGGAAAGCAGUACAGACUAGUCAUCAGCAAAGUGAAUGUGGCUGAUGAG
AAGUCCCUUGUUAUUUAUUAUAACCAACUCGAUUCUCUUUAUUGAAUAUUUUUGUAAAGA
GCCUUUCAUAUUUUCCUCUCUAACUAAUUUCGUUCAGUCUUGUGUUGCACGUUAGGGUGG
UUGGUGUCGGUCAUGAAUGUAAGGCCAAGUAACAAAAUGUUGUCCUGUGCAGAAUGCGUA
AAUGUUAGGCCUAAUAACAGAGUGUUGUCCUGUGCAGAACACAUUUUGCAGUGUUGUUCG
GCAUAACUCCUUAUGCCUUAUAUUAUUGGGGACUGUUAUAUACCAGCCUAACUGGAGCAG
UGUGUGCUAAUUGGUUGUGAGAAGUUUUCUUGUGGCUGUUGCUUCCUAAUGAACUUGGUU
UAAAUCGCUGUUAGGAUUGUUGAGAAAAUGCCUCCACAAUACUUUACAAUAAUGAUUGAG
GAGUAAAUUGUGUUUUAUGUUCUUUACAUCUAAAACUGUGAUGCUGUACUGUCGGAUAAG
GUAUUCUGAUGUUCGGGGCGGCAACUAUGGCAUUCUUUUCAGAAUUAUGUCUGUUUCUUA
UAUGGUGUUGCCUUCUAUGUUGUAUGCACUAACGUUUUAAUUAUUUGCUUACUAAAACAC
UCCCGUUCCAACUUGUUUUGCUUGCUUUACCUUUUGGUUUUUUAAAAGAGAAUAAACUUA
UUAAAUCUUUUUUAGUAGUUCUUUGAUUUUAGUUUUUUAAAUUUUACAUGCACGUUUAAG
AUAAAAAAGUCCCGACAAUUUCAUACCUUCUACAUUUCUUUGGUUUAAGAGAGGAAAAAA
AAAAGUAUGUUGAAAUUCUAUUCAAGUCAAAGUUAAAUCAGCAGUAAAAUGGAGGGAGCA
GUUGAUGUGCUCACUGGGAGCUGAAACUCACUAGUGUGUUCCUAAUACAGACGACGUAAA
UCGGCUCUUUGAAUGCUUCUGAAAAAGUUUUCAGCAGAGAAGGAAAAGAUGAUUUUCAAC
GCCUUUUUGAGGUCAUAAGUUGGAUUAUUCAUGUUGGCAUCGAUGAAAGGUUUGAAGUUC
UGCACAUCUGAUGCUUUCUUUUGAUAUUGGUCAGUUAUGGCAUAAGAAAAGAGGGUAAGA
GCAAUUUUAAAAUGCUUGGCCAACAUUUGCCGUCGUAUUAGUUGUAUUCGUUUGUUCUUG
UAUCAGUUGAUGCAUGUGUGCAUAUAGAUCAUAUGGACGAAUAUAAUCUGCGUGUAUAAU
AAAAAUGUAAGGGAUUGUCUUCAUUCGUUUUAAGUUCACUUGUAUUUAUCGCCAUUAUUG
UCAUAAUUUCUUCCUUCACAUACGUCAUACGAGGUACAAGGCAGGAUUCUCUUGGUUGGG
CACCAAACCAUACAAAUGACGACAAGGCUUUAUGGAGUAAAAAGUUAUUUCAAAUCUUCC
UAUUCCCGGCGUUCAGGCUCAUAAGAAGAUUUUGUCAUCUCACUUGCUUAGAUACCUACU
UUAUGUAAUCUCGGUCAUUUGUAUAGUGUUUCAUUUUUUUGUUCUUUGGGCCUUCCCCUC
UCCAAUAAUAUGCUUUUCAUCUUCAAUCACCUUAAUUGAUUGCAGAGUCUAAGCCCGAUC
AAACAAUAUCGCGUGACUGAAAGAAGGAUUUGUCAUUACACCUUGAGAACUCAAUGCAUU
GGUGGUCUUUUGCACAAACCAUUUGUUAAGCGUAGGUCGGUCUUAGAAAUGGCAAAAAAU
GGAGAGAGGUUUUUUUGAAUUGGUGGAACUCGAAAG
2. MFE structure-
..((((((((.((((......((((((((....))))))))((((....((((((....))))))....))))..(((((((((((.((......)).))))))))))).........)))))))))))).......(((((((..((((((((((((...((((.(((((((((...((((((((((((((((((....
....)))))............(((((........))))).(((((((...)))))))(((((((((..(((((..(((.(((.((..((..(((((((((((((((....(((((.(((..((((....))))....)))...)))))........)))))))))).)))))...))..)).))).)))))))).)))))
))))......(((((......))))).((.((((((.....)).)))).)).......))))))).))))))...))))))))).))))(((((.....))))).....(((((....((((((((((((......)))((((((..(((..........)))..))))))(((((....(((((.....((((((((((
((((((........(((...((..(((.(((((...(((((.(((((((.(((.....))).)))))))......(((((........))))))))))))))).)))..))..)))..........((((((....((((((.......(((((((((.(((((((((((((((((........)))).))))))).)))
))).)))))))))....(((((...((((..(((((((((((((..(((........)))..))))..((((.((((((.(((....))).)))))).)))).(((....))))))))))))....))))....)))))..........)))))))))))))))))).))))))))))....))))).....))))))))
))))))..)))))...((((((..((((((((((((((....)))).))).....)))))))))))))((((((((((((..((((.(((((....)))))))))((((((((((..(((...((((..((..((..((((((.(((..((((((((((((((((.((((..(((((((((((((((...((((.(((((
((((((((((((........((((..((((((...........)))))))))).(((((((.((((((.((((............(((((((((....((((.((((.....(((.((((........)))).)))......))))))))....))))))))).....)))).)))))).)))))))...(((((..(((
((......)))))....))))).))))))))))).((((((......((((((.(((((.((....(((((((((....))))).)))).((((((((((((.....((((....((((((.((((.((((.....(((((((....)))))))...)))).))))...(((.((((((.((((((((.((.....(((.
((((.((((((...))))))....(((.(((....))).)))........(((((((....)))))))....(((((((((((((...((((((....))))))..(((((((.((((((((((((((........))))).....((((.....))))(((.((((((..((((.....))))....))))))..))).
))))...))))).))))))).....((((..((((((((............))))))))..))))..))))))))))))).......)))).)))......)).)))))))).)))))).)))....(((((..((.(((((((((((..((........)).))))...))))))).))..))))).((((........
..))))......((((((((.((.(((((((((((((.....(((((.......)))))..)))))...)))))))).))))))))))(((((.(((..(((((((....)))))))..))).))))).)))))).)))))))))))))))))))))))))))))......(((((.(((((((......(((....(((
(((.(((..(((((..(((....(((((.(((.((((.((.(((((.(((....(((((((((((.((((....)))).))))))..)))))...))).))))).))))))..))).))))).....)))((((((........)))))))))))))).))))))....)))..))))))).))))).)))))).....)
))))))))))))))))).)))))))...((((((((((((....((((((.....(((.(((((..((((.....(((((((.(((....(((((((....)))))))......((((.((((((.....))))))))))((((((((((((..(((((......((((((..(((...(((......)))..)))....
.......((((...((((((......))))))))))........)))))))))))..)))...))))))))).......)))))))))))))))))))..)))(((((((.(((((((((......))).))))))..(((((........(((......))).(((((((((.((((......)))).))))).)))).
))))).....((.((((((.((((((((((.((((.....((((...))))...)))).)))..))))))))))))).)).)))))))...(((((((((((..(((..((((....)))))))..))))))))))).))))))..)))))))).)))).........((((.((((...(((((((((((((((...((
.(((.....(((((....)))))...))))).))))))))))))))).))))..........((((((((((((((................))))).)))))))))...(((.((.(((((((....((((((((....(((.((....)).))).)))).)))).....(((..(((........)))..))))))))
)).))))).....((((((..(((...........)))..)))))))))))))))))))))...)))))))))..)))...))))))..))..))....))))...)))..))))..)))))).............))))))))))))(((((....(((((((......((((.....(((.((((((.......))))
)).))).((((((.((((((.........(((.((((....(((.((.(((((((...((((((((....))))......))))......)).))))))).))))))).)))..)))))).))))))...)))).....)))))))))))).)))))))...)))))..)))))))
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-986.19] kcal/mol.
2. The frequency of mfe structure in ensemble 1.35705e-46.
3. The ensemble diversity 1106.67.
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
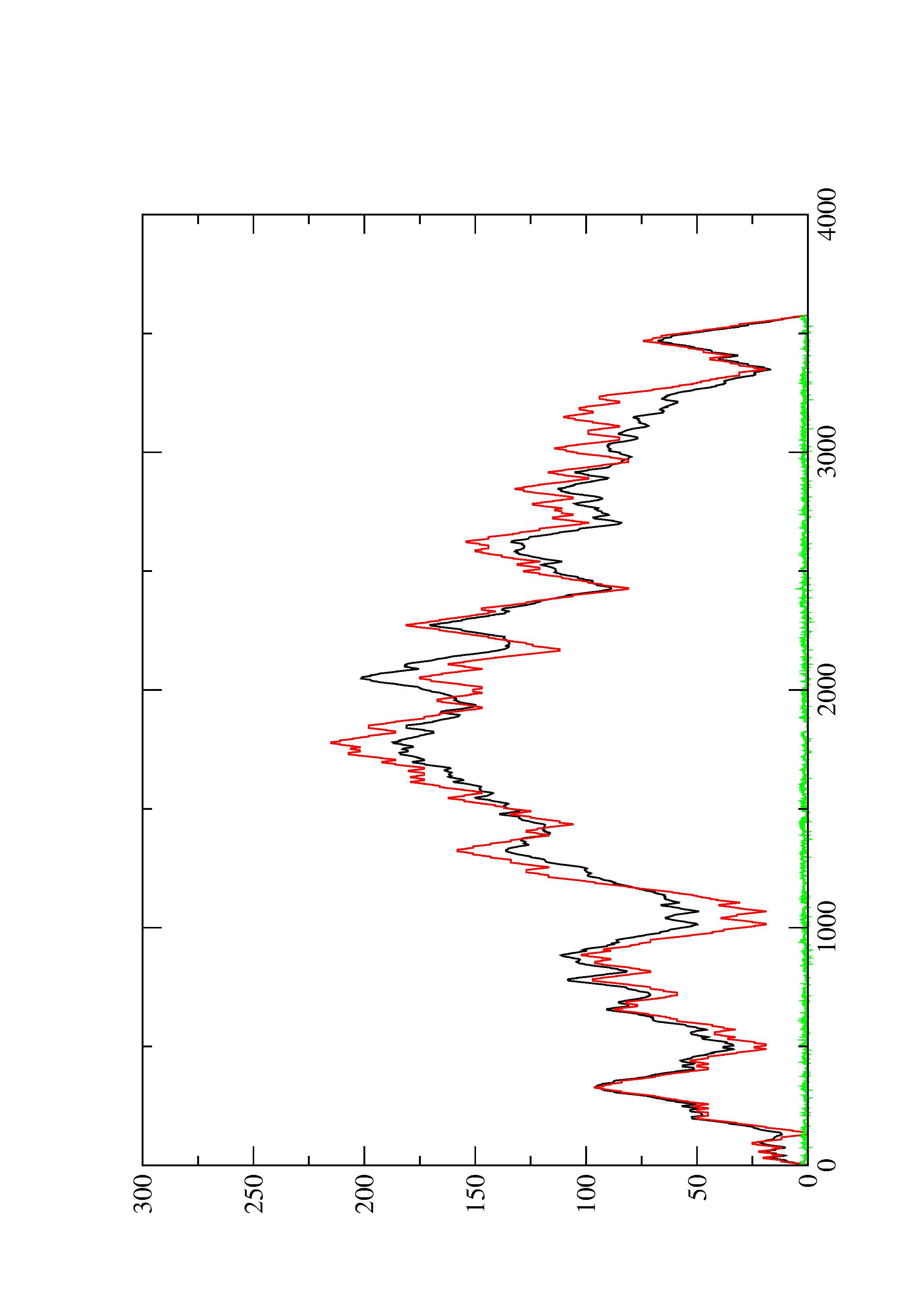
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.