lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01020532.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-705.87) is given below.
1. Sequence-
CCCGUCACUCCUUUUCCCUAAAACACCCGUAACACUCAGACGCUCAAAAUUCCUAAAUCU
CUUCCCCUUUAACCCGACUCUCUCAAGUGGAUGCUCUUGUUUCUCGAUUAUUCUUGUUGU
UCCUGAAGCUUUGAGAUGUUUUCUCUUUUUGUCCUAUGUGCCUCCGUGUCACAACACUAG
CUUGUGAUUUUUUGGCCAUUGUACCUUCAUACUGUGCCGUGUCAAGGCUUCGAUCCUUUA
UGGUUGAGGUAUGAUUUUCUAUGCUCUCUUUGUACAAUUUCAUACACUAUUAGGAACCAA
UGAUUAGCUGAUGGCACUCCCCUUACCCCCUUUGCCCGAUUUGAUUUCAAAAUGCAAAAU
CUUUGAAGGGUGGAGGUCUUUUCGAUUUUGAAUUUAGGUUGGGAUGAUCUUGGUUUGAUU
GUUUCCGGGGUUUUUUACCCUAGAACCGACUUGGAGGGAAUCUUCCAUUCCUUAUUUAUA
CCCUGUUGCCCCCUUUUAUUUUGCAAGGAUAACAGAGAGAGAAGUAAAGAUAUACAAAGC
AUACAUAGAGAAUAUCGAUUACACAGUUAUCUACUAAGAAUCCAGUUAGGGUUUUGGGAA
UCCAAAGAAUAUUUAGUAAUAUAUUAGUAUUUGUGAGGAUUAACAUUGUUUAGUGCACUG
UUUAGGCUUGAAUUUGGUGGUUCUUGCUGUGACAGUAUCUUGGUUCAUUCUUAGUUGUAU
UUUAAGCUAAAAUUGGUUUAAGAGAGGUAAAAUCUGAGUGUAUUUAUUUGAAUUAUUUUC
CAUUUAGGAGAACCAUCUAGGCCAUGUUACAUGUUUAUAAAUAGUUGAAUAAGCUGAUUU
GUACAUUUGGUGAUGGUUAUUUGAAUGGUCUUACUGAAGGAUGCCUGUUGAUACUGUUUU
AUGUGAUUAUUAUGUCUUAGCCUGCUGUUUUGAUCAUGUUUGCUAAAAUGAGCUUAUUCU
UGCUUUUAUGUUGUCUUUAAUAUCUUAAAAAGGAUUUGAUUUCUGUUUGCUUAUUAGGUG
UUCUAUGUUCUAGAUAUCAUUAUUUCUGUGCUCUCUUUCCAUAUUUGAAAAAUGUUUUCA
CUCAAUAUUUUGGCCCUAAACCUUUUUUCUUUCAAUACUAUUUCCAAAAUAAGGGUCAAA
GUGUUAAGGUUGCCUAUUGACGAGUAGGGUAAAGAGAGGCUAUAAUAAUGCCAUUUUUAG
UCCCGUUUUUGCACGAUAACUUUAAUAUAGUCAUUUUGAUACCCCAAAAUCAUAAAUUAA
AGUCCCCUCAGUCCACCCCAUAUUCUGGUUUUACCAACUGUCCUUGUGUGGUCUUAUCUG
CUAAAUGGUCUUUCUGGAGCCCUUUUUUCAUUUUCAAAACUACUUUAAAAGGAAGCAAAA
GGCUUAGUAUUUUUACUUCACCCUCUUACCAUUAAUGCAUAAUGACCUUUGAGAGUGCAA
GGCAAGGAGUAGACUUGUGAAUUGGGAACUAGGGAAACUUGAUACUUGUUUUCUAUCCUA
AUAACCUAUUUUGCAAGGAUGCUUGAGGUGAGGAGUAUGAGUUGAUAUGGGUUUCAACUU
AUCUAUAAGGAGUCCAAAGGUGUGAUAUUCUAAAGGACUGAGGAACAAAAGAUAUUUAAA
UAUCUUUCUCAAAGUUGUUAAAGUAAAAUAAAAGUUAAGGCUCUCUUAUUUGAAGUCUUG
AUCUUGUUUUCUGUGUGCUAUGCCCUUGUACUUGCAUGUGCUCCUGAAUAAUUGGAUUGA
CAAUGUGGUAAUUCUUUUUCUUGUGUGCUUUGUGACUAUUUUAGUAGCGUGUAUAAGUUA
GUUUCUUGUAAAUGGUUGCUUGAUAUCAAUUUGAAGUUAAAACUGGAAAUCAAAUGGUUC
CAUGUCGUCUUUAAGUUUAUAUUUGAUCAUGAUUGCCUAAUGAGUUGUAAACUAUAUUGC
AAUAAUGAUGGACUAUGUACUUUUUACCUGAACGAUACCGGCCUACUUUUCUAUAUUUAU
UCACUGUACUGUAUGCUUAUUGAUUAGGCCAAUGAAUUAAAACUAGUAAUCUUUCAUGUA
AGAUCGCUAACAUGAUUCUUACGUCAAUGUAACACUACAAGUUGCCUAAGGAUGAGAUAA
GACUAACUGAACAGUGUUAAGUUCAUGGUAUAAAAUCUAUUUUAAGUUUGUGUUCAUAAU
GAUUUCCUACCUACCCUACAUGCUAAUUAUCCUCAGUAUUGCCUAUGUAUGUGCCUAUAU
UGAUUGAUCAUUGGACUGUCUAGUAAUGACUCAAUUGGACUAUGUUAUUUAUUUAAUUAG
AAGUCUUAUACUUGCUUACUUGUCUAUACUCACCCAUUGACCUAUUGCAUGCUUAUUUAA
UUAAGUUAAUGAACUAAAAAUUGAUAAUCUCUUAUGCAUAACCCUUAAUGUGAUACCUUA
AUUCAGCAUAGUAGUCUGGAUCAUUUAAAUUUGCUGAAGUUGAGAUUAGAACUGAAUAUU
AAGUCUGUCUUAAGUGAUCAUAGUUUGUAUACAUCCUAGUUGUGUCCAUGACUACGUCAU
UAGUUGUUGAUAUUUCUAUAUGUACAUGCUUGCCUUGGAAACCGUUUAGUUGAUGAAUAA
AGGCUUAAAAUGAAAUAAGAUUAAAUGGGCCUUUUAGGCAUACCUUCUGUUUCCCCUUCA
CGUGUUUGGGCCAAAUUCAACUUUUAAAGUUGAUUGGCCUAUAUUUGAUUGAUAGAUAUU
UGGAGUGGGCCACUCAGGAUAUAUUAUUGAGUUUGGGCUUUGGCCCAUAGUUGAAUUCAU
UUCUAUUUUAAUAUUUAAUCACUUGUUCAUACUUAAUUAUCUUAUUGAUAAUUUAUUUGC
AUUACUAUUACUGGUAACUCCUAACUCAUUCUUAUUUCUUUGCAUGUGACAUCGGGGUCC
UUUGCAUUGGGUUUGUGGAUUAUGGACCAUAUUGGAGCAUUUGGACUUGAUCCGAAUAAU
UUUUCGUGUCUUUCUCUUUUAUUUCAUGCCUUUAUAUAUUUUGUAUUCAUUAUACUUUGU
AUGGAAUCUAGUGAGGGCCCAAGCAUUGUAAAUCUCUGAUAUUUUAUUAAAUAAAAUAAA
U
2. MFE structure-
.((.(((((....((((....((((((.((((((..((((...(((((..((((.......(((((....(((((((((.(((..((((((((((..(((...(((((.((((((..(((.......((((((..(((((((((((((((((...(((.(((.....(((((((.......))))))).....)))))).
......(((((((..((((((..((((.......))))))))))...)))))))............((((((((.(((............((((((((((.((((((((...)))...................((((((((((.((((((((..((((.((((........))))..))))..)))))))).)))))))
))).)))))))))))...(((.(((.(((((.....))))).))).)))..((((((....))))))..............))))............))).))))))))..)))))))))))).....))))).))))))..)))..)))))))))))..))).)).)))))))).)))....)))).)))))..)))))
....((((.((((.(((((((((..((((((((((((.((((((((.((((((...((((((..(((..(((((....)))))..)))..))))))....((((((..((((((.((..((((((((((....))))))))))(((((((..((.(((((....))))))).))))))).)).)))))))))))).))))
)).)))))).)).)))))))))....(((((((((.((((.........(((((((((((..(((((((.(((....(((...)))..))).)))))))...)))))))))))....((((..((((.......)).))..)))))))).)))))))))..)))..))))))).))..))))))))....))))))))).
.))))..)).))))..)))))).......(((((...(((((((...((.((((((((((.((((((...((((......((.((((((((((((((..................................)))))))))))))).))......))))..)))))).)).))))))))))...))))))).....)))))
...(((......)))...((((((((.((...((((((......))))))..)).))))))))......((((((..........((((...))))..(((((..(((((((((.((((((.....(((((((.((....((((((....................))))))...))))))))).((((....))))..(
(((..........((((((....((((((.((...((((..((((((((((..((((((((.((((...(((((((((.........)))))....))))....)))).))))))))..))).((((...(((((((((((((((........)))))))))........(((((..(((........)))...)))))(
((((.....((((((....))))))))))).((((.((((((.(((((((.(((((((((((.((......)))))))))).))).....(((((.((((.((((....(((((.(((((.((((((...((((......))))((((..(((...(((((.((..(((((((((.((((..((((((...((((((...
....))))))....)))))).)))))))...))))))....)).)))))...)))....))))((((((...((((.((((.((.(((((.(((((..((((((((...)))).))))))))).))))).)).)))).)))).......)).))))...((((((((((...(((((((((.........(((((((((.
.....(((((...((((((....((((.(((((......))))).))))...))))))...((((...(((((.......))))).....))))((((...((((..(((((........))))))))).....))))(((((((((((.(((((((((((.(((((........((((((((((((((......(((((
((.((((((((......((((((((..((((((((((.((((((((((((.............))))))......)))))))))).......(((......(((..............))).....))).(((((......)))))))))))......))))))))......)))))))).........)))))))..))
))).))))).))))....((((((((((((((....))))).((((.(((.((........))))))))))))))))))..(((.((((((.....(((..(((((.(((((((......))))))).)))))..)))..)))))).)))......))))).))))......))))))).))))))))))).........
.......)))))..)))))))))..............(((.((((..(((((((..(((((((...)))))))))))))))))).)))..)))))))))..))))))))))..)))))))))))...)))))..))))..)))).)))))..(((.....))))))))))...)))))).))))..........((((((
(.....))))))).........(((((....)))))))))))..))))........))))))).))))...))))))))...))))))))))..))))))...))))))))).))).))..)))))).(((.((((......)))).))).........................((((...((((.....))))...))
))))))...))))).))......................((((((((...))))))))...
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-761.21] kcal/mol.
2. The frequency of mfe structure in ensemble 1.0121e-39.
3. The ensemble diversity 949.19.
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
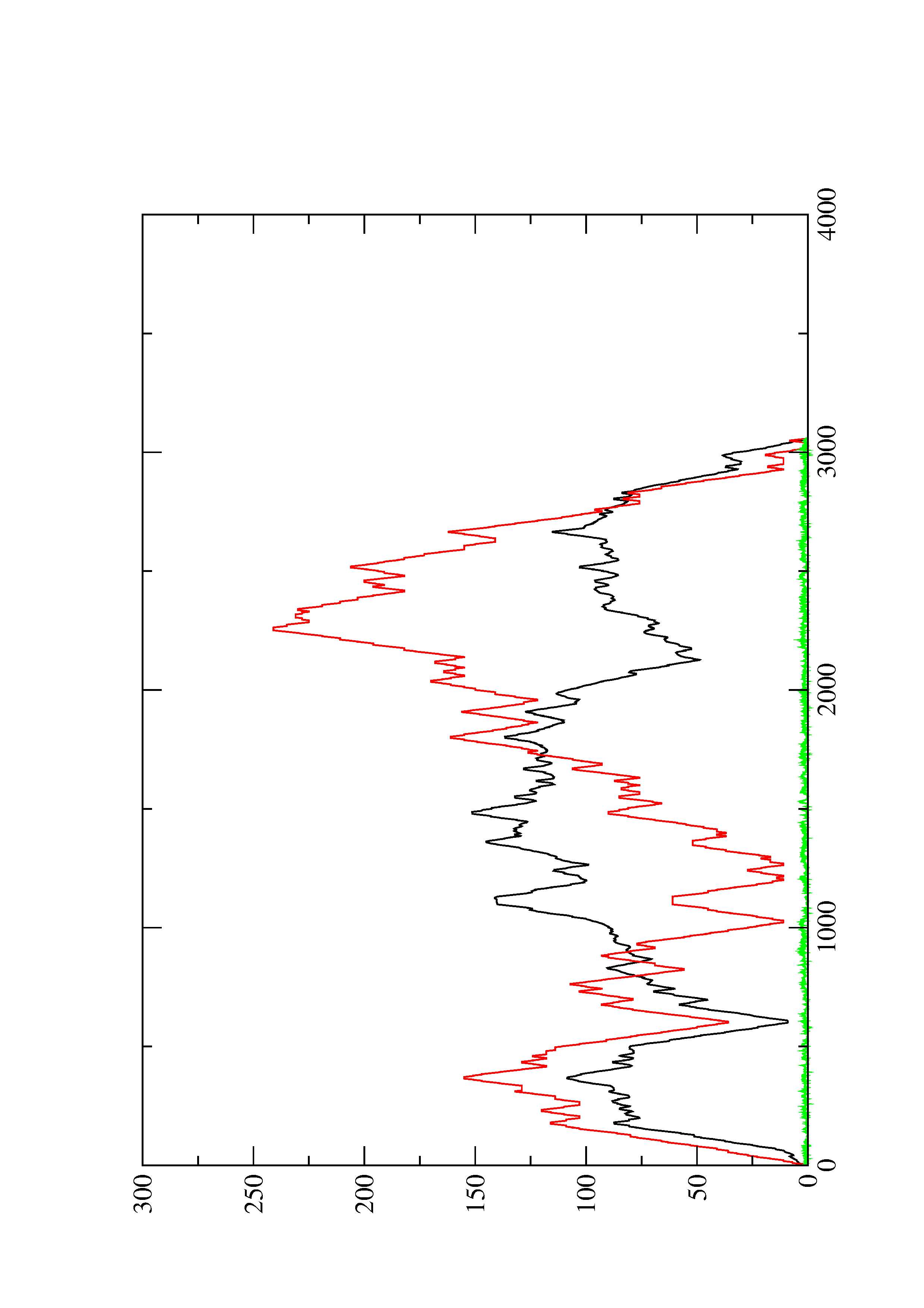
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.