lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01021235.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-1537.30) is given below.
1. Sequence-
AUAAUAGUAUCAUCUUGUUUAACAAUCAAAACUAAUCCUCCUAAAGAGAAAUAGUAAUAU
CUAGCAACAACAUGUUAAAAUAAUAUGAGCAAUUUUUAAGAGCAGGAUAACAUUAUAACA
GCAUACAUUUCUUAACUUAAAUAAAAUUAAAAAGGGUCCAACACGCUUCCAUACCUGUCC
CGUUUUGACCGCAGUUAUCUCGGGUAUCCGUCGAGUCUUUUACCGGACAUUCCAGUCCCA
ACCUAUUGCUAGGUAACUACCCUUGACGUUCCUUCAGGAUAAACCAGCACAGCCUAGUUU
CGUUUUCUACCUCUAGGCGUAAGGUGGUAUAGAAGUGGGAGUCUUUUCUUUUUAUAAAAG
AGAAGAAAUUAUAAAGCUAUAAAAUUUAUGCUCAAGAAAGUAACUCAAACUUUUUAGUUU
UUAUUUAUAAACAUAGAGAGUAGAGGAGUUUUUGUUCGAUUGAUGGAUCGAUCAACUUAU
GCUCCUCAAUUACAUACGAACUCCUUUUAUAGUAGAAGGAGUUCGGAAUUCAAAACUUCA
UUUAAUCAAAAUUUACAAUUAUUGAAAAAUUGUACAAUUAUUGCAUAUCUUUUCUUCCAU
UGCUUGGCUGACUCAUCUUCUUCUUUUGCUUUUUUGAAUUUUGCGUCUGAUCCUGUUUGC
UUUCCGUAUUUCUUAACGUCUGCAUCCACUAUCUUUUGUUGCUUGUCAACUCUUCUAUCU
UUCAUUGUUGCUUGCUGCAUUAUUUCUCCAAUUUUCUGACUGUCGUCCUUUUCAUCCAAG
UUGUAUUUCAAUCUUUAUAAUGAAAUAUCUUCUUUUUGGUAGUGCCUUAUCAAUAUUGCU
GGUUUAGAAAUAUCUAAUUUUAUAUUUUUAAUCGGAUGAUGUUGACAUCGAAUCCUCUAC
CAUUUUUCCUUUCUUCGGAGAACAGAAUUCGCAAGUUCCUCGACCAUGCUUUCUCAUUUC
GCAUAAUUUUUGUUGAGCCUGGGGACUUGAUUUAAUUAUUCCUCAUUCAAUCAAAUUCAU
CUUAGUUUCUAAAUAUCUUUUCUGUUCUCCGAAGAAAGGAAAAAUGGUAGAGGAUUCGAU
GUCAACAUCAUCCGAUUAAAAAUAUAAAAUUAGAUAUUUCUAAACCAGCAAUAUUGAUAA
GGCACUACCAAAAAGAAGAUAUUUCAUUAUAAAGAUUGAAAUACAACUUGGAUGAAAAGG
ACGACAGUCAGAAAAUUGGAGAAAUAAUGCAGCAAGCAACAAUGAAAGAUAGAAGAGUUG
ACAAGCAACAAAAGAUAGUGGAUGCAGACGUUAAGAAAUACGGAAAGCAAACAGGAUCAG
ACGCAAAAUUCAAAAAAGCAAAAGAAGAAGAUGAGUCAGCCAAGCAAUGGAAGAAAAGAU
AUGCAAUAAUUGUACAAUUUUUCAAUAAUUGUAAAUUUUGAUUAAAUGAAGUUUUGAAUU
CCGAACUCCUUCUACUAUAAAAGGAGUUCGUAUGUAAUUGAGGAGCAUAAGUUGAUCGAU
CCAUCAAUCGAACAAAAACUCCUCUACUCUCUAUGUUUAUAAAUAAAAACUAAAAAGUUU
GAGUUACUUUCUUGAGCAUAAAUUUUAUAGCUUUAUAAUUUCUUCUCUUUUAUAAAAAGA
AAAGACUCCCACUUCUAUACCACCUUACGCCUAGAGGUAGAAAACGAAACUAGGCUGUGC
UGGUUUAUCCUGAAGGAACGUCAAGGGUAGUUACCUAGCAAUAGGUUGGGACUGGAAUGU
CCGGUAAAAGACUCGACAGAUACCCGGGAUAAUUGCGGUCAAAGCAGGACAGGUAUGGAA
GCGUGUUGGACCUUUAAUUUUUGUUUAAGUCAAGAAAUUUAUAUUGUUAUAAUAUUAUAU
UGCUCUUGAAAGUUGCUCAUAUUACAAUAAUUAUGUUAUCUUUAGACAUAACAUAUUCCU
CUUCAGGAGGUUUAGUUUUAAUUAUUAAACAAAAUGAUAAUACAAUCAGAUAUGAAAACA
UACCCACUAGGAUAAUUAAUAUGAACCAGUAUUUUUCUGGACCACCCCCCUCAGUUGGUA
UCAGAGCCAUGAUAGAUGUUAAAGACUUAGAAAUAGAUAUAAAAGGAAAAUUCAGUAUUA
GAUAUAAAAAUUGAACAAUUAUCUAAAGAAGAUAAAUUUGAUAAAAUAUUUUAAGAUCUU
AGAUGCUAUGAAAAUUUUAAUAAUAUAGAUAUAGAAUGAAUAAAUAACCUUAUUUUGG
2. MFE structure-
.......((((((.((((((((.(((.((((((((.((((((.(((((.((((((.((.(((((....(((((((((...(((((((((((((((((((((((((.((((.((((((((((.(((.(((((((.((((((((((((.....(((((((((((((((((((((((((((((.(((((((((((((((((((
((((((((.(((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((
((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((
((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((
((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((.((.((((..(((..((((...........)))).....)))..)))).)).))).((((((.(((....
....))).)))))).......((((...)))).......)))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))
))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))
))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))
))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))).))))))))))))))))))))))))))).))))))))))))))
)))))))))))))))...)))))))))))).))))))).))).)))))))))).)))).)))))))))))))))))))))))))...))).))))))....))))).)).)).)))).))))).)))))).)))))))).))).)))))))).))))))......(((....)))....(((((.((((((.........
.((..((((.......))))..))....))).))).))))).....(((.(((((..((((.....(((...(((..((((.....(((((((((((((((((.((((((((...(((((((((.....))))))..)))....))).)))))..))))..)))))..)))..))))).....))))..)))....))).
....)))).))))).)))
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-1551.63] kcal/mol.
2. The frequency of mfe structure in ensemble 7.99861e-11.
3. The ensemble diversity 142.54.
4. You may look at the dot plot containing the base pair probabilities [below]

III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
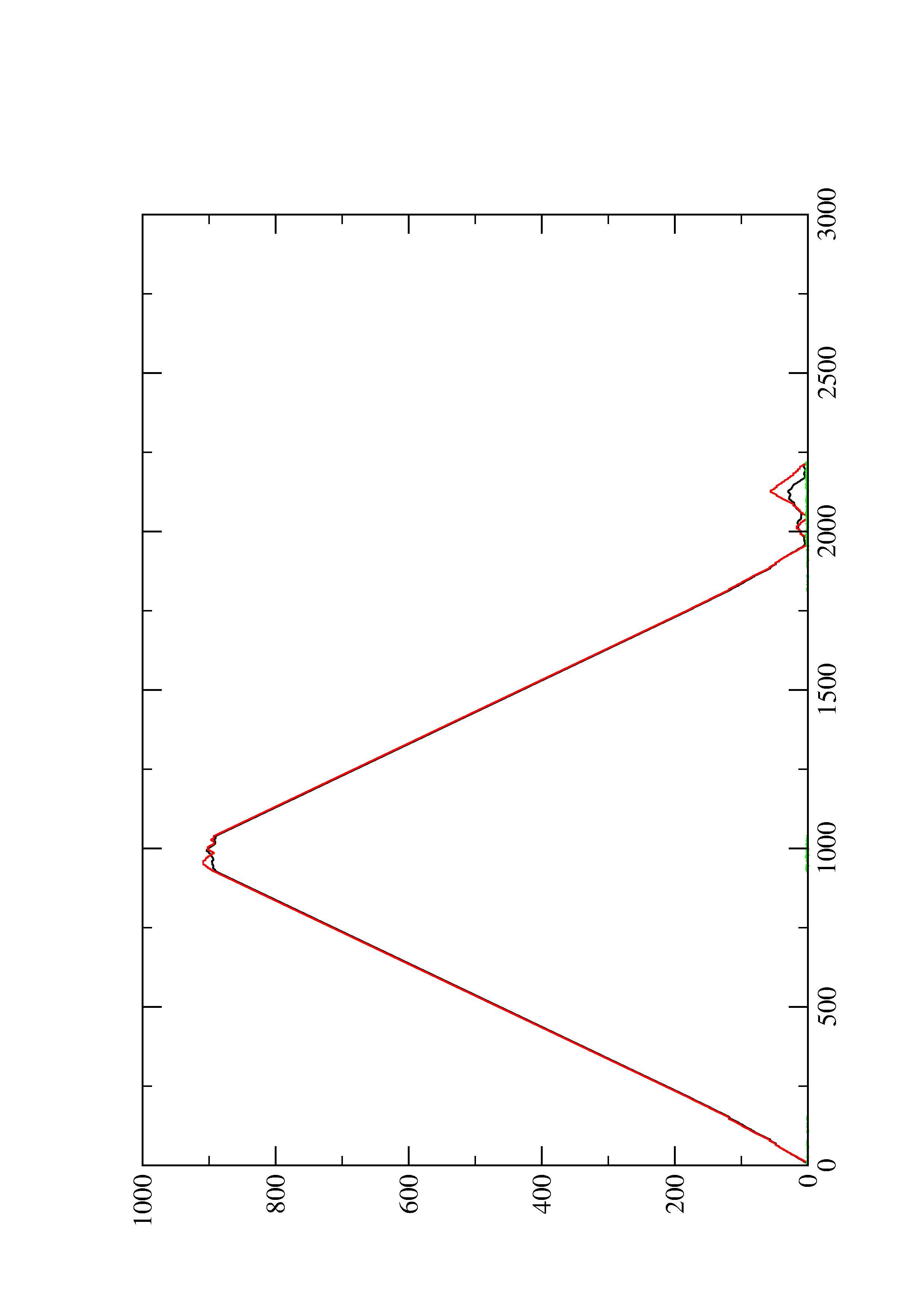
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.