lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01025044.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-515.60) is given below.
1. Sequence-
UUUUAUUCGUGAUAAGCAAAUAUUAGGCCAUUUUGGUUAUUGCUUUACUUUAUUUUUUGU
ACAUUAAUUUUUAUCUUUACUAUACUUGUGUUUAUGGUAUUUAUUUCCUCAAUUGAAAAA
AUGGUUUCAUCAGGCAACCACAUUUGUGGAUUCUGAAUGAUGUCUAACCCGUACUCUUCG
GAAUAAUUUGAACUCUUACCUAGACUCUGGUUUUCAAAUAAAAGAUUUUUUAAAUAAUCU
UUCAAAACAAUUUCUGGAUUUUCCCUAAAGUUUGGUAGCGACUUUCUAAAUAAUACUUUU
AGCCACCUCUAAAUUGUUUACACUUAUUAAUUAUUUUUUGAGUUCAAAGUUCGUGCCUUA
UCAAGAGCUCAAACUUGUAUUAUUUGAUUCCUUUUAUUGUGUUGGAGCCUUUGUUAGAGC
GAUUCGAGUUGAUUCCUAAUAGUUUCAAGCAUUAUCUCCAGAUGUCAGUUAGGCUAUGAA
UUAUCAAUAUUAGAAUUGGUUUAUAUUAAAAAUAAUUAUAUAGCUAAUCCUUUUGGAUUU
UAGGAUUGAUACCUUUGAUCCUGUCUGCUUUUUUUAGUUCGUUUCUUGUGAUCUUCCCUU
CGGUAUUGUGAUAUUAUUAGAGAUUGUCAUUAUGGCUUUGUAUUGGCCUUGUGUUGACUC
UGAAAUAUAUAUGAUGAGAUUUUAGGGAUGUCCGUGAGCCUAAAUAUAUAUAGCUGAUUU
CUAGUUGUUGUAGAAUUAUCUCAAUCUAUGCAGGGCUGACAUGAGUGACUAUAAUCUGUC
AGUAGUAAUUUUGCAUGUUAUUAUCAUGAAAAUGGUUUUUAUAAAUUAUUUUCGACUAUU
UUAAGAUCGAGAGCCCAUGAUCAUUGAUUUCAAAGCUUUGCAUGGCCUGUCAGUUUAUUU
UCAGUAGUAUAUGAUUAAAAAUGAUUAUAAGUUGAUGGUUUCACCUAAUUUUUAUGAUAA
AGCAUUGAUGAUAGACUUUUACGUUAUUUUCAAAUUGAUACUAGACUCAAUUGUGCCGUA
ACACAAGAUAUUGAGUUUUAUACUCAGUUUUUGACUGGGAUGAUGUCGAGCGCAGAUACG
AAGACUUGAUACUAUUGGUAUAGACCAGUCAAAAGUCUUAAGUAUGUUAACAUUCAUAAG
UUAGUAUGCGGUCUGUGCGUCUCCAUGGACCUUGCUGAUCCGGCUAGUGAGCGAUCGGAG
GAAGGGAUACAAAAGAGUAGAGGUAUACGAGUCUUUGCACCUUCCAUGAGUCCUGCCUGC
UGGACCUACGGGUAUUGAAUGAGGACAAUACAUGUUUUGAUUAGCAUUACAUUAUAUCAU
UUUGCGUACUUCUAUUCUUAUUUGGUGUUUGUUGGUUGUACUCUCGAUUGUAUUGAUGGU
CGCGGAGUUUAUUUUUAUUUGUAUGUAUCUCUCGGGUUUAUGGGCGCUCGUUGGGAUGUA
GUUUACUUAUUUUGUUGUUUAACUUCUUGUUUAGUUGUUGGCUAUAUUCUUUGGGUUUUG
UUCUUGUCGUGUAUUAUUUCAUGACUUGAUAUUAGACAUUUUUUCCUUUUGUCCUAUCAU
UGAGGUUACUAGUGUACCGACAUAUCUGCUUUCUAUUAAUUAUAUAUUCUUGAGUAUGGA
UCUAGGUACGUUUCUAUCUCUUUUACCUGAAUCUCCAUCCUAUGAUUAGCUAGCUAUGGG
ACUUUUUGCCUGUUUUGUGUUCUAUUUAACUAUUUUAUCUGUGUUUUUCUCUACUUGUUU
CAUUUCUGUGUCCUCCUUUUAUUAUUUAUCCAUUUAUUGGUUUUAUUUCUUAUAUGUACC
UAAUUGGUUUAUGAUGCCAAGUCGAACCUUGGUUUUCUUCGUUCGAAAGAGAGGGGGGAA
GAAGCGGGGUAGAGGAAUGGUCAACUCAUCAGGCUCAUGACUUGAAGAUUGCAGGUUCGA
AUCCUGUCCCGAAUACCUAAUUGCUUUGGGUAUUUAGUACUCAAACUAAACUUCUGCCUU
UUUUUAGUGCAAAUCCUAGUUCUAGUCAGGAAUGCUGAGUUUGGGAUUCGUGUCAAUUGA
AUUCGGCUGAAGUAAAAGAUGGUUCCUAAGGAUUGGAGUCUAUCUUUCUCUUCAUUAUUU
UUAUUUUCUGUAAUUUUAUAUUUUUAGAGACAUAUGAACCUAUUUCUUUUGGAUUAUUGU
ACUUUCCUUUGACAUUUCUCACAUAGUGUCAGG
2. MFE structure-
........(((((((.((((...........)))).)))))))...............(((((.(((((........(((((((........)))))))..........(((..((((....((((((((((..((.((((....))))....))..)))))((.((((.(((.(((((((....(((((((.....(((
........))).))))))).(((((((........)))))))......(((((..((......))..))))).((((..((((((.((((((((....((((......)))))))))))).(((((..((....))..)))))..))))))..)))).....(((.((((.(((..........................
))).))))))).((((((((.....(((((((((..............((((((((.(((((((((((((.(((((((((((((((((......)))).............)))))).)))))))(((((....))))).(((((((((.....))))))))).((((....(((((...((((..((.(((..(((((.
.(((.((((.((((((((..(((..((((.(((((((.......))))..))).)))))))..)))).)))).)))).)))..))))).))))).))))))))).....(((((((.....))))))).(((((..........))))))))))))))))))(((((((((.(((((((((((((((((.......))))
))))...(((((((((((....)))))))))))...........((((((((........)).))))))((((.(((((((((((((.((((((((((..(((....((......))....)))..))))))))))))).............)))).)))))).))))))))))).)).)).)))))))..(((((((((
..(((((((((((((......))))....)))))))))..)).)))))))....)))))))))))).(((((((((((..(((((((......((((((....))))))...)))))))..((((((((((........))))))))))((((((((......))))))))..((((...))))((((.((((((((.((
((.(((((.(((((((((((((((((((.(((......((((((((((((.((((((....))(((.((((((((((((((((((......((((..((((..............))))....)))).......)))))))))))))))))).)))...((((((((((((....))))))).)))))............
............)))))))))))..(((((..(((((((((((................(((((........)))))..))))))))))).))))).........((((((........)))))).(((((...((((...........)))).)))))..))))).))).)))))))(((.((((((...(((((...(
((........)))..)))))..)))))).))))))).))))))....)).))).)).)))).))))).))).))))((((((................))))))...........)))))))))))(((((((((((((((.(((((...((((((((((.........(((.....))).............(((.(((
.....))).)))((.((((((......))))))..)).......))))).))))))))))))))).))))))))))..))))))))).....)))).))))))))))).))).)))).)).(((((....((((((((((.....))))))))))...(((..(((((......(((((......)).))).....))))
)..))))))))..((((((((((((((....)))..)))))))))))(((((..((((((((((((........)))).))))))))..)))))..........((((((.((........)).)))))).....)))))..))))..)))))))).)))))......((((((((........)))))))).
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-554.45] kcal/mol.
2. The frequency of mfe structure in ensemble 4.18024e-28.
3. The ensemble diversity 752.46.
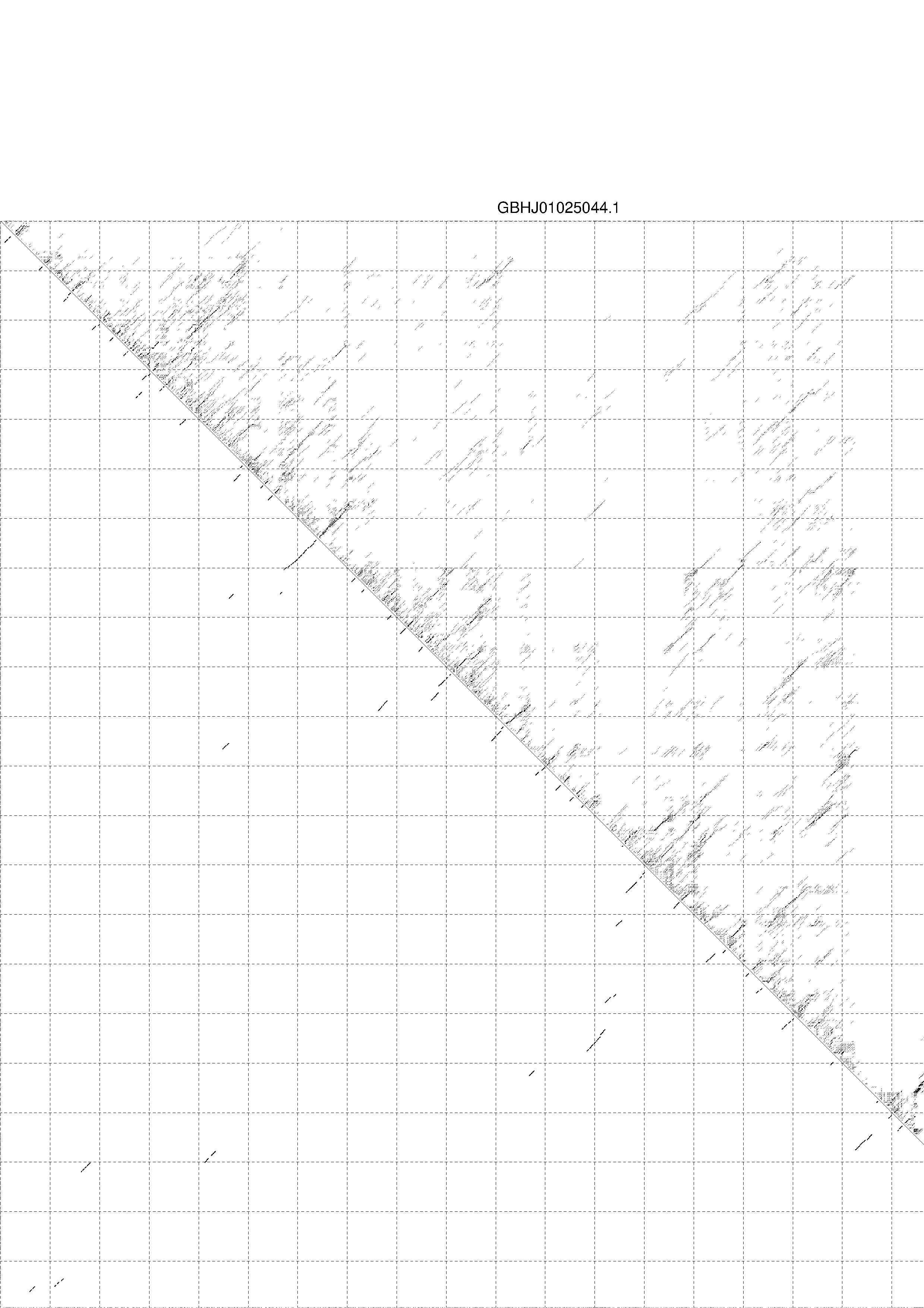
4. You may look at the dot plot containing the base pair probabilities [below]
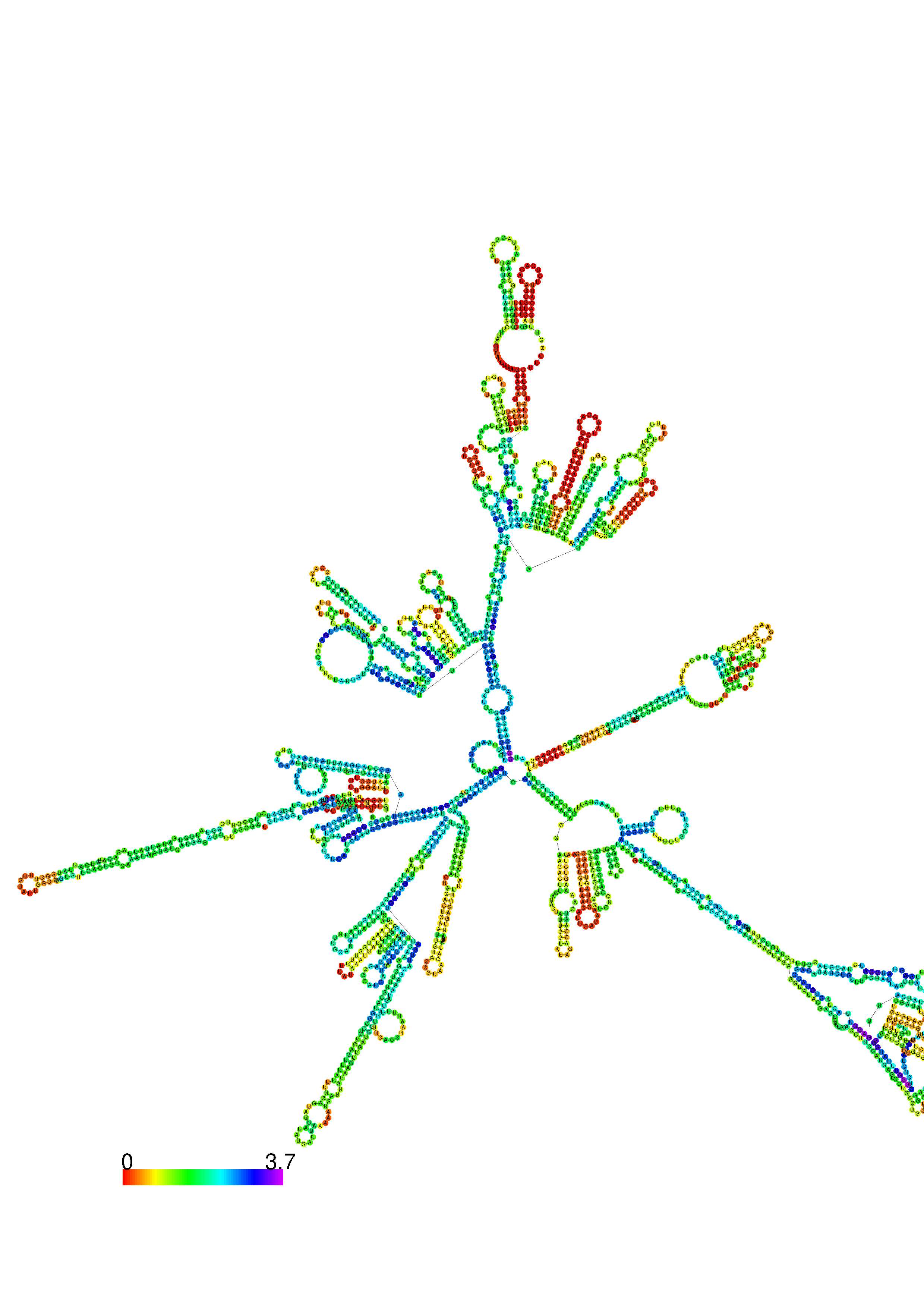
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
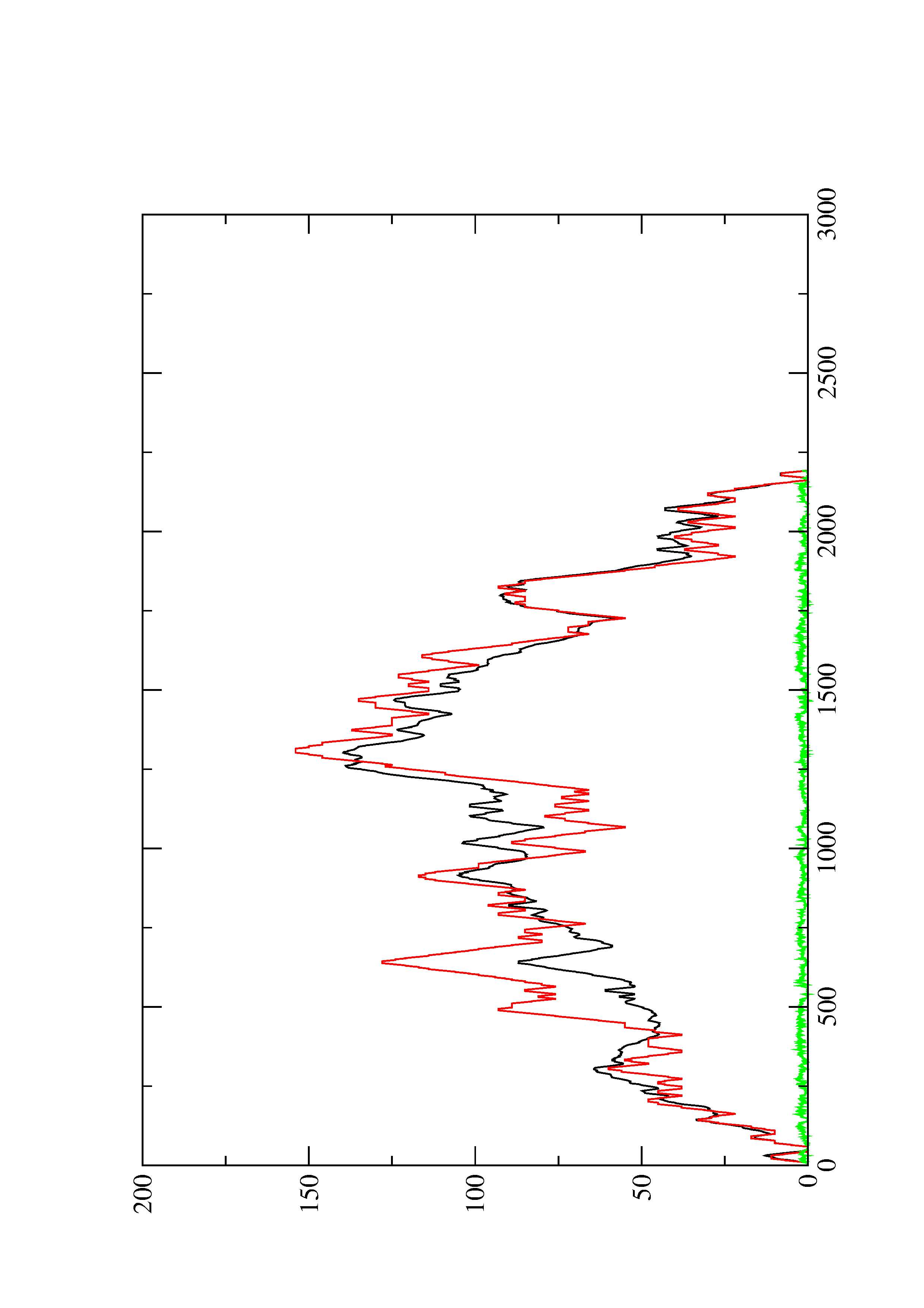
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.