lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01025889.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-648.00) is given below.
1. Sequence-
GUUAAAAAGAAAAAGAGCCUGGAUAUCGGCUUCGGGCCCAAUUCUUCUUCUUUUGAAAGU
CUUCGCUUUUAUGCCUUCAGCCCUUUUGGGCUUGGAUGGAGGCAGACUCGUUUAGGAUUU
AUCCCGAGGAAUGCAAGAGUAUAAGUCCCAAUUGGACUCCAUCAGGGGAGUAUCAUGCAA
AUAAAGAAAAGGAAUUAGCAAAUACCCUUGUAAAAAUUAAUCAUACAACUAGUAAUCAUA
UAUUAAGCACAAAGCACGCCACACGUCGUAAAGCUUGGAAAGAGUUGCCCUAUUUACAAA
UCAUAUUCCUACAAUGGAACAAAACACAAAACCAUUAACGAAAAUCCAGGUCACUGUGAG
UUAGUAUAAUCUCAUGUAAUACGACCAUUUUAACAAUUAACUAACAUUUAAGUUCUACCG
UGGGAGAUGAACAAAUUAGCCAUAAAGCUUUAACCUACAAGCCGACAAAGGUUAUAAUUA
GCAGACCUUGAGGAAAAUGAAGGUCUCUGGUAGAAACUUAGGCCUCUAUUGGACAGAUUA
AACAAAUUCUAAUAUCCUAAUGUAAACUCAAGAUUUUUCACAACAUUUGUGAGAAAAAUC
CACCCAUUAUGGUAGAGAUAACAUAGUUGUUUCUCAAACUUUACCACUGCCCAUCAUGUU
CAAACAAGUUUCACAAUCUGCAUACACUAAAUAUUGGAGCCAAUUCUCUAAGACAUAAAC
UUCUUACUUAGAUCAAUUGAAGCAUCAAGACAUGUUCCAAGAAAAAUCCAACAUUUAGGC
AAGUUUUAGUUAUUUAAGGUAUCACAUAUAUAUAUAGGCAUCUAAGCAAGCUAUUGGAUU
CUUAAAAUAGAUUAUGACAGUUCAUUGUUUUAUCUGCAAACAGCGUAGGUACUUACAAAU
UCACCAUGCUUAAGCAGCUACCAAGUAUACGUAAAAAGCACAAAAGCAUCCAAAACAGGU
CGCAGGAUAUACGGUCAAAUUUCUUAUUUCAAAGUGUGCAAACAUUUAAAUAGAAACACA
UAGCAUUAGACACCCCUUCAGAAGUUAGACAAAAAGAUUCUUACUGACAUUAUUAGACAA
AUUAUCCCGCUGAAUUUCAAACAUGUCGUGCCAAUAUAAUACUGGUUAUUAUUACUCCUA
CACACAUUUAAAUGCUAGAGAUGAUCAAACAUGCUAACUCAAGUUCAUGCGAUACACUUA
AUAGAUUCCACAUUUUCUCUUAAACAUUCUUGUAAGAACAAAGACAUCUUUUAAAGAUAG
GUUUCAACUUAGCAUAUCAUUCUCAAUAAGUUUCAAAGAAUAGGUCUUUUAGUAACAGGU
UUGAGAUAUACAAACACAUUUGUCUCAUAUUUACACUCAAAAGGAACAAAUUUUAUUCGG
UAUAUACAUACAAAUGAAAAGUUCAUGUAAUCAAUCAUCCUAUAUUUUAGAGCUAGUAUG
UCAAUCCAGGUUAGGAUUAAACUACUAGGUGAAAAGUGAUUACAUUUAUUCUAAGUACAC
AUAGUCAAAAUUGUCGGGACACAUUGUUACCCUUAUUUUCGCAUGUCUAAACUGAGUUUU
GUACAAAGAUUUUUGUUUCAAAUGCACCGGGAAUUUAUUUCAGUACAUAUGCAUGAUUAA
UAAAUAGGCAAGUAUUAUGCACAUGUGAGUACAUAUUUUCUUUCAUAAUGUCUAUUCAAA
UUUAAAAGUCUUAGACAGGAAACCAACUGCUUAUGAAACACUUUCGAGAAGAAUAUUGUU
CAAACAGUAAACCUACACAAUUGCAGCCAAGAGGAGACUCAACUGAACAUGGUAUUGAAU
CUAAAUCAUGCAAAUUCAAUUCAAAUAAUCAGGUCUAUCAUACAGACUUAUAUAUGUUGC
ACUCCACACACUCUCGGAAUAACAUAGCUUUAUUUAGGAAUUAUCAAACUAAUACUCAAA
UUCAAUAACACGCAGACGAAUAUAAUUUUACUAUCAUUUGGAGGAAAAAAUAUAGCAUGU
CAUUAAUCAAAGUAUCAGCUCAAGCAACAUGGUUUGCAUUGCCCAGUUUGAAAACAUGUC
CAGAUAAUAUUUCAUGCGAAAUCCAUACCAAAAAUUAUUUGAACUUAAAAGGAGUUAAGG
GACAAGCAAAUUGGAACCUAUACAUUUUAAUUAAGGAAUUUUCAGCUUACCAAUAAGGUU
AUGCCAUUUUUUCUAGAAGAGUUAGAUAGCUUACAUCCAACAUAGAGAGACCAAAGAUUG
AAACCUGGUCUAUUGUACCCUCAAAUUAAAGGUAAAAAAGGUUUUAUAGAAUCUCCUGGU
GCCCUUUAAAGUCAAUUAGUAGAUACAUUUUAACUCCAACAGAAUACACCAAACAGAUUC
UAUACACCCUUAUUGAUAUGCAGUUAUUUAUUCCUUAUUUUUUGAACCAGCUUCAUUAAG
ACAAGUUGGUUUAAACUUUAACUUUUUUAGUCACUCUUUAGUCACACAGUGGCAAUACAC
AAAAAUUAGAGCCCUCAAAUGCUUAUUUUUAGAUGAAAUAUCAUGAAAAGGUAGGCAAAA
GCUCAUGUCCCGUACUCCAAUUGUUAUACUACCAUGUAUUCUCACUCUCCCUGUCAAUUA
CAGCAGAUUAUCAGAUGAUCAUGCUAAAAUAAAGCAACAGCUAGGCAACCACGUUCCAAA
ACAAACAAGAGAAGGUGAAGAAAAAUGACAUUUUAUAGGUAGGGAACUCAAAUCAAUCAA
AGUGAUUCAAGAUAGCCUUUGGAUCAAAUUAGAUAGGUUCAGCAGAAAUUGCCAAAAAAU
CAAUUUUAGUGCUAACAUGCAGUUUGGAACUAAACCUAUUCAGUACAUCCAUACUUGUUU
CUUGAUUGAACCCCCUAUUUGUUUAAUCAGAGCAGGAAACAAUAACAGGAACCAUAUGUU
AAGGGUAUAAUUUUCAUUAUAAUUUAAACAAGAAUAAAUACCAUAAAAGACCCAACUUAC
AAGAAGCAUCCAGUAUAUUUUUACACCUGCAAUAAAUUAACUUAUUAUGGAAAAAUUGAC
UAAUACAAGAUCUAGGCAUGUUAUUAUCAUUCGCCUGAAACAUCUCUUACCGUAGCAUUA
AGUCAGGUAAAGAAAAUACUAAAACACAUGUAAUAGGUAGACAAACUCAAGAUUGCGAUG
CUAUCAUCUCAUAAAUUAUAUAUUGAAUUAAGAUUUCAUACUUUUCGAUUUAUCAAGGAC
AGUAAAUAACAGAUAUUAGAUCUCAAAAUGCCUAAUACCCAACCCAAAGCAUAAACCAAC
AUGCUUAAGAUAGUAGAAACAAACAAUCUGGACCUGAACACAUUGUACAUACAUAUCUGC
ACAAGGCAGUUCACUAUUCUCGUCCAACAUCCAUCCUUACGCACUCAAUCAAUAGUUUAU
AAUAAACAGCACCAAGAUAAUUACCAAAUAAACUAAACAGUUAUCCGAAAAGGAUAAAAU
CUCACCUUUUAGAGGAGAAGGAAC
2. MFE structure-
....(((((((.(((((((((((.......))))))).....)))).)))))))((((((....))))))...((((((((((....))))).((((((((((((((..((((.(((((((...((.(((((((....)))).(((((.....))))).....(((((.((...((((..........((((........
.....))))....................(((((......))))).((.....))..((.......((((..((((((....((((((............((((.(((((((..((((((....((((..(((...............)))...)))).((((((.((((....(((...............))))))))
)))))......))))))..)))))))))))........(((.(((((((..(((.....((((......)))).......(((((.((((..(((.((((.((....(((((((.(((((((....(((((((..............))))))).))))).))..........((((((((((...))))))))))....
.(((......)))(((((((((...))))))))).....)))))))....)).)))).)))...))))........))))).....((((((.(((((..(((((.((((((..............))))))..))))).(((((......)))))..........))))).))))))....)))..)))..)))).)))
..............((((.((((((........)))))).))))...(((((...(((((....)))))..))))).....(((((.(((............))))))))...)))))).)))))).))))......))......((..((......))..))..(((.....)))...(((((.((((..((((((...
.)))))))))))))))......))))...)).)))))(((((((...............))).)))).................))))).))))))).))))..))).)))).........(((.(((((..(((((((..................((((((((....((((..(((....)))))))........(((
(.(((..........))).))))....(((((((((.........(((((...))))).(((((...((((((((((..((((......(((((...((((((((((((((((...((((.(((((((............))))))).))))..))).)))))((((...(((((((..((((....)))).))))))).
))))(((((((((.((((.(((.....)))..((((((.((.(((((......)))))..))))))))))))....)))))))))........(((..((((.((.(((((....((((((...)))..)))..))))).))))))....)))(((((((.....)))))))..((((((((((((..(((((..(((..
...(((((.((((((((................)))))))).))))).....)))..)))))......((((((.................))))))((...))..(((((((((((((((((((........((((((....)))))).........((((((((...((((((((.........((((((((((((((
.............))))))((((((((((.((((((.......))))))...(((((((...(((..........))).)))))))...((((((.(((....(((((((........................((((..((((.((((.((.((....)).)).)))).))))...))))..((((((((.(((..((.
...))...))).)))).))))...))))))).......))))))))).(((((....)))))...........)))))))))).......(((((...(((((((((..(((((((........)))))))((((((((....((((((.((((((((((((...(((((((...)))))))...)))))..........
.......((((((............))))))....((((...........)))).....(((..((((((((((..(((((..(((((((((..(((....)))..))))))).......))....)))))...)))))))))).)))))))))).))...))))....))))))))...(((((((((((((.......
..)))))))))))))((((((.((((...((((((............)))))).......)))).))))))........(((((((((((..((((....))))..)))))))))))...)))..))))))..)))))...........))))))))..)))).))))..))).)))))..((((((((((...))))))
.)))).......(((....))).((.(((...))))).............))))))).............)))))))))))).(((((...(((........(((((((((......))))))))).....))).)))))(((((((((((.........)))))))..))))....)))).))))))))...)))))))
)((((......))))(((((..(((((((((.........)))))))))))))).))))).......))))..))))))))))(((((....(((................)))...))))).....)))))...)))))))))((((((.(((..((((((...((...(((((......)))))..))))))))..))
)........(((((.((((((((((.((.((.((((((.......((......)).......)))))).)))).)))).......))))))..)))))..................)))))))))))))).......(((.((((((..(((........))))))))).)))..))))..))))))))))).((((((.
...........)))))).........((((((........)))))).((((.((........)).))))((((.(((((...((((.((........)).))))....)))))........))))....)))))))................((((((((...........................)))))))).....
((((((.....))))))...(((........)))..)))))...
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-710.02] kcal/mol.
2. The frequency of mfe structure in ensemble 1.98848e-44.
3. The ensemble diversity 922.88.
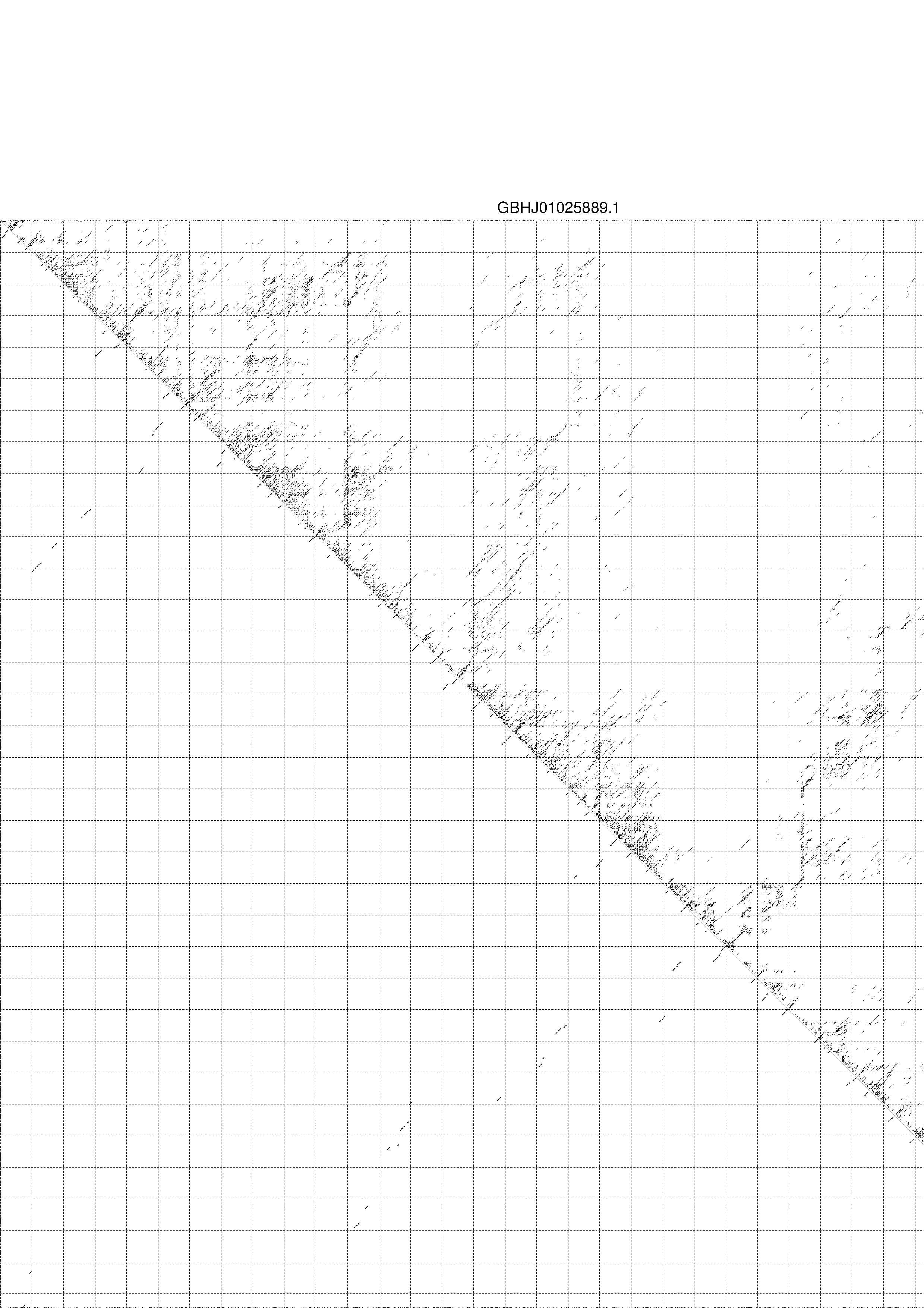
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
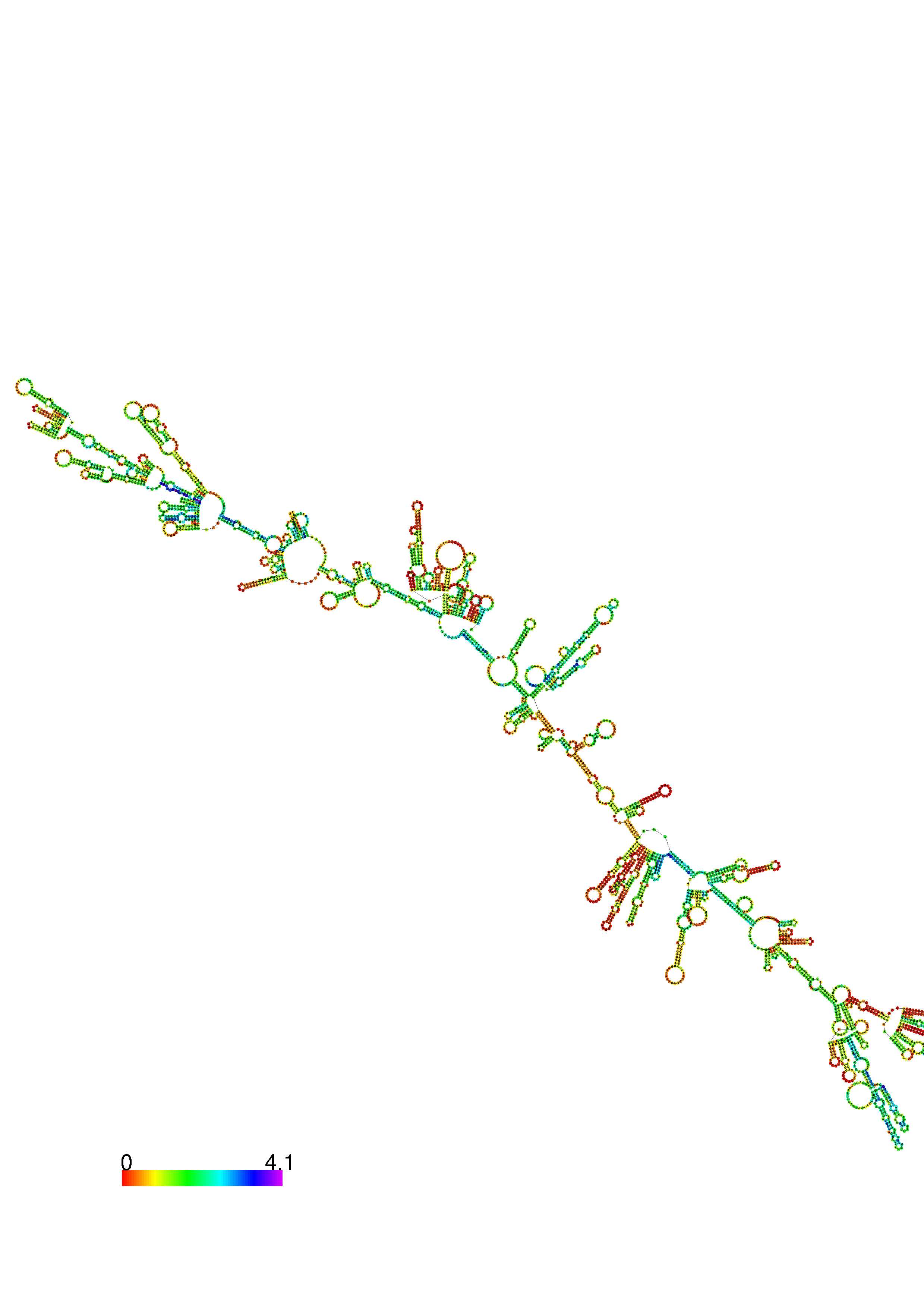
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
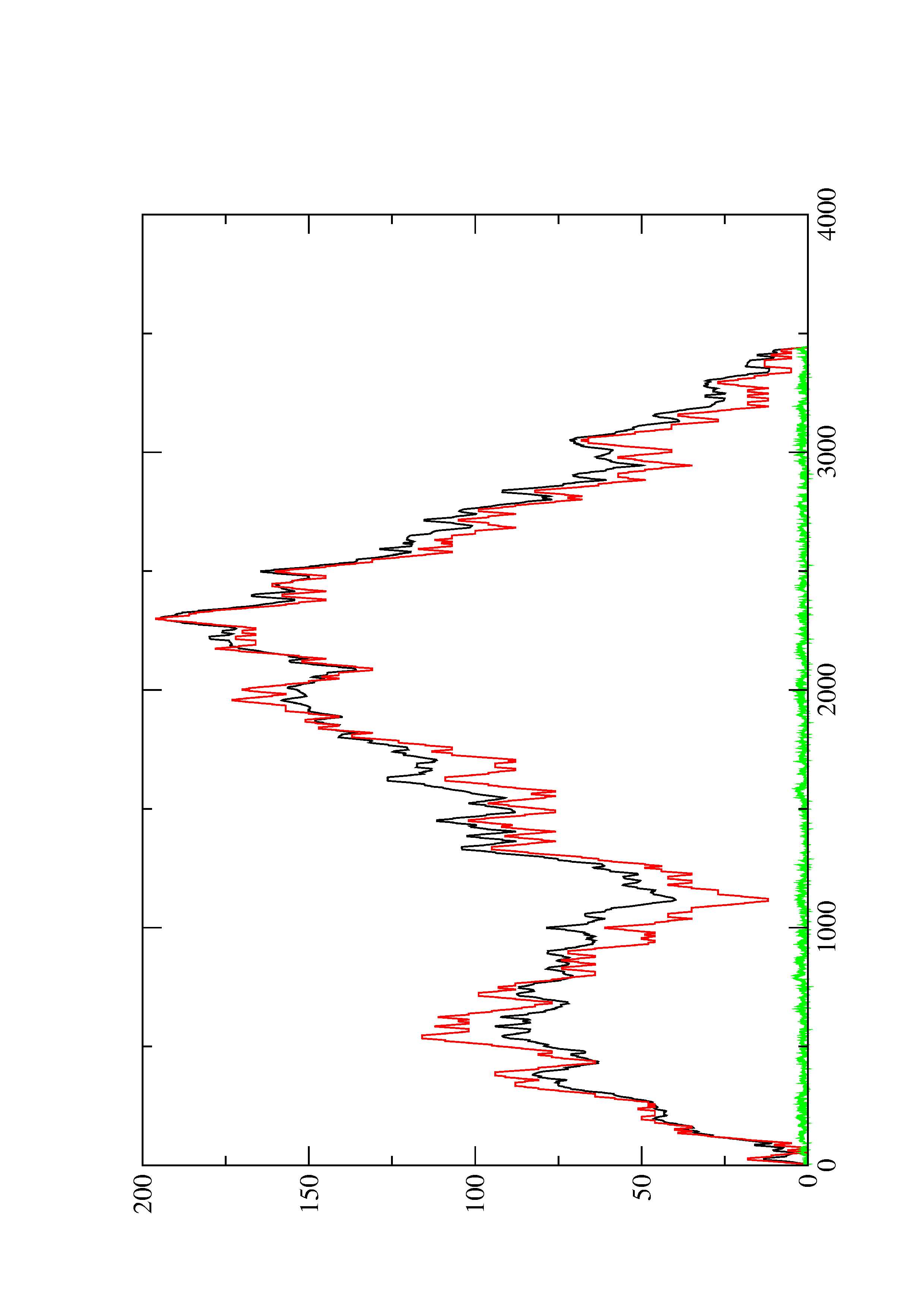
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.