lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01026892.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-910.00) is given below.
1. Sequence-
CGGGUACAUCUCUUUCUCUUACCUCAUUCUCUUUCUUCAAUUCCUGUAGCCAGAGAAAGA
AACCCUCGGCGCCAUUUAUUUUCUGUGUCAAACCCAUACAUAAAAUGACCCCGAGUCCAC
CACAAUUCCUCAUCAAAAUGACAAUCCAUUUUUUGGAUUCCGUACAACCAUUAUAACGCA
UCAGUUAUUCCGUCGUUACAGCAUAAUAGCUGAGUUGUGAGGCUUCUACAAGUUUCUUUG
AAUUCUCAACAACGAUAAGUUCAAUUUCUCUCUUUAUUCAAAUUGUUAACGAGUGUUUCC
GGUACGUUUUUAUGUGUGGUGUCGAUUUUUUAUUUGUUUUGGUUUCAAAUUUCAAAUGGA
AAUCCUUCGAGGCUGUUUGAUUCCAGCCUCCUCGUACAAAUGAUGUGUGAUUUUGACACC
UUUCCCUUCUUAUCUCCGUUUCCCCCAAUUUCAUUGUCUUGGAAGCCUUAACAUUUUGUU
CCUUUGUAUACCCCUUUAUGGGUAUGAUUUUACGAAGCCAGCCAUUAGGGAAAUUUCUAG
GGUUCCUUUGUUUCUGAGGAAACGCUGAUAGGGUUUCACAAAUCAGAGGUUCUUCUUUAA
GAGGUUCUGAAAGGGAGAGGAAAGAAUGAGGAUGUAGGGGUUAGAGGUUUUGCAAGGGUG
GUUAACUCUCUCUCUCUAAAUCAGCCAAUGAUUUCCGCAUUUUGUAAGAACCCCAAAAAC
CCUUUUCUUUUUAUCGAUUUUAACUGAGGGAGUAAUAGAAAGAGAGCUUGUUUGGAUGGU
UUCUGGUUGCGUAUUGUCUCAAAGAGUUUCUGUGACUCUCAACUCUUUUUUUUCUCUUUU
CUUAUGCCUCUGGCGGAACUUUGUAAUCUGCUAGUGGGUGUUUGAAACUGUGAGGGUGAG
UAUAAUCGGGGCUUACAAGAGACGAGAUGAUCUUUUGUAAGUUUUUAUUCCCUUCUGAGU
UGAGUAAUUGGGUGUUUGAGGUUACCCGACCCGUAUCUUUGCGAGUUAUGAUCCAGAUUU
CUGGUGGUUUUGCAGCCAUGGUGUUGUUGGAAUAAUUUUUUCCCUGAGCUUAACACAGAU
CUGAGCCUCAUUCCCUCUGUCAGUUCCUCAUUUCCUCUUUAUGGACCUAGAAGAAGCUUA
AAAGAAAUAAAGUCAAGGCUUAGACAGUAAAAAAUUUACUCCUCGACUUGAAAUCCUAGU
UCCUUCCCCAAAAUAGGUGAGAAAGAUUCUGGUUCUUGCGUAUUUUAUCAAUUUUCUCUU
AAAUGAACAUAUUAGUCAUAAGGGUAGAAGUUAGGUGGAGUGCCAAUGAGCUGUCAAAGG
CUUGUUAAGUCUAGUUUCAGCUGUUUUGAUAAUAAAAGCCUCAGAUUUUUCUUUUACAAU
AUGUUUUCUUUUCUUUCCUUUCCUUUCCUUUCCUUUGGUCUUCUGUUGAUUACUUGAUUA
CCAUCACAUAGUUCUUGUAAAGAAGGAAUCCCUUAAGGUUGAAACUUGUGGGAGUAUGAU
CAGGGUUCUAACUAGCCUAGUCAUUGUUCAUUGGUUGGUAAAAUCUAGAAGCUUCACAUG
GAUCUGUGUGAUUCCUGCAUGAUGUUAUGAUAAAAAGGGGAUCUUUGCUUAGUGUUAGUU
ACUUUAGUGCUUCAUAUAGCUCUAUGUCGCUGCUUGAUUUAAGUGUCGCAUAUGUUCUUU
GGUCCUGAGUAUUACCUUCAUUCUUCAAAUAUAUAAAGUUUGAAGUCGUGUUAGUUUGCU
AUUUGAGGUUCAUGUUAAUCUAGGCUCACUCACUCUCUAUUUCUGUUUGGAGAAGGAGUA
GAUAUCCAGGGUGAAAAUGCUUGUUUGCCUGUAUAAAGUUUGAAAUCUGUAGCAAUGGAA
UGCUUUAGAGAAUUUUGCUAGUCAAUCACCAUUUUUUGGUUAAAAGAUGCUUUCUCUUGA
UGUUUGAAAUCAGUACAUUUUCUGUUUUGAAAAGGAUUGUCAUAACUAUACUGUUUCUUA
UUUAAAAAAUGGGCAUGAGGGUUGAGAAUGAAUUGAGAUCAUUAUAUGUCGAAAAAUUGA
UUUAGUAUCUCCUUUAAGUUCCAUGUUGCUGUUUACUGUUGGUUGUGUGUUAAAAUUAGG
AUCUUUCAGGUAGAAUUUUGUGAUCCGUAUAUUGUCUGCAUGCCUAUUGUUGGAGAGUAU
GUUUGAAUUUACUUAAUUGGGAUUUACUGAUCUACAUGUAGUCUACAGGCUGGCUGCCUU
ACCUUAUUUCACAUGCUUUUUUUUAUAACCAAGACUGAUCUUUAUAAGCUAGAAUGGUAU
CUGGAACUUCAUUGCAUGAUUAAUCUUAACAUAAACAUGUCAAAGGACUUGUUUGAGCUU
UAAAUCUGACUAGAUGUCACUUUGGUGAUCUGUUUACUGUUAAGUGCAUAAGACGUAUGU
UUUGAGUGAAGUAGAACUAGUUUGUUCUUGAACCUGAGAAUCACAUAAUAAUAAGGUUAA
CUGCCUAAAAUCUUGUCAUUGUGUGGGUAAACUGAGGUUUUUUAUAGUUUGCAAGGGGAU
GGUCAUCAUGUGUUAGUUUUGGAUUUCUUGGAUUCAUAUAGUCAUUAUUAAGGGAUCCAG
AAUCAGUGUAUGACAUGUUGAAGUGCUGGACUGUCUCACUUGUUUUCUUUAUCCUAAAAU
UGAUCUUUCUACUGUCCAUUCAAUAAAAGGCCUUACUUUUUACCUGUGACAGGAGCAUUG
UCCAAAGGUUAUUUGGUUUAAGAGUUGUUUGUUUUGUGCUUCUAUACCUAUUAACGUGAG
UGUACUAGUGUUUUGUUAAAUAAGUUGACCCUUAAUUUCCAGACUAGCCUGAAGAAAUCC
AUUGAAGCUAAGUUAACUAAUUGGUAGGCUAUUACAAGAUUCUGGGUGGGGAUUGCUGAA
UGUUGAUUGACAAUAGCAUCCAUGGUUUAAGUUGAAACUAAACUUAUUAAUUUACAUCUG
UUGUGUGAAGAAUAUGAAAGUUGUAAAUUCGAUUUUUUGGGGGUUGUCUAUAGCAUGUUU
AGCUGAAUAUCUUCAUGAUCCAUUCACUUCUCAAUUUUCCAACAUAUAAUGGUGGAAGGG
AGGUACAAUUUGAUCUUGCAUGAAGUGGUUUAUUCAGGGAAGGACUGCAUUGACGUGAUG
GUUUCAUCUUAAAUUGAAAUAUGUAGGGGCUAAUUGAGUUCAUUCCUUGUAUUCCUUUGU
UGGUCGAAACUUGUGUAUAUAGAUGAAAACUUUUUUGGAUGCGUAAGACUUGUUUACCUU
UAGUUCUUUAAUAACCACCUUAAGCAUGCAUGACUUUGUUUAUGCUAAAUGUGUCUAGCU
GCCUUGAUCCUCAUUGAUGUGAUCGAAUUUUAUUGGCACUUGAUCUGUUUGGUGUUCAUU
UUAUAUUAAAUCGAAUAAUGACUUUCUGGCAUGUUAGAUGCUAAUCUUUCUUUCUCUUGU
UUCUCUUGUUUCUGCAUGAACCUCUCCUUACCGAGUCUAUUGGACUCAGCUUCCAUUCAU
UUCGAGUUAAAUACAUUUCCCAGCUCCUUUGGACCAAAGGAAGUGAAGAAAAUGAAGCUA
AUAUAUAGGGAAGGCCAUGAAGCGGAGCAAAAGGAUUACGGGCUUAACCAAAAAAAUCUG
GGUUGUCCAAGUUGGACAUUAGUAG
2. MFE structure-
((((((.(((((...........((.((((((..((.((.....)).))..)))))).))....(((((......(((((((.(((((.......))))).)))))))..)))))..((((.(((((.((((.....(((..((((((.....)))))).)))............(((((((((.....(((((....((
((......)))).(((((((((.(((.............))).)))).)))))(((((((((..(((((((((((....((((((.(((.(((.........(((((....))))).((((((((.((((((((((.((((....))))...))))))))))......(((((((........))))))).(((((((..
...)))))))..))))))))..(((.(((((...(((.((((((.((((...))))..((((((((((((.((.....((((((..(((((((......))))))).....((((((..((((.((((((....)))))))))).))))))....))))))....)).))))))))).))).((((((.(((((....))
))).))))))..))))))))))))))...)))...(((((((...((((((((((((((((................((((((.....)))))))))).)))))).)))))).....)))))))..))).))).))))))......))))))....)))))..)))))))))..)))))...)))).)))))........
..((((((.....))))))...................(((..((((((((((((((.........)))))))).))))))..)))(((..(((((.(((((.....(((((((((((((((........)))))))))))))))))))))))))..))))))).)))))))))...)))))))))))((.((((....)
))).))...(((((((((((.((((((((....))))...((((((((((((.......))))...)))..))))).....(((((((...(((...(((............(((.((((((((.(((.((((.((((.....((....(((((.(((.......((((((...))))))))).)))))....))..)))
).))))(((......((((((..(((....(((((.((((..........((((......))))(((.(((..((..(((((((.(((((..((((.(((((((((((((((......)))(((.(((((.(((((((((((((((.(((((((..((((((((((........................(((((((.((
.......((......((((..((.((.....))..))..))))......)).......)).)))))))((((.((((.......)))).))))....((((((((.....(((((((...............))))))).(((((.((.(((...(((((((.((((((((.....((((...((..(((((..(((.((
....)).)))..)))))...))....)))).)))))))).))))))).))).)).)))))................))))))))...((((..((...(((.((((((((.......))))))))..)))...)).))))))))))))))..)))).))).))))((((((..((((((........))))))..)))).
))((((((((((.......(((((..(((((..(((((((........((..(((((((...((((((........(((((((.....(((((((((((....((((.((.......(((((.((....((((.(((.....))).))))....)).)))))..........)).))))..)))))))))))...(((((
(((((...((((.((((...(((((((((.((.........((((((..((((((((((((.....((((.(((((.((.(((.(((((.(((.((..(((((...((((......)))).)))))..)).)))..)))))))).)).)).))).)))).....))....))))))..))))..)))))))).)))))))
))....((((.....))))...)))).))))........((((((.(((((((..(((((((((((((((.((((((....(((((((((((((((........(((((((((((((.((((((((((..(((.((........)).))).....))).)))...)))).)))))).)))))))))).((((((....))
))))))))))))(((((......))))).((((((.((..(((((((((.((((((((..........)))))))).))))))))).....)).))))))..((.((...((((((((((((((..((((((((((((((((((((((((((((.....)))).....))))))))))))))).......))))))....
..)))...))))))))).)))))...)).))................))))....))))))..(((((((.....)))))))((((...))))))).))))..)))))))))))))))))))))(((((((......(((..(((((.(((....))).)))))..))).......))))))).))))))))))......
.)))))))))))))...)))))))..))......))))..))).)))))....))))))))))))))).((((.((((..((((....)))).))))))))........))))))))))).))))).))).(((((.((.((((((.....)))))).)).)))))((((((((...((((((((((...((((((((((
(((((((((...(((((........(((((((....(((((.(((...))).))))))))))))............(((.(((.((((((.(((....(((((((((((((.....((.((((((((((.........(((.((...(((((((.((((.((.....)).))))....)))))))...)).)))......
))))).))))).))...)))))(((((.....)))))....))))))))))))))))).))).)))..)))))...)))))).)))))))))).)))...))...)))))))).......)))))))).)))))))))))).))))(((((((((.......))))))))).(((........)))((((((.....)))
)))...)))))..)))))))..))..)))))))))).)))))))).))))))((((((...))))))((((((..(((((((((((((.............)))))(((((...)))))))))))))......))))))........))).))))))))))).))))))...)))....)))))))))))...)))))))
))))(((((...)))))........
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-977.14] kcal/mol.
2. The frequency of mfe structure in ensemble 4.86975e-48.
3. The ensemble diversity 896.74.
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
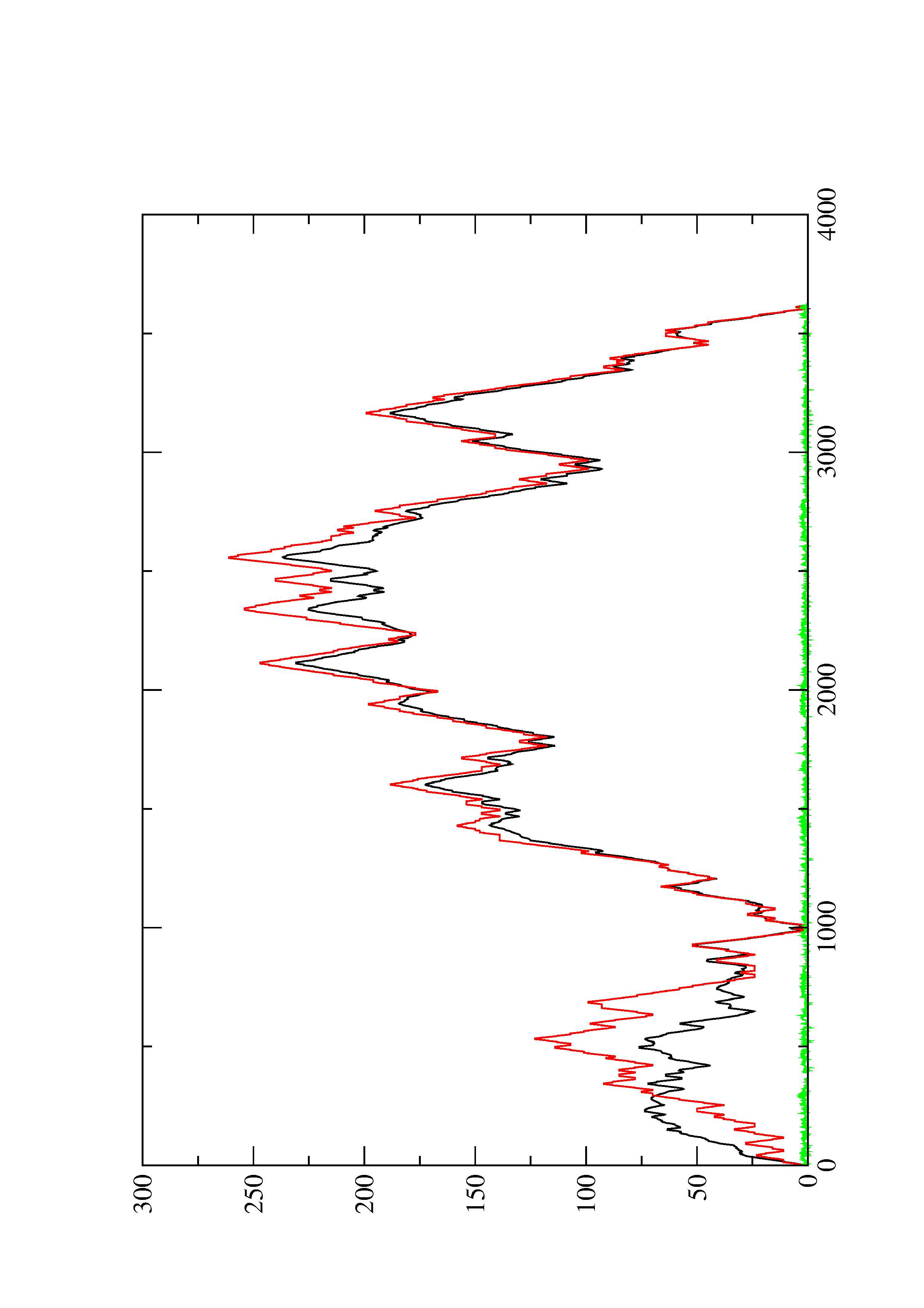
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.