lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01027354.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-598.64) is given below.
1. Sequence-
UUUAUUUUCAGCCCCAAUUCUCUGGAUCCUCAAAUCAUUUGCCUUCUUUUCCCUUUUCUC
CCAAAAUCGUGAACCCCGAUUUUUUUCUUUGAUUUCUGCUUGAUUCUUCUCAAACUUUUG
AAAAUGUUCUGGUGAUGUCACCAUGAUAUCCUUGUGCGUGUACAUGGUUGCACCUCCUCC
CUUCGUAUUAGGAACGUUGUUCCUUCGUCCUUUUUCCCGCUUUAAGAAUUUUCAGUUGUG
GCUGAUGUAUUCUGUUUUGGUGCUCUUUUGUACGAUUUGGUACGUCUCGGAGGCAACGAU
UGACUUAUGAUUAUCCUCCACCCCUUUCCCUCAUUCGAAUUUUGGCAUGCAAAUCCCUUA
AAGGACUGAGGCCUUACCAAAUUUGAAUGAAGGGCUGGAAUUCAUUUUUGCUAGGUUACU
UUUGUAUUUUUCUCUAUUUUGUGACUUAUUGAUCAAGUUGGCGUUCCCUAUUGAAAACCA
UGCCCUUUAGGGUUUUUAAGGGCUUGAGAAGGAGGCCAACAGUGAUUUUUAGGGUUUUAA
UAACAUUUUAUAAUUUAUAUUUUGUGCAAUUUAUGUUAAGGGUGCUAGUAUAAUCACCUC
UAAAAACAAAAUAGGUUGUUUAUAUACAAUUGUUUACCACUUGAAUAGGCCGGAAUCUUA
CUAUUUUUGAGUUGUUCUUAGUCUAAAUCAGUGUUAAAAAGAGGUAAAAUCAGAGUGAGA
CGUUAUCCUUUGGGUGUUACAUUUAAAGGAUCUCUCCUUGCACCCCUCUAAGGGAUCAGU
AGGCUGUGGAGAAAUUGAGCGAGUAUUUUAGCUUGUUUUUAAGUCUUUUCAGGUUGUUAC
CUUGGUUCGUUUUGGUUCCUGCACUUGAAAAGGUAAUAUGUUUCCCCUAUUUAAUAUCUU
UAUGUCAUAUACGUAUUGAUCUAGGUCGAUGUAUGUUUACAUAGGCUGUUUAUGUUCUCA
UUUGUGUGAUUCUUGAAUCUAUUUGUGAUGUGAGCUUAAUAUGAGUACAUUCUUACCUUA
UGUUGCAUGUUUACUGUUGACUGCUUAUUUGAUGAGAUUAUGCUAGUUUUUUGUUCUUUA
GACUAUUUGAUUGGCUAAUAAUAGCUCAAUUUGAUCAUGCAUAGCCUUUCUGUUGCUUAU
UAGCUUGGUAAGGGUCAGCAAUAGUCUUAACAAAUUUGUUGUGAAGCUAGCAAUAUUAUU
GUCCUUAUUCUAUGAGAUAAAUUACCAUAUGAGCUUUAUAUUUUGAUCUUGCUAUAAGUU
GAUUAGUAAAUCCCUUCUUCAAAAGUUGUCAUCAUGUCUUUUAGGAAUUAUGCAGUAAAC
CUAUUUGUAUUUCUCUUAAUGUGCCAGGCUUAAUUAAAUGUGACAGCUGAGUAAAAGAAU
GUGAGCAUGUACCAUGUAAUUUGAUAAAUCCACUUGUUGUUUUCCUUUUUUAUUUUUAUU
UUUAUCUAAUCCGUGGUUGUCUUGGAUUUUCUUUAUCUAACUGUCUACCUGCUAUGUACU
GAAUAUGUUCUUCAUAAUCUGUCAUCCUUCUAGUUUUAUUUGUUGGUCAUUUAACUGUCA
AAUAAGAGCUUCAUAUGAUCAUAUAUAGCCCUCAUGCUAGAUUAUAGAGGUUAAUACUGG
UCUUAAUCUACCAGUUUUCUUCUAGUUGGUUGAGUCUUAGUGCUACUCUUAUAUGAUUCA
ACUAUUCUACUAAAAUAUAGGUUCUAUGCUUGGAAUGAUAGCGUAACAUGCUCGGUAAGA
UUUUGAAUUGUAUUAUGGCUAGCUGUCUUUCUCUCUUAAGUUGUAUUCUCUUUUAGCUCU
GUUGUAUAUUCAUCACUAUUGGAUAAUUGAGUAAAAAUUGAUAGAGCUUUUCUCUCUGAU
CACUAAUGAAACCCUUACAUAGACAUAAUAAGACCAAUUACUCAAAACUAGAUAAUACUA
GGCCUGUCCAUUAUAUGAAUCACCUGGACUAUUUUUUUAUUGUAGAUACACUCUCUAAAU
GGCUCUGUUUUGAGUACAUUGUUUGGAAUAAGCCGUCUAUGUAUUUAUCCUUCCUAAUAA
CCAAUUAUUUCCCAAAAAAAAGUUUGCAUAUCAUAUGUGAUUAUCCUGUUGCCUGUUAAA
GUUGCUAAAGAGCCUUUAGUAGUCUUAUACAUGUAUAUCCAUGUAUGUAAUUGAUAAGUU
CAAAUAUUUUUCGUGUGAAAUUGGGUCGGUCCUUUCCUAUGUAGGUCGACUGGCCCAUAA
UAUAUCCAAUUAACCAGGCCCUUUUAAAGUAUCGUUAGCCCGUUUCUUUUUAUGUGCCUU
GCUGGGCCAUUUGUUUACUAUGUUUUCUUUUAUCUCUGUUCUUGGGGCCUGAACCAAUAU
AAAUCUUAUGGAUACAUUACCUUACUUACUGCUCUAUGUUAUCUUUUAAGAUUUAUAUUA
UUGAUUUUUAAUCUUUUUCUUUCUUUGCAUGUGAACCGUAGGGGCUUGGGAUCCCUUUUG
CAUGACUUUGAUGGCCAAAAGCCCACCCCUUUGAAGCUUUGGAUUGGUCCAGUCAAGGCC
AUUAUAUCCUUCCUUAUUUUAUUUAUACAUUUAUUUAUAUUUGUAUUUGUAUGAAAUCUA
GUUGGGGGGACCAAGGGUCUAAAGUCUCUGAAUUUUAAUAACAAAAAACAAUUUUUUUUU
GUUCAUAUUUUCGUGCUAUCUUUU
2. MFE structure-
......(((((((((((......(((((((.((((((((.(((((((((((...........(((((((.......))))))).......((..((.....)).))...((((....))))..(((((.(((((....))))).)))))....(((((..........)))))..(((((...((((((((((((...))
))).((((.........((((...(((((..(((((....)))))...))))).....)))).........))))..)))))))....))))).((((.((((((..((((((...((((.((((.....((((((((((((.((...((...(((......))).....))....)))))))))))))))))).)))).
...........((((((((....(((........)))...)))))))).))))))))))))))))((((..((((((((...........))))))))))))...)))))))))))....))))))))..)))))))............(((((((((((.....((((((((((((.((((((..........)))).)
)...))))...))))))))..))))).))))))(((.((((.((.(((((.(((((((((.......))))..)))))..)))))..)))))).)))..((((((((((...(((((((((((.(((((((((((((........(((((....)))))))))))....))))))).(((((((((.(((((....((((
((((......)))))))).....))))).)))..(((((((((..((((.....((....))...))))))))))))).......))))))....(((..(((((.....)))))..)))...((((.(((((....))))).))))....))).))))))))((((((((((((((......(((((((..((((((((
(((((((......)).))))))))....((((((((((((((((..(((((.(((((((((((((((((..........)))))(((.(((((((((....)))).))))).)))........(((((((((..(((....))).))).)))))).))).))))))))).)))))..))(((((((((..........((
(((.((((...)))))))))...........)))))))))................)))))).))))))))...........)))))..)))))))...))))))))))))))(((((((......((..(((...(((((.((.(((((((.......(((((((.((.((((((((((.((((((...((((((..((
(.((((((................................)))))).))).)))))))).)))).))))).))))).)).))))).)).....(((.((.((.((((.....)))).)).)))))...(((((((((....(((((((.((....((((...((((...(((((.((((((....((((......)))).
....((((((.....((((((........))))))...))))))..((((((((((..................)))))))))).........(((((((.((........((((.((((((....((((..((((..........))))...))))....)))))).)))).....)).))))))).......((((((
((((..((((((...............)))))).....))))))))))........))))))...)))))...))))....))))....))))))))).....)))))))))......))))))))).)))))...)))..)).(((..((((........(((((((((.....((.((((((.(((((((((......
.))))))))))))))).))))))))))).......)))).)))....................(((((((...)))))))..........((((((((((((((((((((...))))))))).)))(((((((......)))))))(((.((((.....)))).))).((((....)))).((((((((((....(((..
..)))..)))))))))).............))).)))))......((((...((......))...))))..(((.((((....)))))))..)))))))........))))))))))....)))))).)))))..(((((((((((((.((((((((..................))).)))))..))))))))))))).
..........................(((((.(((...(((((((((.(((.(((((....(((.(((((((((((((((((.((......))..))))....)))).))).)))))).)))....(((((((........((((((((..(((........)))..))))))))........)))))))..))))))))
.))))))))).........(((((((((......)))))))))....))).)))))........
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-650.73] kcal/mol.
2. The frequency of mfe structure in ensemble 1.98137e-37.
3. The ensemble diversity 747.96.
4. You may look at the dot plot containing the base pair probabilities [below]

III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
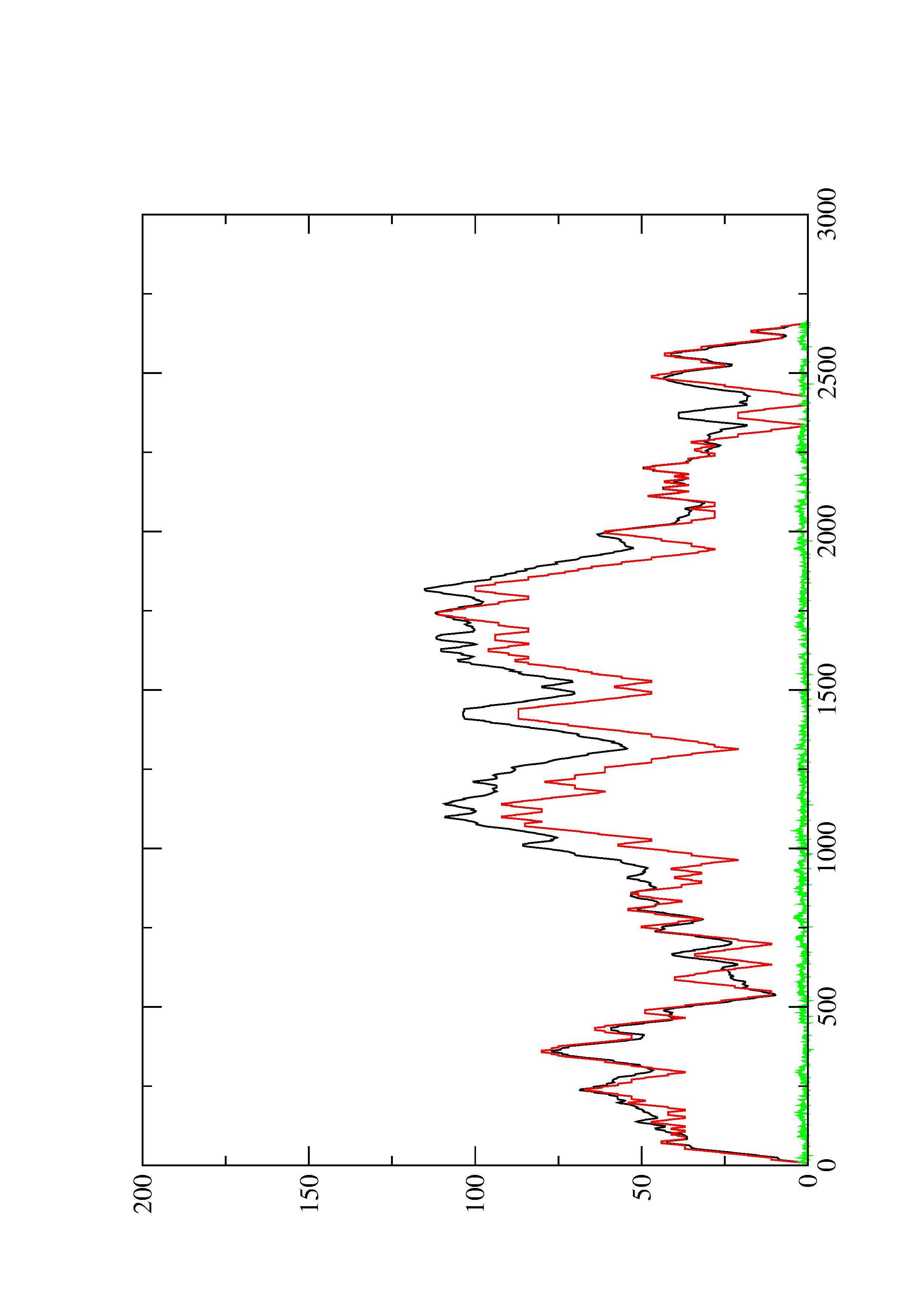
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.