lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01030667.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-827.40) is given below.
1. Sequence-
AUGGUAGGUUGGAUUUAGUUGUUAGCGUAGCCUGUUUUGAUUUGGCUUUAACUCUAAUCU
AUAAAGGUCUUAAAAGGUGGUUUUGAUAUGGUGUUUUUGGAUCAACUCAUGUUGCUAGGC
UGAUACUGUUUCCUAGUGAAAUUUUAAGAAAAGGCUGUUUUCUGAUUGUGUUUACUGGAC
CUGGCUAUGCUCCUAGUUCUAGUAUUAGUAUUCUAUUCAUUGGACAAGUGGAUUUGCAUA
UUGAUUUGGUAUACUUGAACCUGCUAGUUAAUAAUGUUCUUUAUGUUAAUUAUGGUUUAA
AUAUGGUGUCUUAUGGCUCUGGUAAUCUGUCCUGUUCAUGAUCUUAUUUUAGUAGUCUUC
AACAUCUCUAAAACAAGUUAAGAUUUGUGUCUGUUGGUUAUAUAUGUUUCUGCGAAAUUC
UAUGGUAUAUGUGUUAACUGUGUCUAUGUUCUUCUUUUCUUGAAACUGGUUUUCAUUCAU
AUCCUAGCUGUAAAAAUCUUUGUUUUACAAAACCUGUUUCAAAAACAAGGGCUAAGAUAU
CUAAGGUUGGCUAUGUGAUGAGUAGGUUAAGGGAGGCUAUCCUGAUUCCAUUUGAAUCCC
UGGUUUUCAAGGUGAUCUUAAAGUAUCAUAAUAAAGACCUCCUCAGCCCUCUGAUUUUGG
AAACUGUCCUUAUGUGAUCCUUUAUCUGCUAAGUGUGUUCUUAGAGUCAUUUUCAGUUCU
AAAUCGAAGUGAAAUGGUUAAUCCUUAUUCACUUCAUCCCUCCUUUACCUAUAAUGCACA
AUGACCUAUGUGAGUGCAAGGCAAGGAGAGGACUUGUGAGAUGGAACUUAGGAAUUUGAU
AUUUUGCCUAUUCUAAGAACCUAUAUCUUUCAAGGAUGCCUAAGGUGAGGGAUGUGGGAU
GAUAUGGGGUUCAGUCCACCUCCAAUGAGUCCAAGGGUGUGAUGAUCUAAAAGGUUGAGA
UAAAGAGGACAUUUUGUUUUAAAUGACUUGAUAGGUCAAAAUUCUCUUAUUUAGAGUCUU
AGAUCCAUUUUCUUUGCUUACAUGAUUCUGAAUAAUGUCUCUUAUAAGGUUGUAAUAUGU
UCUGUGAUUAGCUGAGUGGCCCAAUUUUAUGUCAAUUCUGCAUAUUCUUAACUUGAAACU
GAAAUUGGAAACUGAGUAGUUACUAUACAUUCUUCUAAGGUUUGCUAGACUAUAUUCACU
AACUCCAUGUUAGGUUUUAUGCUAUUUUAGAAUCCUGAGGUGUCAUGCAUGAUUUGAUGU
GUUACGGCUACAUGUCUACAUUUAUUCAUUAUGCAUGUUACAUGCUUAGUUAGUUAAGUU
AACUAAGUGUGAAUCUAAUGGUUUCUUGCCUAUAUGAUCACCAAUAUAAUUUCUUUAGAU
AAUAUAAUGGAUUGAUUGCAUAAAUUGAAGUUUAAAACUGAAAAAACAACUUUGUAUUAU
GAAGAUCAUGGCCUGUUUCAUUCUCAGUCAUGUUUAUAUUGCUAACUGCCUGAUCAUGGC
CAGUUAUUAGUCUUAAAAAUGCUUUUUAUAUAUUCAUUUUGUUAGUUGUCGAAUGAGAGU
UCAGUGUGAUCCAUAACCCCUUGCUAGCUGUUAGUUUGAUAGUAGUCUUAGCUUGUCUAC
UUGUGUGCAAGUAUGGAAGGUCUAGUAUCUUUGUCAUGAGGAAAGAUGGCUAUGCUUUGU
CUCAAUAUGUUCUAUGCUUUCAAUUGAUAUAAUGCUAUUGAUUUUUGCCUUAUCAAAACU
GUACAACCUGUCUGCUUUCCUCUUGAUUUCCUUACAUGUAUGACAACUUGCCUUCUAUCC
AGCCUUUGUCUCUUUUGAAUGACUCUUCAUGCUAGUUUUAUGCAAAUAUAAUAGCUGGCU
UAGAGCUUGACUUCUAUCAAGCCCCAGUUUCCUUUUAUAAAUACAUGAUGUUUUAUUAGG
CCAGUUCUAUUUACUGUUUUUAUUUUUAGUAUGACUGCCCUGGGUUGCCCUUAUGUGACC
ACUUAUCUGCAAUAGACUUGCUCUGCAUGACCUAUCAAAUCCCUGCAUAUAGUUGUUCCU
CUUUUGAUCCAUUAGGUUCAUAAUUUGUACACAAACAUAUGACUUCUUUCUCUCUAUCUU
GCCUGCUCUCAACCUCAAUACUUCUUUAGGCCAGUGUUGAAAUAUUAAGUGUUCACCUGC
AGUUGCUGUCUUAUUUGUGAUUGCCUGAUUAAUCUCUAACCUCACUAUGUGCUGGAACUU
GUUCUAAAUUCCUGGUGAGUUCCUAUAUGGCAACCUACUCUUGUUUUGUUUCAUUAGAUC
GGCACCUUGUAUCAUAGUAAGCCGUGCAUGCUAGUGUAUUCCAACCGAUGACCAUGUUGC
UGCCUGCUCAACCCUCUCUCUUAUGUGUGAGUCUAUGGUUACAUGAUAUCAACUUACUAU
CUAAAUAUCUUAUUUAAGUCAAUGUUGGUCCUUGUAGACUGCUUCCAGUUUACCAUUUCU
GUUCAUUUUGGGUUGUCUGAAACAUGGUAUCUAUCUUCCUUUAUAUUAAGUCCAUGAUGU
AUUGUAUGAAUCAGCCUCAUCUUUCUGUUAGGUAUUAGGCUCAUGUCCCCUCGUAUGUAC
CUGCUUCGUGUCUCCCUCUUCUUAGUCUGAGUUUAUAUUGAAAUCUGACGUCUCCUUCCC
UUUCCCUUUGUGUUAAGCUUGUACUGUCACAUGUUAUCCCUUUCCCCUUUCCCCUUCCCC
UUGUGAGUUGAGUUCAGGUUAUAAUUAUGAGCCUGUUGUUUGGCUCUCAUACUAAGAAGU
UGAUAUCAGUAAGCAUGCCAUCUGCUUUCUCCUUCAGCUUGCUGUGGAUAUGCUCCUGCU
UUUUCCUUUUGUUGGUUGAAUUGAUUCUCUUAUGCAUCUCCUUCUCUUUUUCUUGGUACU
GAGUUCAUGUCUAACUGGCUAUCUGUUUCUCCUUUCCCUUCUAUUGAACUUAUGUGGUCU
UGUAUACAACUUCUUGUCUGUUUAGUUAGUUGGGACUGUGGCUUUUAAACCCAAACAGUC
CCUUACCUUUGACUUGAUUGUUUAGCAUUUUCUAUUUGUUCCUUUCUAUUUUUGUUUUCU
UCUUAUUUAUAUUUCUUAAAUAGAAUGUCAUUAGGCUAAUUGCUAGGCUGAGUGAGCAUC
AAGUGUUUAUACUUUGUUAUCUUCAAAUUUCAUGAUGUCUCUAUACUGUGUGUAUCACCU
UAAUUAUAAAAUGAGUGGUAGCGAGCGUCUCAUGGUUAACUAACCAUGUUAAGUCUUUUU
UAAUCUGUUUCUUUGUCUUGCAAGUGGGUCUGACUACAAGUCGACUAGCCCAUCUUGUUU
AAGAUAAAUUUAAUUGGGCCUAUAUUUCAAUUUCCUUUAGUCGUUUUUUCUUUCUAAAUA
UGCGCCUUUGGGCCUGGCCCCAAAUUUGAUUUUGCUGCACUUCGGCUUACUUGCUUAUUU
AUUUGUAUUAUGUUAUAUUAUCGAUGUCUAAUUCUUCUCUUUUUUCACGCGUAACCUUGG
GUCCAGGAUUCCUCUGCAUGGCAAAGUGGCCUAGGCCCAACAAAAUUGAAGAUUGGACUC
GGUCCAAAUAAGUUUCAUUUCCUUUCC
2. MFE structure-
...((((((((((.......(((((...((((...........)))))))))))))))))))...((.(((((((((.((....(((((.(((((((.(((.(((((((.(((((((((((((((((((...((((((((......((((((.....)))))).........(((((((.((((........)))).)))
)))).(((....))))))))))).(((((.(((......))).)))))......(((((((...(((((((((..........))))))))).)))))))..))))))))....))).)))))))).............(((((((((((((.((..........)))))))).....)))))))...(((.(((((..(
(((((.....((((...((((((((((((.(((((....((((((..((((((.((((..((((((.(((((................(((((((.......)))))))))))).)))))))))).))))))..))))))...((((((.(((((((..((((((((...((((.....))))(((((.....)))))..
.((((((....(((((((...)).)))))....))))))((.((((((((..(((((((((.((((.((((((((.((((.......((.....))(((((((((((((..((((((((((..((((((((((..((.....))...)))))))).(((((((((..(((....(((((..............)))))))
).)))))).))).....))..)))))))...)))..))))...........))))))))).((((((((((((((...)))....))))))))))))))).))))))))..)))).....))))..)))))..))))).))).)).....(((((.((((.(((((((.(((((......)))))..(((((((((((..
......((((((((((((.......((.......)).......))))))))))))..........(((((((....((((...(((((((.((.(((...(((((((((.((...(((................))))))))))))))..))).)))))))))...)))).......(((...((((((((((..(((((
(((.....((.(((..(((((.((((((((....)))))))).((((((((((..((((((....(((.....))))))))).....))))))))))...(((((((((((((.....)))))))))))))........(((.....))).....((((((........((((....((((((.(((((((.(((.((.(
((((......(((((.(((((............(((.((((((...((((((.((.........)))))))))))))).)))((((((((.......)).))))))))))).))))).......))))).)).))).)))))))))))))....)))).......)))))))))))..))).)).....))))))..)).
.)))))).))))...)))(((((....)))))(((((((((..(((.....((((((((((((((..(((...((.((((.(((((.((...........))))))).)))).))..(((((.........))))).............)))..)))))))).................))))))))).))))))))).)
)))))).(((..........)))(((....((((((((((((....))))).)))))))..)))((((((......))))))..............(((((.(((...))))))))((((.(((((.((((....((....))....)))).))))).))))((((((......))))))........((((.....)))
)......))))))))))).......(((((..((((........(((.((....)).)))..........))))..))))).))))))).)))).)))))))))))))......((((((((..........))))))))............(((...((((((((..........)))))))).)))............
(((((((.....(((((....)))))......)))))))....))))))))))))).((..((........))..))...)))))..)))))))))).))....)))).....)))))).))))).)))...(((((.((((...(((((((............)).)))))...)))).)))))..(((((((((((..
.((((((...))))))))).)).))))))....((((((((....))))))))...............))))))))))))))))).)))))...)).))))....))))).))..(((((((.....((((((((((.((((.......))))...)))))...........((((((...((((..(((((.....(((
((((((.(((......((((.(((((((..((.............(((......))).........(((((..............................)))))...))..))))))).))))...(((((........)))))))).)))))))))..))))).)))).))))))..............(((((((.
((...(((...((....))....)))....)).)))))))..)))))....)))))))..............((((.((((((((((.((((....((((((.((.(((................)))..)).))))))..(((((.(((..((((..((((((.((((((((((((((((........))...))))))
))........(((......)))(((((.((((((..((.....((((((((..........................))))))))...)).))))..)).))))))))))).)))))).)))).)))))))).((((((((((((..(((.(((((......((((.....)))).......))))).))))))).))))
))))((....(((((((....)))))))....)).................((((((((((((((((((.(((((...)))))....))))).)))))..)))))))).......((((.(((((((............................))))))).))))((((((((((((.........(((((((((((.
...(((......)))........((((......)))).....((........))............((.((......)).))..(((...))).)))..)))))))))))).)))))))).........))))))))))))))))))....................
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-893.96] kcal/mol.
2. The frequency of mfe structure in ensemble 1.25323e-47.
3. The ensemble diversity 771.12.
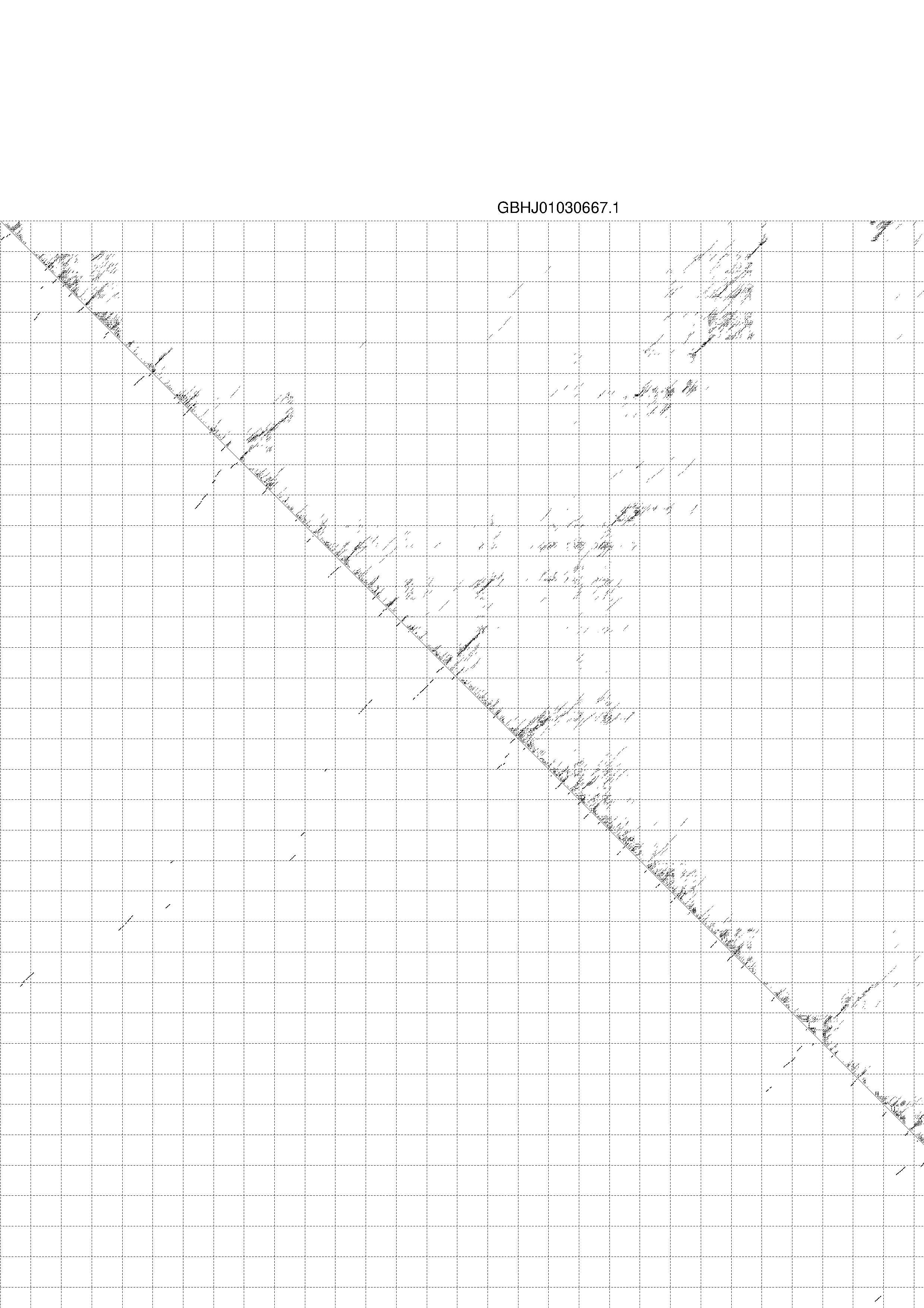
4. You may look at the dot plot containing the base pair probabilities [below]
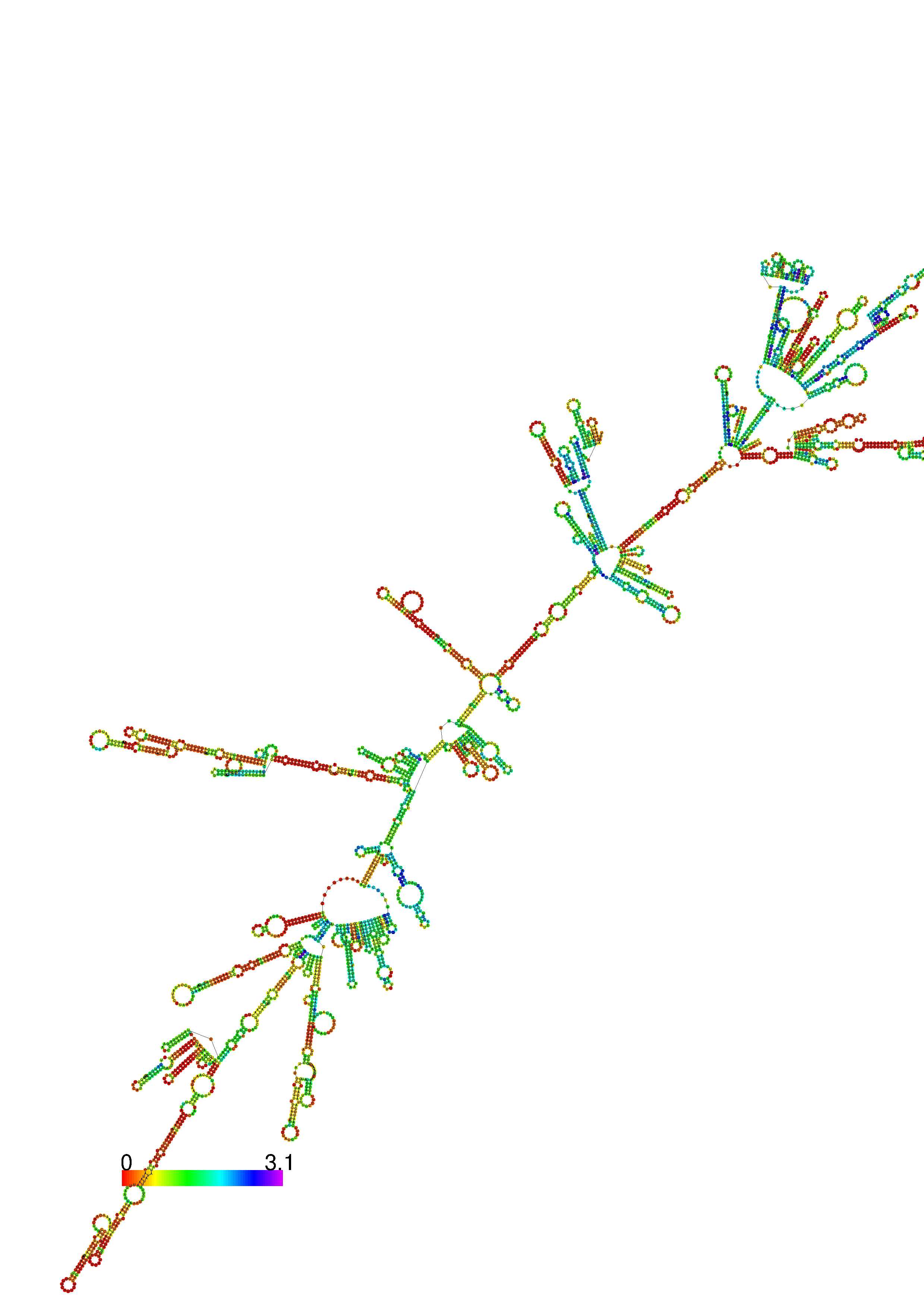
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
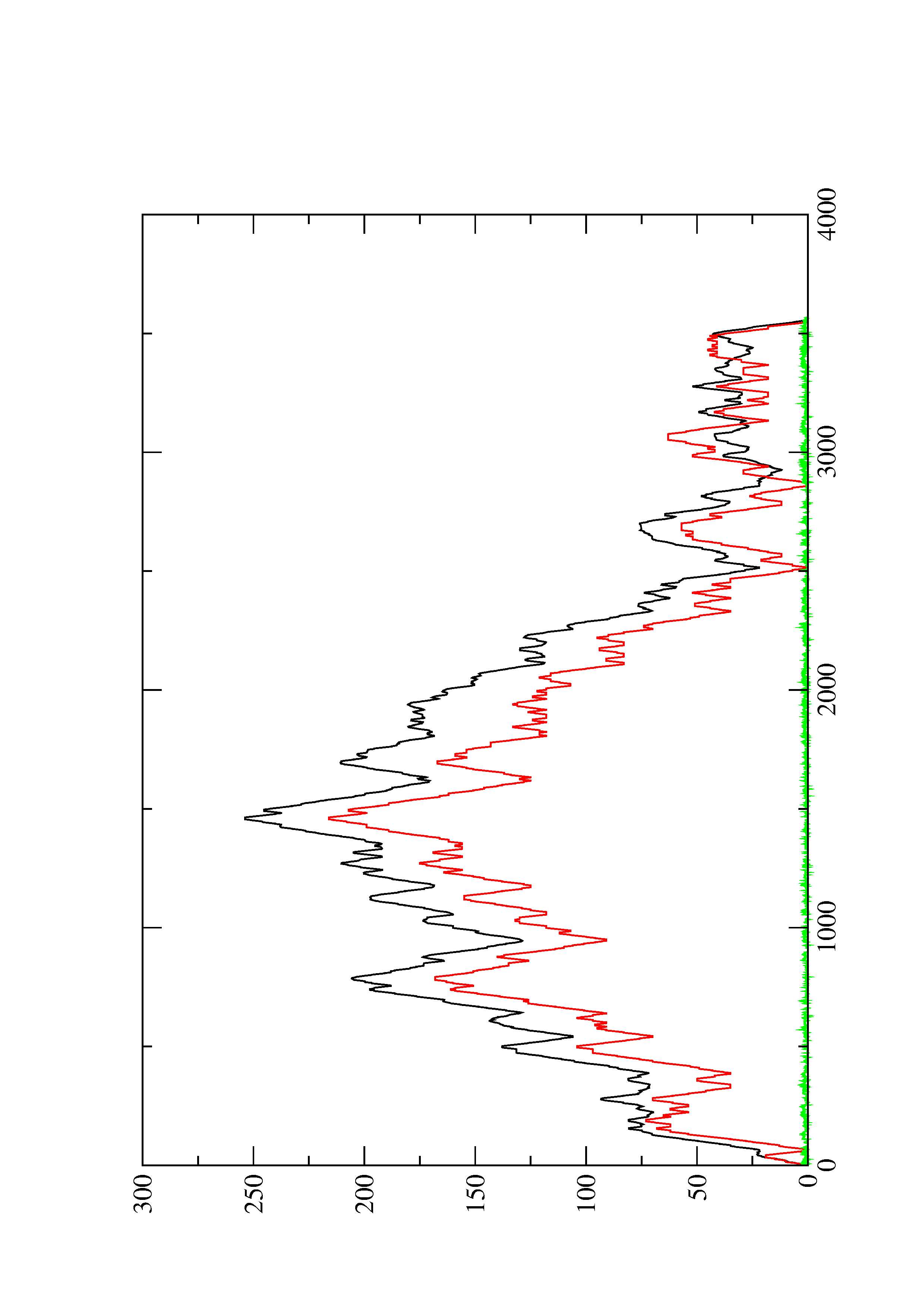
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.