lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01031507.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-555.80) is given below.
1. Sequence-
UCUCAUUAAUAGAGGUUAAUAGGGUCUCUCUCGAGAUUCUAAAUGGGCUGAUACUAGUUA
UCCAGGCCUAGGAUACACAAUCUCAUUGGAGAAGUUGAAGCCAAAGCCUAGAGAACCCAG
AUUCUUGCUCUAAAAAAGGUGAGAAACACCUCAAACCUUUGUUUAUUAGUUGUUUUCUUU
GAGUAAUUUUGUUGUGAAUGUAAGUUUAAUGAGUCUGUGAGCAUAACAGCAUGAUAACCC
GUUAAACUAAAGUAUAAAAUGUGUUUUUGCCUUAUCUGAAGAGUUCUAUUUGAGUCUCUA
CAUGAAACCAACCUAAAUAAUGUUAUUUGUUGCUAGUUGAUGGCUUUAUGAUUUAAAUGU
GCAUUAAGUAAGAAAUCUGGCCUUUAUAGUAGAACCAAGGAGAGAACAACAUGUUUAGCC
UAGGGUACUUUCUUACUAGCUUCAUAAUAAGGAUUGGAUCUUUAGGGCCAAGUACUUACC
UUAUCUGGUUAUGAAGUGUUGUUCCUGUUAUACAAGAUACAACUUGCAUGUUAAGGUCCU
GUGAAAAGUCAUUUCAUGUUUGAAAGUUUUGAUUUUGAGCGUCACAGUUUGGUAAAGGGC
AUCUUGUGUAGAGGCUUAAUCUCGUGUUUGUGCAAAUUCAUGAUAAAAGUUAAGAUUUUU
CUUGUGUUUACUAGCUGCUUGGGCUUGAUUGUUGUAUGAUUGGGACGAGCUUUUUUGACU
GUUAAGGUCAAGGUAGGCCUCCAGUAUGUUUGUCUUAGGUAAAUCAAAUAGAGGCUUAUU
AGAUUUGAUUGCUUGUUUCUUUGUUUAUAUGUUUGGAACAUGAGUAUCUUGAUUACAAGG
UAGGAGCUUUACAGAAGGUUGUGCUUCUAUUAGGUUUAGGUUUAAGACUACAACUAUGAU
AGACCUUAUCUAUGUCUUAGUUGAAUCUUGUUAUUCGAAUUGUUGUAUCUUCUAUUGAGU
AUGUUCAGGCCACAUAUUUUCUGUGAUUCUCCAUAGUUGACUAUACUAUGGCAGGCUCAU
UAAAAACCUCUAGAAAGGAGUGGUAUCUUAAUUACCGUUUUCCUUUAGAGACCAUGGGUA
AAGGGAUUUUAACAAUAUCUCACUCUUACCCGAAGAUCUAUCCCUGUCAUUUCUGUUUGA
AUUUUUUUCUAUUUAGGAAGAUAAAAGUUUGGGUUUCAUGUAUGCUUAGCUAAAAAUGGG
GCUAGUUCAUGUAUAUUUUCUUAAGUAUUUAGGGAGUAAAAGCAGUGAAACAAUGAUUAU
CUUGAGGUAUAGCCUACUUUUUUUGGUCUCAAAGUUGAAGUCUGAGUUUUCUUUUGAUUU
AUCAAUCUAGCAUCAUGUUUAGGAUUGGAGAACCUUUACUCUGGUGAGAAUCUUGUAUAA
AGUCAGGUUCUUUAAUGUAUUAGCUUAAAUAAACAUCUUUGUUUGAUCUUCCUAGUUUGC
UGGUCAAGAGAUGUUUUAUUAAGCUAACUUCUUUUAAAAGAAAUGGGGUUCUCUGAUUCA
CGUAGGGUUUAUUUGGUCUCAAUUCUUGUCUGAGAUGUUUCAAUACACAUUAGUAGUACA
AUCUUGUUUGAAGUAAUUAAAUUAGUACCUUCUUUUUUAGGCCAUGUAUGAUUCAUAACU
UUCAGUCUAUGCUUGUUUUGUUUCAUUUUGUUUUUAAGUAGUCAAACUGAGUGUUUCUUG
UGAUUAAGGUUUGUACCUACUAUUUUAGAGUAGUAUGCAUAUUUAGGUCUUGAAAAGGUU
GGAUUUAGGGGAGGGUAUAUAUGCUAAUUGGAUCUGAGAAGUAAAUUUAGUGUUUCUCUA
AUGUUUUUUAUUGAAGUGUUUUUGAGAAUUUGAAUGGCUUGCAUCAUUGUCUGUAUAAUG
CUUCUUCUCUUCUCCUGUGGGUUCGAUCGGAACUCUUGUACAGUAUUCAUUUUAUCCUAG
GUUAGGAUCUAAGUAUGAGGCAUUUAUGCAUUGAGUUAAAUUGUGCUUUUCUUUAGUCAU
AUAUGUAAUAUGAGGUCUGCUUGUGGACCUCAAUCAUGCUGCUGACCUCAAUAACUCUCC
UAAUUUCUCCUAUAUUUAGAGGGUUGUUGGAGAGGUUGGGUUCUUUUAAGUUUAGGUAAA
GGCAAAGUAAGUACUGUCUUCUGUCAAAGCAAGAGAAAAAGAGAAGAAAAAAUGGUCUCC
UAAGGUCUAUGAAAUCUGUAUAGGAUAUAGUGUUGACUUUAAUGUGUGGCAUGAAUAAG
2. MFE structure-
.....((((((((......(((((((((....)))))))))..((((((((((.....))))..))))))((((((((((((((....))).((((.(((((.(((((((((((...(((((((((((((....(((((((((......)))..))))))....................))))))........(((((.
...)))))..)))))))..((((((((.(((.((..((...(((.((....)).)))...))..)).)))....((((.((((((((..((((((((((((((((..((.(((..(((((......)))))((((.....)))).....(((((.((((((((...(((((((((....(((((.............(((
...(((((...)))))..))))))))..))))))))).((((((((((..((((.((.....((((........))))))..)))).))))))))))(((((..((........))..))))).)))))))).)))))((((((((((((((((((....)))))....))))))).....))))))...)))...))..
...)))))))))))))))).(((((((((..(((...((((.((((.(((((((....(((..(((((....))).))..)))))))))).))))))))..(((((((((..((((((((.....))))))((((((((((.....((((((............))))))))))))))))......))..))))))))).
.........)))...)))))))))(((((((....))))))))))))))).))))..))))))))))).)))))))).)))))..)))).((((((.((((...........)))).))))))...............)))).)))))))))))))))...((((((.(((((((((.....((((.((((((((((...
....))))))).))).)))).............((((((((((((.......)))))..))))))).((((((((((((((.(((((.........)))))....)))))).(((((..(((..(((..((.((((((..(((((((..((((((((((((((........((((((((....((...)).....)))))
)))..........))))))))))))))...)))))))..)))))).)).)))..))).))))).((((...)))).((((((((..((((...((((((((((.((.......((((...(((((...(((.((((((...((((((((((((((((((......(((((((.((........)))))))))....)))(
((((((((..(((((((((...((((((.............)))))))))))))))..))))))))).(((((....)))))..))))))))))))))))))).)))))...))))).)))).......)).))))..))))))......((((((((((((((((.((.(((.(((....(((((.....(((((..((
((.....((((..((.(((...((.....))...))).))..))))...))))..))).))....)))))..))).)))..)).))))).))))).))))))......(((((((((((((((((((((((((...(((((((((((.(((((((((....))))..........((((((((.......))))))))..
............((((((((....(((...(((.((.....)))))...)))....)))))))).......))))).)))))))))))(((.....((.........)).....)))....))))))))))))))))..((((.((((.((((((..(((......)))..)))))))))).))))....((((((((..
....))))))))..)))))))))((((.((((..((((((.(((..((((((....))).)))..))))))))))))))))).......((((.((((((((((..........)))))).)))).))))..))))..)))))))).....)))))))).((((((((((...((((.....))))...)))..))))))
)))))))))).))))))..
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-599.25] kcal/mol.
2. The frequency of mfe structure in ensemble 2.40298e-31.
3. The ensemble diversity 735.47.
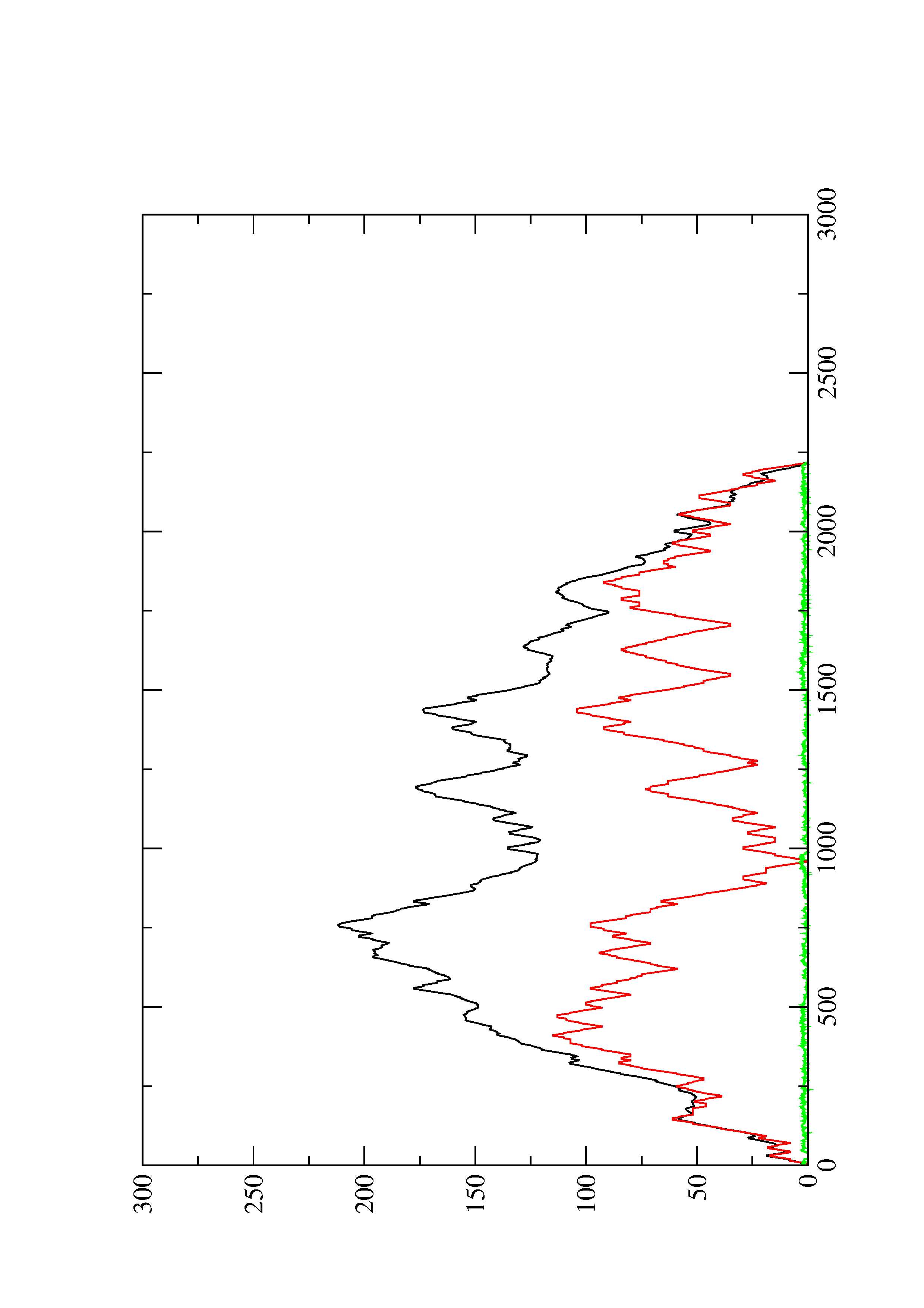
4. You may look at the dot plot containing the base pair probabilities [below]
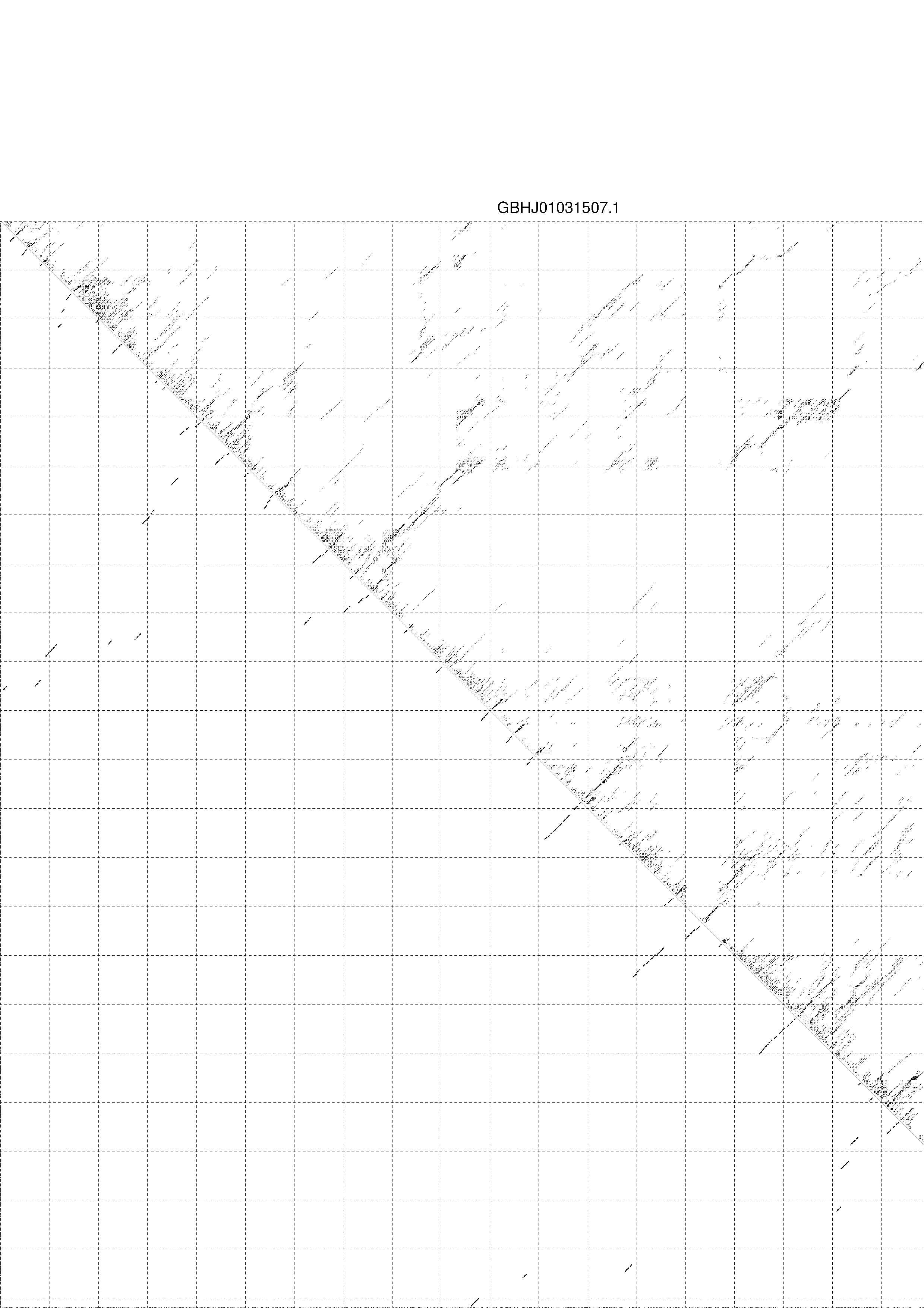
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.