lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01033107.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-703.20) is given below.
1. Sequence-
GUUUGUUGCCCCUUUAAAGGUAAAAAUCUUUUCAUUUAUUUAUUUAUUUCAAGCUGGUUU
UAGUUAACUAUGACUGCGCAAUGUUAUAGGACUGAUUUUUUGCUUGUUGGAUGUGAAGUA
GACUGUGUAUGUUCUUUGUUUGUGGAAAAAAAUAAGUUCUUCUUGAAUGGUGUUGGGCUU
UGAUGCUUUUAUUUGCUUUUUUUUUUUUCUGCCAAAAACAUGAAAUAGAAAAUGUAUGUU
UAAGGAAAGAACUAAUAGAAAAAUGAUAAUUGUGUGACUGUGUAUGUCUAAUAUUGAAUU
AAUCAGCAUGUUCUAAAUAUUAUGUGAUGGGCUUAGAUAUUGGUUGAGCAUAAUGUUAGU
AUGGAAACAAGUUUCUUUGGAGGUCUAAUGCAUUACUUGCAAAUUUGUGGCAUUUUCCUU
UCUUGGUUGAUUGCAACCAGUAGCCAUGGGCUAUAGGACUUUAAUAAGAUUAGCUUUUUG
AUCUGAUUUGACCUCAAGAAUUCCAGGGUCUAGUUAAUGAUCUAUUCUUUUAUGUCUGAG
UCUUGGUUAAAGGAUCUGACUUUUGUGGGAGGUGUGCAUAUUGUUUGUUUGGAAUCUGAU
UAAUUUUUCUCAGUGUUGAAUGUGUUGGAACUUAAGGAUAGAAUAACUCUUGAAAUCAUG
CUCCUCUAUGUUUGUAAAUCCACAUAUUUGAAGGUCUUAUUGUCCUACCUCAUGAAUAUU
UUAUCUCUAGUGAAGUCCUGACCUCUUGAGUUAUAAAAUAGGUCUGUAACCAUCUCCGUU
GAGUCAACAUUGUGGCUGAAUAAGACUUUUUGGCUGUUUUGUUUGAUGCAUGACAAAUGA
GUCCAGUCUUUUUUGAUUUGUUUCAAGUGCUUUAUGUGUGAAACUAAGUGAACCACUUCC
UGUUUAAAUGUUAUUUUCAGCUUAAUCCUUUGUUGCAUGCAUAGGGGUCAAUUAAGAUCC
AAUCGACCAAAGCCUUACCUCCUUGAUCUCAAACCUAUUUUGUUAAGCUUAAUUUUGUUU
AGGUUACUGUUUUGUUCUAAUCAUCACUAAGUCCGAGAAGUAUGUCUAAUCAGAAGUUGU
UUGGAUGUUGUUUGCGUAUGUGAGAAGUAGGCUUAGUCAUUUUCAUGUUCCAUGAUUACU
AAACUCAAGAUUAUUCUUGUGUUUAUGCAACCGUUUGGUCUCUCUAAAGUAUGUUGCAGA
CUGCAUUAAUUAUGUGACUUGAUUAAUAAAAUGGCUUUAAAAUCCUUGAAAUAAAGUUUU
UUUUCAGUUAGUUGUUUCUAUUGGCAGCCCCUGUGAGUUUUCUUAAAUUAAGUAAGUAUU
UUGCCUCUGGAUAUUUGAACCUGUUUUCAGUGUCUAAUAUGCCGUGAGGUGCUAAAGGUU
GUAUCCUUAUAAGAUUAAAGAAUGAUCUAUCUGUUGUAAUACAUCCCAAGGCCAAAUACU
UGACUGUUGGUGCCUUUUCUUUAAACUUGUCUGAUUUGCUAAGGAUUAAAUGUCUAAGAU
AUCUUCUUUAUAAACUAGUGGAAGUUUUCACCUUGUGUGCUGUCAAUAUCAAUCUCUGAU
AACUUUUUCUUGGAGCUUCUUUGGGUAUCACUUGAUUAUUUCUUUAUUUUUUUCCCUUUA
AAAAAUAAACAUAGAAUGCCUUAUGAUUUUUGCAUAGAUAUUAUCUUCUCCAAAUUUGUC
CAUUUUGUAAUGGUGAUGAUUGCUUGGUGUUGUUUGGAUUGCUGUUUUCUAACUAGUUCA
AGUUUCUGAGUUGAUGGGUUAAUUCUAAUGUUGUAUUGAUUGAAUAGUUGGGCAAGUGAA
UGCUUUUACGAGAUCUACAGGACAAAGAAAGUGCAUUUCUUAAGUGAUUUGCAUUUGUAC
CUCUCUCCGACCUUCAGAACGCUCCUAUAAUCUUGUGUCAACUACUGUUCGGCUGACCUA
GUUAAUACAAUUAUACCCUUUUUACCAUUCCUAAUUUUCAAUAGAUGGAAUAACUUUGAU
AAUAAUUUUCUGGUUUUGAAAUGUGGUUUGGUUAUGCUGGUGAUUUGUGAAAUCUGAUAA
AAAAGCCUAAGCAAGUUUAAUACAUAUGCAUUUUCAUUUGUCACUUGACUUAGUUUAGCU
UUAAUAUUGUUUGAUUGAUAAAUGGUAUGGAGCUUUAAAAGUAGCGCCUUUGAGCGUAAU
AUUGGGCGUAUUGCUUGCUAGUAGCACCUGGUUGAGUUGAGUUCACUUAUGUCAGCAUGU
GAAGUCUGUGAUCCGGGAGUAAGGCUUUUGGGGCUGCUUGUUAGCUGCCUCCAAAACACU
UUUAAGAUGGGGUGGCUUGCUUUAGUAGCUAUUUUCAUUUUUUAGUUUUUUUUUUUUGUG
UCGUAAGAUAUGAAACCACAGUUGUUUGAAAUUAAAUGAUGGGUUUGCUAAGGCUAGUUU
AAAGGCUUGAUUUUCUACUUGAAGUGUUUGAAUUGUCCCUGUCUAAUUUCCCUGGAGAAA
AGUUUCUUAUUUUACCAUUCUAGUUACAGUUUCCUUAUUGCAUUACCUCCGCUUUAGUUC
AUAUUUUGAGAGCCUUGGUUUGUUAUGUUUCGCGUCACCGUGUUCAGUGUUCUUGCUAAC
UCAAGUCCCGAUCUAUGUGCAUGAUUACAUAUGGAUGGUAGCUAUCUUAUUCAUGUUCGU
GUAGGGUCUGUUUGAGCUAAGCUUUGUGAGCAUGUUUUCCCAAUGUUUAAUGGCUAAAGU
GUGGUUAACCGACUUACGAUUUGUGUGAAAUAUGAUAAAAGUUAUAUACAUAGCAUGAGC
GUGUGAACCUUUCCUUAACCUGCUAUCUCAAAUGUUUGUUCUCUUACUUGUGUGGUGUUC
AUCUAAGCCUAGAAGCAUGUUAAAUUGCAGCCUGAAGUGUUGCCAUUCUGACACCAAAAU
UGACAAAUGUGUGAUGAUUACUCAUAUUUGGCAAUCAAUAAUUUCUUUCUAGUCAAAUUA
AUUAGUUGAGGGGGGACACUAAUUCGUUUCUUUUCUUUGUGUGACGUGAAAUCCAUCAGG
UCCAAACGACCCCCC
2. MFE structure-
...((((.((((((.((((((....)))))).........((((((...(((((.....((((((..(((((((........)))))))))))))......))))).)))))).((((..((((.......((((........)))).......)))))))).....((((((((((...(((((((((...((((..((
(.....((((.((..(((((((............)))))))..))..))))....(((((...((((((((((..(((.......)))..)))....))).)))).....))))).....(((((..((((((((((.......(((((((......((((.((.((((((......(((((((..((((((.((...((
((((.(((((((((((((.....((((((....))))))((((((...)))))).........((((((((((.(((((((((.((((((((...((((((....(((((........)))))))))))....))).)))))..)))))))))..)))).))))))))))))))).))))..))))))..((((.(((..
.((((((((((((((((((((((((.(((...((((.(((((......................))))).))))....)))))))))))((((((((((((...((((........((((((((..((...((.((....)).))...))..))))))))(((.....)))......((((...))))...))))...))
)))))))))).(((((....((((.((((((((((..((((...((((......))))...))))..))..)))))))).)))).(((((...)))))...................)))))....(((.(((.....))).)))((((.(((.......))).)))).(((((((((.((((.((((.((.((......
(((((((((......((((((((((..........(((((.((.((((((((..((((((.(((((((..((((((((((..((((((.....))))..((((((((.(((((.(((((((((((.(((.((((((((.(((((.(((((....))))).)))))((((((..(((((.....)))))....))))))..
......)))))))).))).)))......)))))))).......((..((((..(((((........(((((..(((..((((((((((.....((((....)))).....(((((((((((.((((.((((((((.((..(((....)))..)).)))))...(((((((((........))).)))))).((((((...
..))))))...(((......)))..))))))).)))))))).)))...((((..........((((((..(((.((((.((((((....(((((...)))))..)))))))))).)))...)))))).))))......))))))))))..)))..))))))))))))))..))))))))))).....)))).........
..((((((((((.......)))))))))).........))..))))))))))..)))))))...))))))..((((...(((((....)))))....)))))))))))).)).)))))..((((((....))).)))(((.(((((.(((((((....)))))))....(((((((((((((..((((((((((.(((..
......((((((((.((.((((......(((((((..((......))..))))))).........((.((...)).))....)))).)))))))))).....)))))))))))))...)))))))))))))..((((......((((..((....((((((....(((((.................)))))...)))))
)...))..))))......))))......))))).)))......)))))))))))))))))))(((((((.....(((..((.((((((((((....(((((((.(((((((((.....))))))))))))))))....(((((((((((..............))))))..)))))(((((......)))))))))))))
))..))..)))...)))))))..)).)).)))).)))))))))))))(((((((.(((....))).))).))))))))))....((((((((......((((((((((((.....((((.(((((((((......((((((((((...)))))...)))))......))))))))).)))))).))))))))))((((((
(((.((((.............)))).))))))))).))))))))..........))))))))))......................))).)))).)).))))))))))))).............(((((((.....((((((((((((((((((.((.((((((((.....((((((......((.(((....((((.((
((((.((.....((((((...)))))))).)))))).))))..))).)).....))).))).....)))))))).....(((........)))...((((.(((....)))))))......)))))))))))))))))..(((((....)))))..(((.((....)))))......))).....)))))))..))))))
.)).))))......)))))))))))))))))...)))))..)))..))))...)))))))))....))))))))))..((((.((((((((...........)))))))).)))).............(((((.......)))))..)))))).)))).....(((((...........((.(((.(((......))).)
)))))))))......
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-760.80] kcal/mol.
2. The frequency of mfe structure in ensemble 2.58461e-41.
3. The ensemble diversity 769.53.
4. You may look at the dot plot containing the base pair probabilities [below]

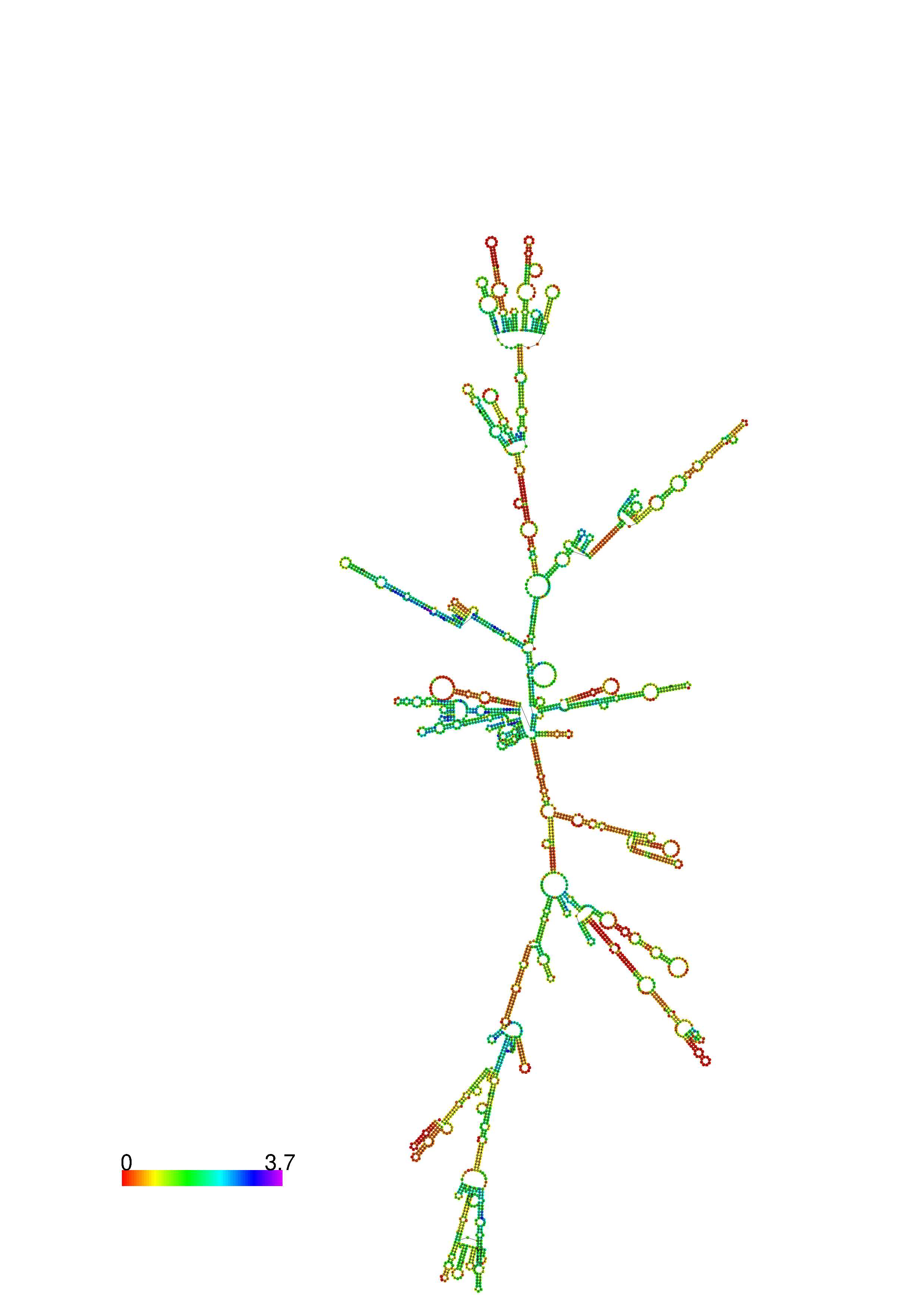
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
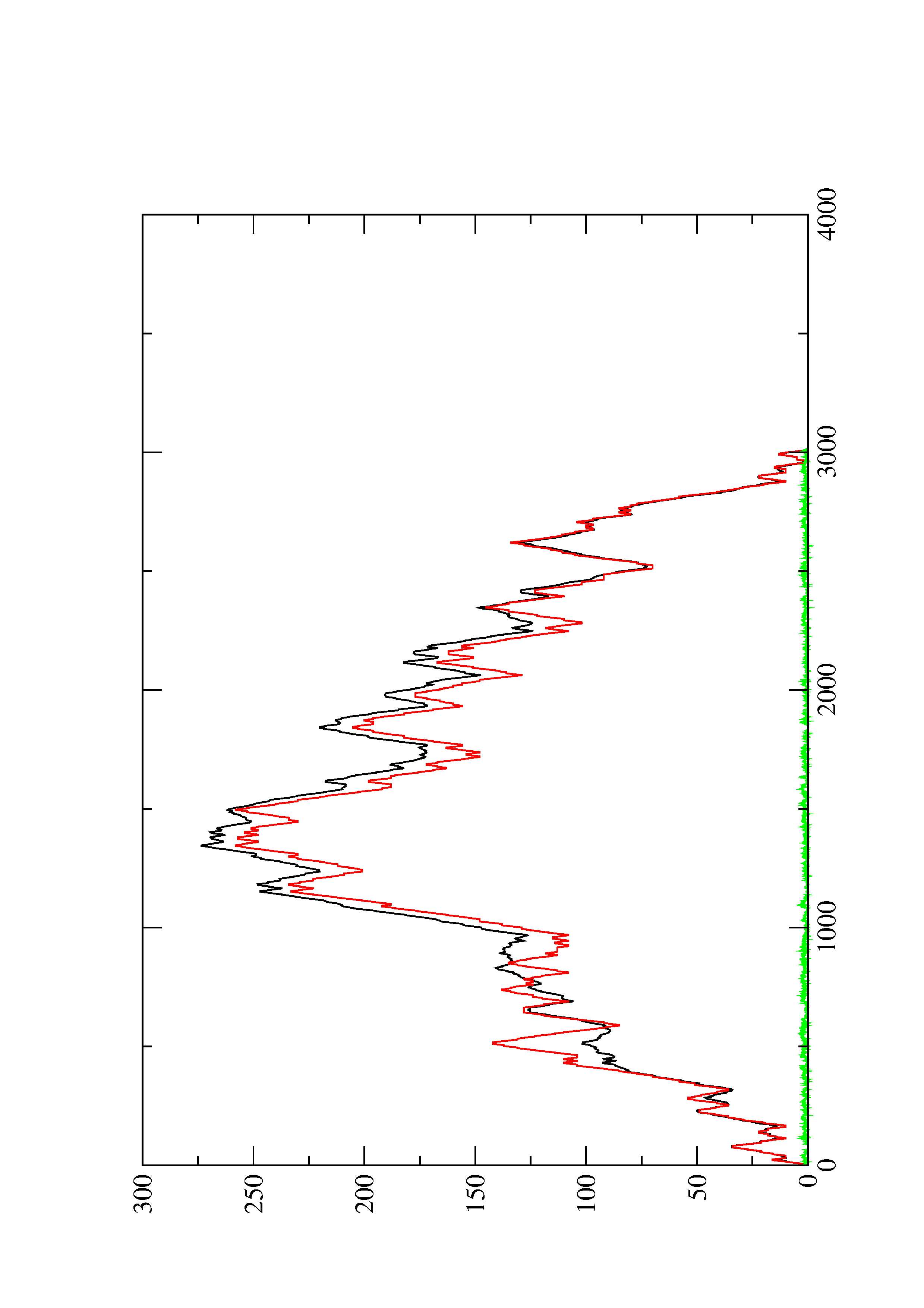
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.