lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01035159.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-789.70) is given below.
1. Sequence-
CAUGGCUUUCCUGUGUGUGCUAAGUUGGGUGGUUAUUAUUGCAUUAUUUCAUUUUUCGUU
AUUCUUCUUUCGAUUGUUUAUAGGGUAGUGCCUUCCAUUUUGAUUAAAUUUCAUUGUGAU
UGCUCAUAUAUAUAUAUCCUUAAGAAAAAUUUUAGACUGUUUGGUAGGUACUAGUUAAGU
UGAUGGUCUAUCGGAAACAACCCCUCUGCCUUGACGAGGUUGAGGUGAGGUCUGCAUAAU
UAUAUUGGGAUGUUGUUGUCAUAUAAUUCUUGAUCUGUCGAUCAUGUCUUUCUAAAUGCA
UCUAUUUCCAAAUAGUUGGGGUAUUGUUAUUAUGUCCGACUCAGAUUUUUGAUUGCUACU
AGAGUUUGAAAGUUUCGGAAUAGUGAUAAAUAAUAUUCCCUAUGACCCCCAUUUUGAACU
AGACGUGAAUUAAAGAAAGUAGAUUUUUUUCGCCCUAUGACUCCCAUUUUGAACGUGACG
UGAAUUAAAGAAAGUAGAUUUUUUUUGCAAUUUAAUUAGAUGUGGAUAAUAUACCAAAUG
UACUUCCAUACCCAAUAAUUUGUCGUUUAAACUGGUGGGCCUACGGCAAUUAUGUCAAUU
GCAAAAUGGGAACAAAAGUUAACUGGUUUCUAAAUAUAGAAAGAUGUCAAUCUUUUGCAG
GGGGAAAAUGGAAAUAGUGCCAAACAACUUGUUUGAUACAGCGAGUGAUCAAAAUUAUUU
AUUGUUUUGGUGAAGUGCAUGUUUUAGAAUUUAACAAGUGAUUAUGCAUCAUACCUUCAU
CUUAUCAUCAUUGUUCUCCUCAUCAUUAUUGUCCUUUUAGAGGUUCUGCAUAAGUGCACA
UUUAAGAUCUUGAGUGUGGUGCACAAUCAUUCUUUUUAAGAGUCCGAGCAACAUAGGUUG
UCAUCUCUUGUCUUCAAGCAGUGAUGUCUUUUUUCACCUCUUUAGCAGUUUUCAUCAUUG
AACUUUGAGCUGCUUUGCUGGCCAUCCAUACUGACCAAGUUCCAUCCACCUCUACUGGUU
AGCCAUAUGAGUAUGACAACUAUUGGGCCAAUCAUCAGUGACAAAGCCUUCACUGGAUGG
AUCACAAAAAUGAUCCUUUUUUUUUUUUGCUGUUUCUGAUACAAAACAAAAUUUCUCAGU
CAUGUGGUGAUCAGACUUUUCCGAAACCUAUCAAGAUAACAAUUCCCAACACGAAAUGGU
CCAAAAGUUAGUAAAGUCCUAGAGGAGAGCUGAUAUAGUUUUUCACUUAUUAUGACAGUU
CUUGCUAAGUUUCGACACUCAUUGGAGCUUGUAAGAUUCAAGAGGAGUUUGUCAGCCACC
AUCUUAUGCAGCUAAUUUUCUAGAGAGUUUAUCUUAAUACGUAUUGAAAUUGUCAGACAC
CAUCAAACAUAACAGUAAAAAGAUGAUAUCCUGUUAUAUAUUGGAUGCAGAAUCCUUUAA
AAGCUUCACUGUCUAUUAUCAAUGAUAGCUGCUUCUGCAGAGAGUAAGGAAGUUGCUUAU
GUAGUGAUUGAUUAUUGAUUAGUGUGUUCUGUAGAAUGUUAAGCACUGACUGAAGCAAAA
CAGUGAUGCUCAAAUAUUAGUCUAUUGGUCGUUGAUAAAACUAGAGAGCUGUGAGUUUUG
UAGAAUCCUGUUAUAUUUGAACUAUGAUUAAAUUGCUAUUCUCAAGAUCUGUAUAUUGUG
AAGAUAGUUUAAGAAGUUACUGCUUUUGAAAGAAAACAUUUUUGGCUUUUAUUUAGCUUC
UAUUCAAAGUUCUCCCCAUCUUUACUGACAAUACAUUUCUCUGUUUGUAUCAAUAAUUGU
UAUAUUACCUGGGAAUUUUGCUGCCUCAGUUCCUGUGAUCACUUAUUGAUGUAAGGAUUA
CAUGUUAGUUCUUUGACCAAAUCCGGUCUUUUUCUUUGGACAUGUCUAUUCUGCUGGAAA
GAAAAUUAUUGCUCUACUUGUCAAAAAAUAUUCUGCUGGAAAGAAAAUUUUGCUCGUUAA
UAGGGAUAGUUAUAAGAGAUGUCUAUGGGAAACAUAUGAUGUCCUACUCUAAUGACAAGA
GCCAUCUGUGUGCAGUGUAUUUAUGUCCUACUCCAACUACCUUGUGGCAAGUUAAAAUGA
UUUGAUUUUCUGGAUCUAAUUAAUUGCCCGCUAUUUGGUAGUAUUCGAGGCGACAACAUU
UUGGUGGAUGAACAACUUGAUGCUAGAGAAGGAAGACAUAGAAAGACAACUAUCGUUUGU
ACUUUUUGGCUAUUAUGGUGAGAAACACGUGCUAGAAUCAUUAAAGGAUUUGAGAAUUGA
AUUUGAAAAGUCAAAUUUCUCUCUUGCUUGGUUUGUGUCGUUGAUAUGAAGAGAAAGUAU
CAUGCUGUAUAGAUGGUCAGACUUAGGGAGCUCUAACGCAUACUUUGAUCAAACUUUUGU
UAGUUUGUAUGACUAACGUGGUUGCACCAUCUAGAUUUGAAUCAUGAGAUGUACUUUGAC
UUAUUAAAAGAUUAAUUGCAUGCUAUUCUUUUUUUAGUUAUCAGUAAUUAUAUGGUGUUC
UCUUGCUGCAGAUUUUUCAGAAGGCCCUGAUAAUAACCAAGCUUCUGCUACGUGCCCUGA
GAAAGGAGAGAGAACCCAGAAAACUGGAGAGACAACUGCUCUACUUCUAUCCAUAUCACC
UUUUCUUGAGGUAGUCCCAACUUUAUAGCAAUGUGGAAGGUGCCGCGGUACAUGUAAAUG
UCAUCUGGCCAAAGGCACACUAGUGUAUUGCUAUUACUUUCAGCUAUUUUUACAUCUUGC
AGGUCAUCAUCAGCUUCAUGGCAAGGAGCCAAAUGAAAUUUGGGAUGCUGCUUCUAGUAG
UUUCUGACCAUGCUUCGGUCCUGGAACAGUCAUUUGCUGCUCGGGACUUUCAGUCCUUUU
CUGCAGCUUUUAAUUAUCCCUGAAGGAAUACUUGAAUGCGAUGUUUUCAGACUCGGCCGA
GCUUAGGGGUCGGGGUUCAAUGGGUUUGACCGGUUGACCCGGACGUAAAAACUCGGUCGA
CUCAGAUGACGUCAUAAUGAUGUAAUAAAUAUUUUAAUAUUAUAAAUAUAAAAUCUGUGU
UUAGAAAUUAACGACAUAACUCUAUGAAAGAGUUGAAUAUUUGGAAGAUGAAAUUUUGAU
AUUGUC
2. MFE structure-
.((((((.(((...............))).)))))).....((..(((((((((((((.(((((..((((......((((((((((.((((((((.....((((((((......(((((....)))))............(((((....)))))((((((((..((((.(((....((((..((((((....((((((..
..((((.(((((...))))).)))).(((((.(((((((((((.((((((.(((((......))))))))(((((....))))).....(((((((.(((.(((((((((.((((..((((((((((((((((.(((((.(((((((((...........))))))))).....)))))...))))).))))))))))))
)))..(((((.........(((..(((((..(((.........)))..))))).)))....((((((((((....((((((((((((((....(((((.....)))))..)))))))...((((((..((((.....))))...)))))).........(((((((.....((...))....))))))).)))))))...
.)))))))))).((((((((.((...((((((....))))))...)).)).))))))..)))))....))))))))))))((((((...))))))..((((((....((((((((........))))))))....))).)))))))))).....))))))))))))))....)))))(((((((...(((((...(((((
..((((.......((((((((((((((((((..((((((((((((....))))))...))))))....((((((...))))))((((.....(((((.((((((((((((....))))..((((.........))))......(((((((((((....))).....))))))))....(((((...(((((((((((.((
.............)))))))))...))))(((......)))....))))).....((((((........))))))...((((((......))))))........((((((((((.((((................))))....((((((.((.(((..((.(((.((.(((.(((((((..(((((((........))).
...(((((((((...((...(((((...(((((((.........((((.((.(((....((((((.(((((((((......))))))))))))))).))).)).)))).)))))))..)).)))...)).))))))))).....))))))))))).))).)).))).))..))).)))))))).......))))))))))
)))))))).)))))......)))))))))))).))))))))))........(((((((((((((((.((((((((((((((((((((...(((((((.....)))))))..)))))..........)))))))))).))..)))...((((((((((.........))))......)))))).....)))))))))))..
.))))(((((((((...(((.(((((...((...((((((.......)))))).)).))))))))((((((((...(((((((((((((...((((((((((((((...(((((((......(((.........(((((((.....((((.((..((((((.(((.....((((.(((((((..((((((((((...(((
(...(((((..(((((.(((.((((((......(((((((.(((((....))))))))))))............((((......)))).........(((((((..(((.((((.....(((........(((((...(((((.(((((.((((.......)))).))))).........(((((.(((........(((
(((((((.....)))).)))))))))))))).))))))))))..)))....)))).)))..))))))).......((((((..((((..((((((...((((((......))))))...))))))..)))).....))))))....)))))).))).))))))))))...))))(((((((((((((((....(((((((
(........)))).))))....)))))))).)))).))).....(((((.(((((((((.......)))))((((...((((((((....)))))))).)))).....)))).))))).)))))))))))))))))))))......))).))))))..)))))).))))))).....((((.(((((((((((((...((
(((((.....)))))))(((((...)))))..)).)))).)))).)))))))(((.(((((....((((((...............))))))...)))))..)))........((.((((((((.((.....((((((((..(((..........((..(((....)))..))))))))))))))).))))))))))(((
(...(((((((....)).))))).))))..))).......)))))))))))))))...))))))(((((((((((..(((((((..(((((((....))))......)))...))))).))..))))))))))).......)))))))))))))...)))))))).))).)))))).))))..)))))...)))))....
))))))).((((...)))))))))))))))))))).)))))))))))))))....(((((..((((((...)))))).....))))).)))))))).....))))).............((((((.((.((.(((((((((.......(((.((((.((((.(((...))).)))))))).))).......)))))))))
.)).)).)))))).....(((((.....)))))(((((....((((((((.....))))))))...)))))...))).))))))))))))))..)))))..)))))))))))))..))........
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-847.02] kcal/mol.
2. The frequency of mfe structure in ensemble 4.05695e-41.
3. The ensemble diversity 710.25.
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
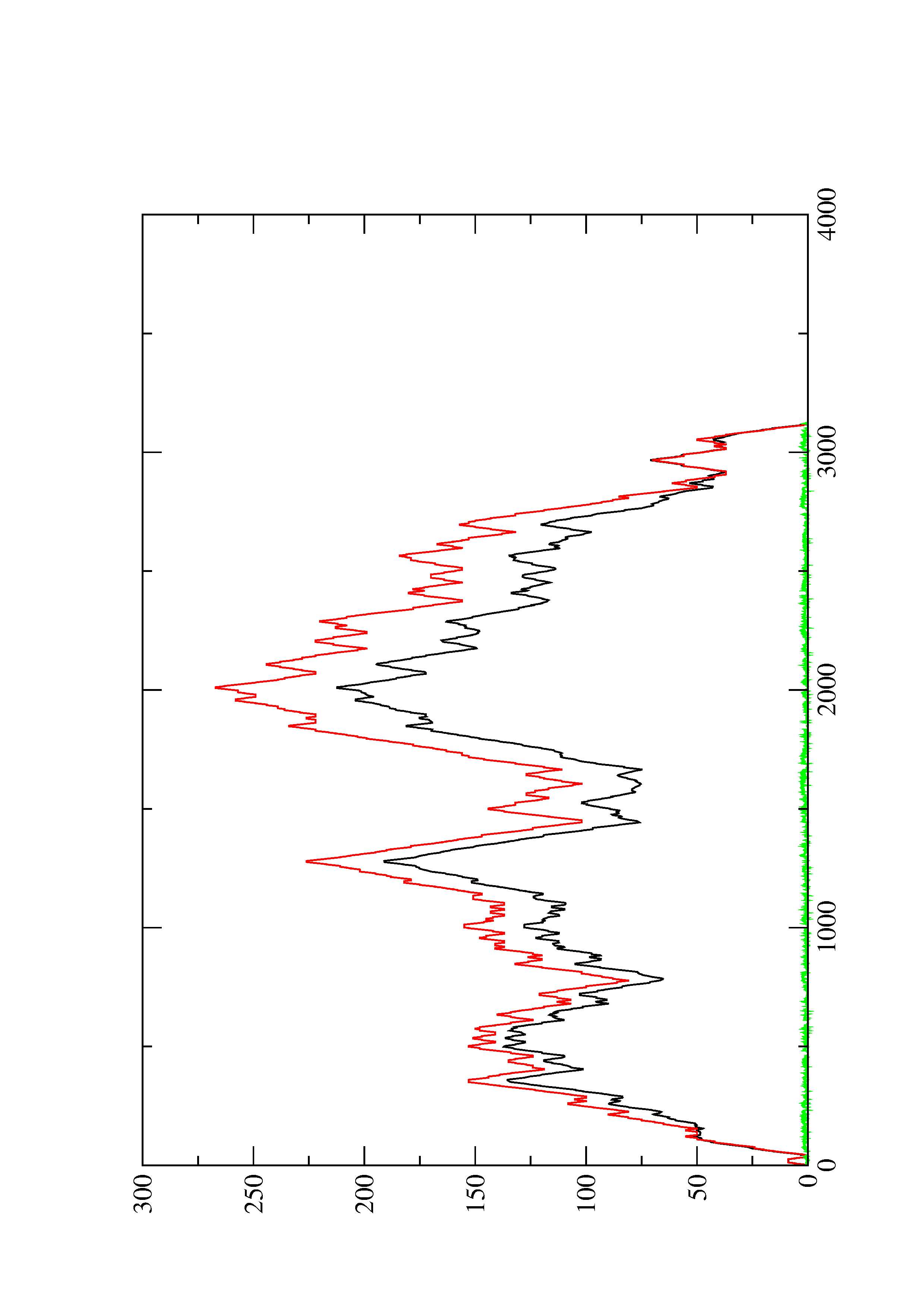
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.