lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01038710.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-702.40) is given below.
1. Sequence-
GAAUAUCUCAAAGUUUAGGGUAUAAAUUAAAAAUAUUUUAAUAAUCCUAAAAAAAAUCCC
CUCCUUUCUCUUUCGUAUUUCAAACUUAUUAUCCUUCCCUUCGUUUCUUCACCUUCCCCC
UUCAUCUUCUUCUUCCUUAUCAUCUUGGUCAUUUCUUGCUCGUUUUCUUCACCUGCCCCC
UUCAUCUUCUUCUUCCUUAUCUUCUUGGUCAUUUCUUGCUCGUUUUCCUCUUAGACUUUC
UUUUUUUCCUUCUUCGCUCUUUUCCUUCCUUUUCUCAUCUAGUAUCUUCUUUUUGUGUUC
UCAUCAUUUUUGAUUUAGUUGUGAAAAUUUGGGGAGAAUAUUUGCUUAAAAAAAGUUUGA
AGUAUUAGAGGAAUUUUGCAGUUCGUCAAAAAACAACAGAGAGUUGUUUAAGCCGUCGAG
AGAUGAUGUUUAAACAAUUGAGAGUUGUUUAAAGGACGCUGAGUUGUUUAUAGUUCAAUC
AACCAGAAGAUGUUUAAACAUCGGGCAAUUGUUUUAAAUAUCAAAUAGUUGCUUGGAGAU
UACAAAAAUAUCUGCUAGUUGUUUGGAGAUGUGAAAAUAUUUACUACUUGUUUAAAAAAA
UGUUUAAACAUCUAUUAGUUGUUUAGUUAUUUGGAGGUGGUGAUGACGUGAACGAGAGGG
GAUGGAUGGAGGAUGGAGGAUGGACCACAUCUUAGGUAUGCGUCAAGGUGCACUGUUUCU
UAGGUGUGAAAACAUGAUUAUUGAAGGUGUUAGUGAGAUUAUAGGAAAAUAUGUUGCCUU
AAAUUAUAAUAUCAGAGGAUUCCUGGAAGAUCUAACAAAAGAUAGAAAUUAAUGAACCAU
GAAAUUCCAUAUAGAUGAUAUGAACUAGAAGACUCGUACUUACUAAAAGAUCAAAAUGUU
AGCGAAUAGAGGUACGGCCAGUAUAGUACAUUAGAGAAUUCUAGUAUUAUAUCAUCUUAA
GUAAGACAUCUCGAAAUAUAUGGGGCAUGGAAUUAAAAGAGAUAUUUGAGAGAGAGACUA
GAGAAAAGGGGACACAAGUUUUAAGUAUGUGGUUAAGGGGGCACUUUUUUAAUUAGUGAU
AAUGACACAUAUUUUAGAAGUCCCCAAGUUUUUUUAUUUACACAAACCACUCUUUUCCCC
CCACCCUUUUCUCCACCCUCUAGCCCCAACCAUCUCAAGCACCAUUCUCUCAAUUUGUCC
UCUAUAUGCACUACUUGCACACGGAUGUGGUCAUUCUAAUCGUCGCUAAUCUUGUAUGAC
CACUAUUCGUCUCAGCAUUCAUUUUGUACAUAUUUGGGUCUUAAGAGGAUGUGAGAAAUU
GCUCCAAAAAAGUUUUUACAAGACAAAACCUAUCUGCACUAAGAUGGACUUUAUACCCCU
ACUCAAAGAUGGACUUGAUACUUACAUUGAGGCCUUGAGGGACUCCUAGCUAUUUAUGCA
GCAUUGGGGUAGCUGCUUGGAGUUUCUAAUAAGUUUGUUCGAGCAGUCUUUGUAGGUUCA
ACAGCUUUGUGGUGGGUUGUGUGUUGCUGGGGCAGCCUGCUGAAUGUGUGAACGGGCAAU
ACCCGUUGCUGAUGCUACACUGGGUGUUGUGGGCUAGAGCUUAUGUAGCUAUGACAUAUG
UAGUUGUAAAUACGCGUAUUAGAGCAUUACCAAGGUUAGGAGGUCGUCCAUGACACAUGG
GGCACAUUGGAGACUUUGAUAGUCACUUAAUGUAAUUGGCUGCGACUGAUGAUUCAUUUG
ACUAUGUUAAAUUGCACCCAUGCAUAUCAUAUAUUGUGUUGCGUAUGGAUCCUUGCAGGA
CUUUAGCUCUUUGUGCUACUUCAUGGGUAUGCAGUAUAUGCAUGCCAAAUAGAAGAGUCA
AGAGUGUAGACAGAGAGAAUUUACCCUGGCAGAUAAGAAAAAAGCAGCUUAAUGCAAAGC
AGAAUCAGUUUCCCUUCUUGAAGCUCUAGCUCUAGUGAACCUUCAUGUAGGUAUCUCGUG
GAUCACUUCAUCUGUGCAUCGAUGCACUUGUUCCCGUGUUCCUUCCCUUAGGUUCUCGCG
GAACUUGAUUAAGUGGUGGUUGCUUGAGCCAAAUAGCUACAGCCUUCAUCAUAAAGUCUG
UACCUGUUACCUGCGGCCUUGUGGAAAACUUGGGGUAGUCGUCUUAUCUUACUGCGUGUG
CUUCUCAGGUUUCUAAUUUCCGUGCCUUUUUUUAAAUGAUAGAGUGGUUUCACUUAUUGG
CUCUAUAAUCUUAUUACUAGUGGUUUAACCUGUAAAUCUUCAACUUAGCCAUUCUUUGAG
GGUGAACUUUUGUUUUGGUAAUGUAAUACGAGUCAAUUUGUGUAUGAAGAAUAUACUGAU
UGUUUGAAUUGCCAUCCAGGUUGUCUGUCUGGGAAUAAAGUGGUACUGCUUGAACCCGUU
GUAGAUGAUGACCAUUCUAUUAUCAACACUUGUGUGACCUUUCAUCAAGGCUAUUGUGUG
UUAGGGAUUUACUUUUACUGGAAUGUUUUAAAGAUGAUUAUAUAUUUGAGUAGGAGGUGU
AAUGGAUUAUUGAUCCUUAAGACGCGCGACUGCUUAAUUAUGCAAUACUAUGAAUUUUAU
GGAGUGGUUGACAAGCGUAACAGUGGGAUUUGGUUAUCAAACAAGACUUUUGUUUCCAGA
GUAAGUUAUCCAAAAA
2. MFE structure-
......(((((((((((((((....(((((((...))))))).))))))))..............((((((...(((((...(((.((((.((((.....((.((.((.(((((((....((((((((((((((((............(((((((((.(((((((((.(((((.(((.((..............((....
......)).......((((.....(((((((...(((((((((((((............(((((((...(((((.((.((((.(((........(((....))).......))))))).)).)))))...)))))))..........))))))))...)))))...)))))))....))))..((((...(((((((...
.((((((((((((((((....))))..))))))))))))....)))))))....))))(((.((((.............))))))).(((((((((((((((((((((((((((........)))))))))))))............((((((.((((....))))))))))..(((((.........)))))......)
)))))))))))))(((((....))))).....)).)))))))).))...))))))))))))))))..)))))).)))))))))).(((((((.(((..((((......)))).....))).)))))))..............)))))))..)).))))....)))).)))).)))............))))).)))))).
...(((((..((............................))..)))))...((((((((((((((((((..((((((((((((.....(((....((((....))))..)))......)))).)))))....)))..))))))).)))))))))))(((((.((((..(((.........(((((((((((.......(
((.((((((((.((((....((((((((((............(((...((((((...((((.((((((..(((..(((.......)))..)))..))))))))))((((......)))).....)))))).))).........))))))))))....))))................(((((((((((((........((
(..(((((.((....(((..((((((((((((((.....................)))))))).))))))....)))........)).)))))..))).....))))))))(((((.((((((((((.....((((((((((.......((((((......))))))......((((((..(((((....((.((((((.
.....)))))).))))))).........(((........)))...))))))((((((((((....((....))....))))))))))..((((((((.(((((((((((((((((((((((.((((((.((((((((.((.....))((.((((((.....)))))))).(((((((((..((((.....((((((((((
((..(((..((((((.......))))))..)))((........)).....(((((...((((((((((((((...)))))((((((((((.((((.....))))..))))))).....)))))))))..(((((((((((((...))))))).....((((((.((.((((...)))).)).))))))...((..((((.
......((((((((.(((((((.((((((((((.....))))))))............)).))).)))))))))))).......))))..))..............((((......)))).))))))...)))..)))))))))))))))))......))))...)))).)))))......(((((((.(((.(((((..
..)))))..(((((((....(((......)))....)).)))))..))).))))))))))))))).))).....))))))))))..)))))))))).))))...))..)))))))).))))))))))..))))))))))..)))))...........))))))))))))).)))...)))))))))))............
.)))..)))).))))).(((((...........((((..((......))..))))...........)))))..)))))))((..(((((....(((((....(((((.((((.(((((.((((((.....)))))).))))))))).)))))...))))).......((((((((((((((....((((...........
)))).((((((((((((((((....((.(((((..((((((((((..((((..((((((((((((((((..((((((((.((((((..(((......))).)))))).))))))))...............)))))))..)))))))))..))))..(((((......))))).....)))))).)))).))))).))..
.)))))))..)))))))))......)).))))))))))))..)))))..)).....
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-748.70] kcal/mol.
2. The frequency of mfe structure in ensemble 2.35133e-33.
3. The ensemble diversity 563.89.
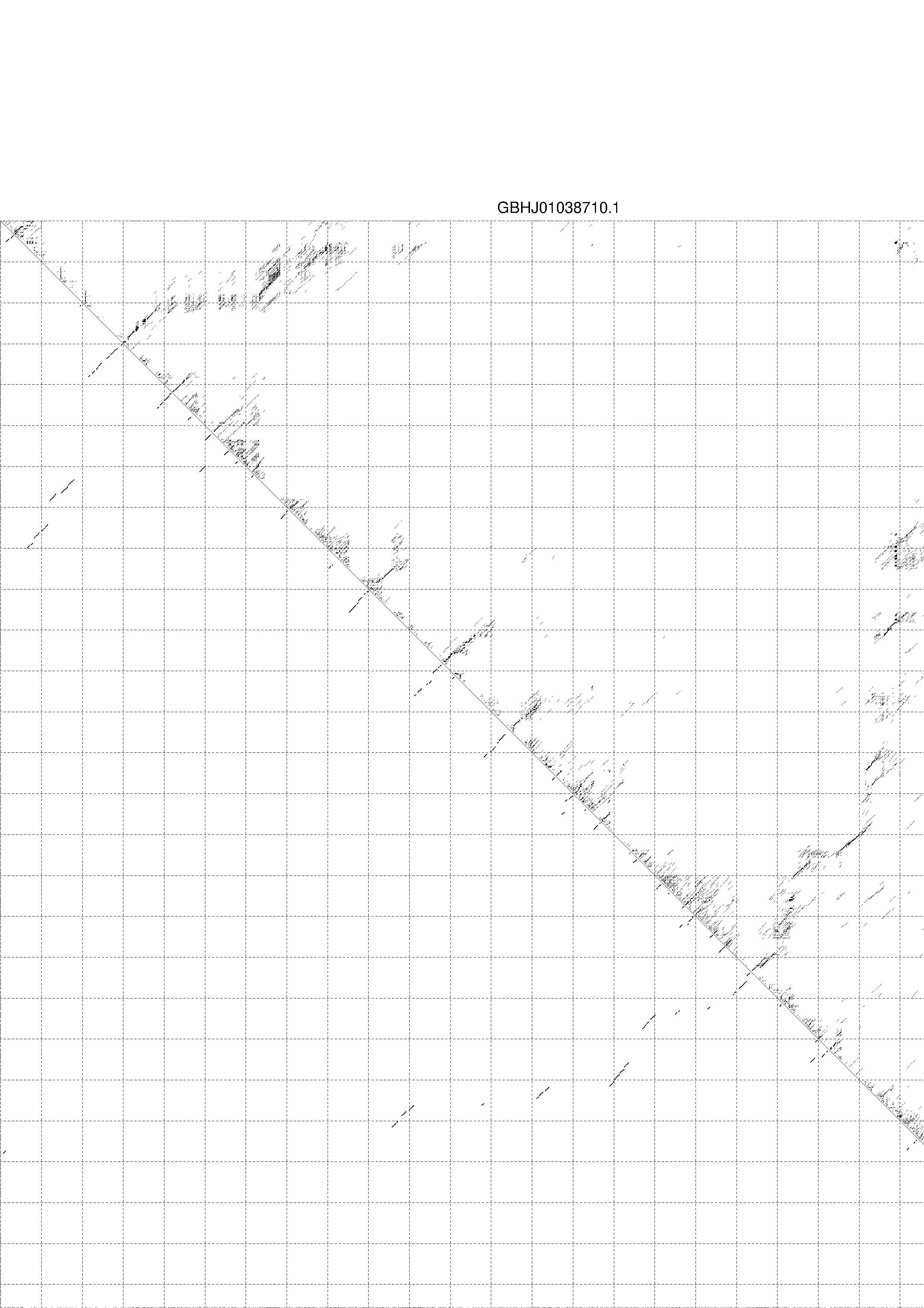
4. You may look at the dot plot containing the base pair probabilities [below]
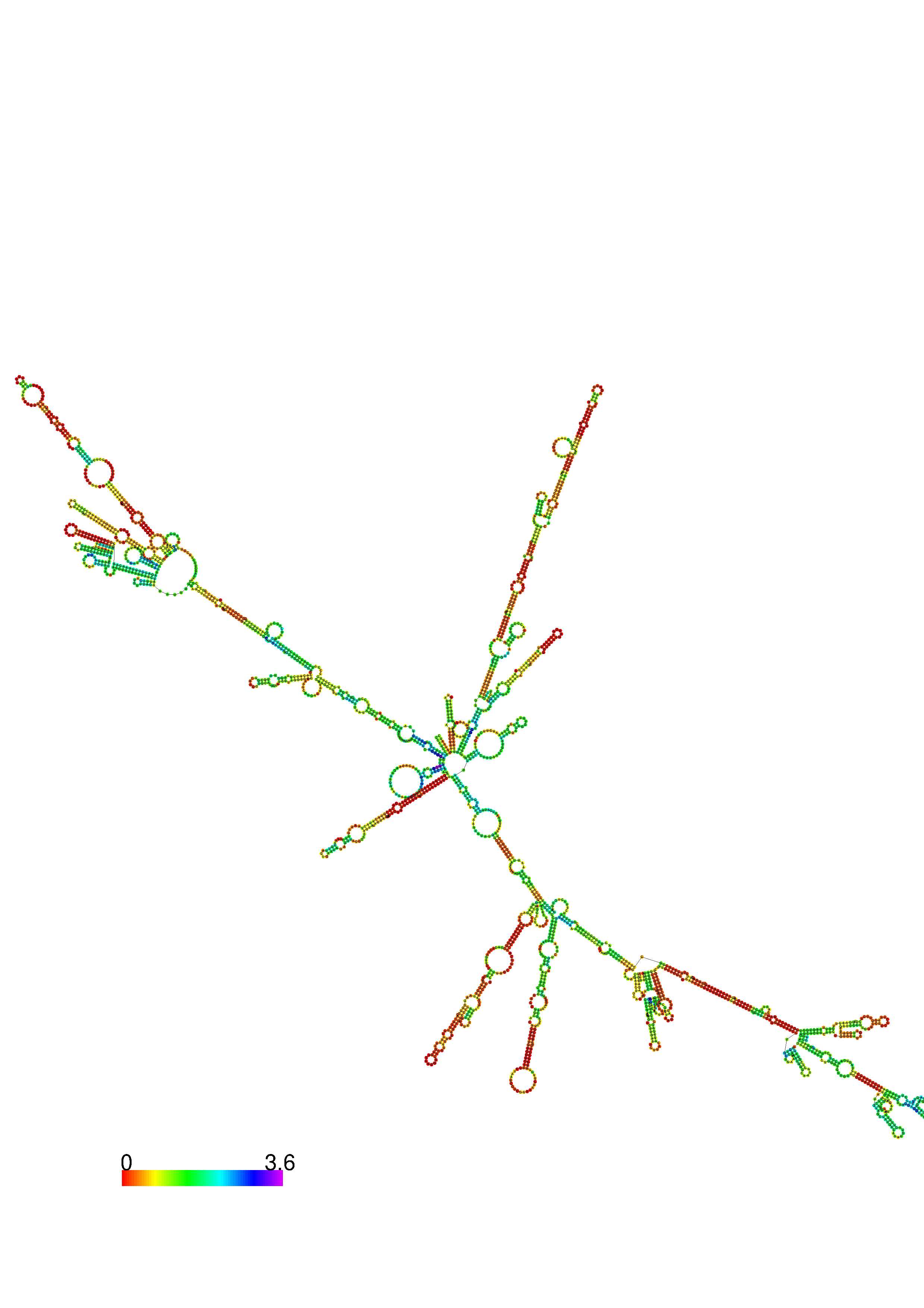
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
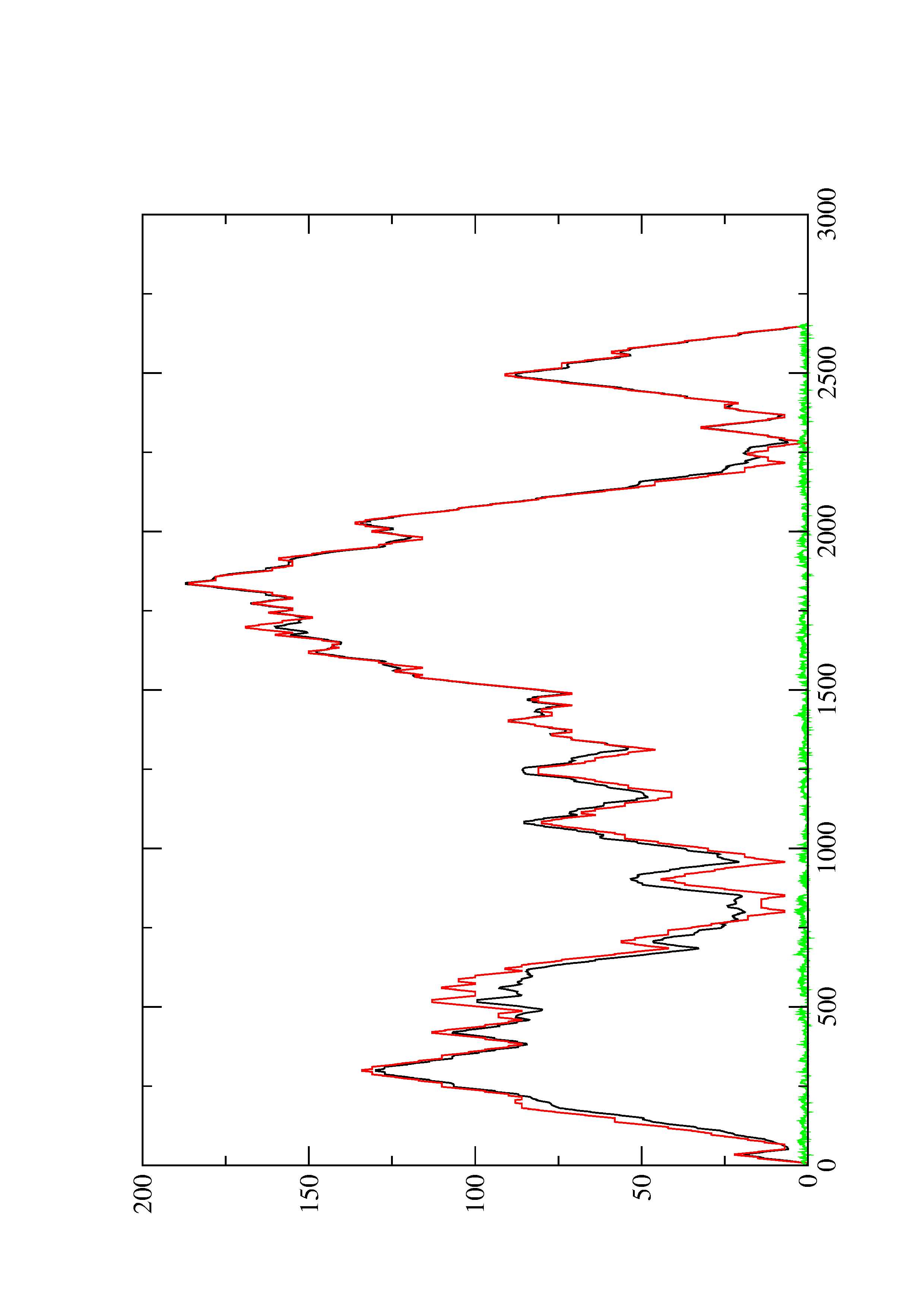
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.