lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01038725.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-625.80) is given below.
1. Sequence-
GGCCGAUAACCACUAAUACUAGUCGGCCCACUUUCUCCUCUCUCUAAAAUAAUACACAUC
CCUCUAUCUAACUCUCUCUCUCUUUCCCCCAACUCCGGCCGAUAACCACUAAUACUAGUC
GGCCCACUAUCUCCUCUCCCCUCUUUCGUUCACUCUCUUCGCUGAUCAGCAGCCAUCGAU
GGCCGGGAAAACCCAUCAGCCUCCACCCUAAUCGCCUCGCCGUUCACUGAUCUCGACUUA
UAGAAAGAGAUACUUGUUAGACAUUUAUUGAAGGAAUUUCUUGUUCUUUCUGGGCUUUUU
UCUUUUUGGAAACUUUUAGCUCAUUUUGGACUUAGACGAGCUCGCUUUAUCACAUAUUCU
UAGUUAUUCAGGGUAUAUCUUGAAGUUCUUUUUGUUUUUCCUGAUUGAGAUGAGCUUGCG
GAAGGUGUGAAUAUUUUUUCACAUCUUCUUUGGUCUUUUGGAUGAUACUAUGUUUUGGUU
UUAAUUUUUUAAAGUAAAAUAUUGUGCUAGUGUUAUAAUUUUCACUGAUUAAUUUUUUCU
GUUCUGGAUUCAAAAUGAGCUUUUAGAAGACUAAUCAAUCGAGAUUCUCAGUUGUGUUAA
UAUAUAUGUUGUUGGUAUUAAUUGUGGAGUAUAUUUCACUUAUAGCUGACACUGGAUGAA
UAAACUUGAGACUGAAGUUGAUUCUUGAAUCAACUAUUGAUCUUGAAUUUUUUUUUAAAG
AAAACUGUUGAUCUUGAAUAAUAGUGUGUAGAUAAAAUUGAGUCUCCCACUCAUUCAAAA
AUGCCCUGAAGUGAGUUGAUGUCAAAACCAUGAUCAGUACAAAUAGCUCUAUUUUUUAGC
UACUGUUUUUCAGUUUGUUAAACUAUAGUGAAGUAAGAUCUUAAGAGUUGCAUCUUUCUU
UAUUGUGAGUGAAACAAUCAUCUUCAAUUUGGCAAUGAAAUUUUCUAUGAGAGUUUUGCU
CUAUCUAUGGCAUUUCUAAUCCUCAAUAUUUUGUUAUGGAAUUUAUAUUGAGGAAUAAGA
CUCAUAUUUUCGAAGAUUUGUGCCCAUUCAAUUUACUAUUUGAAUUGUGGCUAAGGACUU
UGGUAAGAAGCAGGAGUUCUGGUUGAUCUACUUAGAUGCUAAGGACAUAUAGUAUCAUAC
UAAGUCUAGUUUCUCCUAAUAAUGUUGUUAUUAUUGAACCUUUCAAUUACUUCAAAGUUU
CUUUUUUUUAUGCUGUUGUACAACUCUACUAGUCAUGCAAUUGAGAAAAACAUAACUGAA
UCUACUAACUGCAUUUUCAUAAACAUCUGUACAUCUCUGGAUGAGCAACUUUGCUAGUUA
CCAGAGAGCUUGGAAUGUAUUUAUAAUUAUUCAAACAUCAGUAAAUCUGCAUUCAACAAU
UUUUGUGAGAAAUGCUAGGUGAAGCUGAUAAUCAUAACAAUAUCCGAAAAACAUAGAUAC
AUAAAGAGCUUUGACACAUGGAUUCAUUAUCUAUAACUUAUGAAGCAACUGUUGACUUUG
CAGGUUGACUUGUGGGAAGUGUAUAUGAUUUUCACUGUAGGAUCUUGAAGUUAUAACUGG
GUGUAAUUUUGAGACUGAGAAGGGGAAGACAAUGAUAUUAAUGUGUUUAUAGACUUGAAU
UUUUUAUGAAUAACUCCAUGGUGGUAUUUUGAUCUGACCGGCCUAUACAUUAGAGUUAGA
AGUUGAAUUGGCAAAAUUUAAAGAAAUUAACAAAGAGUUGUAGGAAAAGCAGGUAUGUUG
UCAUUGAAAUUCACAGCACCACUAGAAGAAAGUUAUUGUAAUAACUUUUUGCUUUUAAAU
GUUUUUUGGAGGAUGAAUUUAUCUACAUGCAGAAAAAUCAGGUAGUGGCCUCUUUUAUGA
CUAACAAGUAUCUCUUUUAGCCAGAUCACAAUAAUGGUUAGGGGAUUUUGUAAUGCUUUA
UGGUUUCUGGGACCCUCUAUUGAAUAUGAAACUACUGAUGAGUGGAAGGUGUUGUCUUUU
AAUACCACUGACUUGACUAGAGUCUUUCUUUCUAUAGCCUUCUUCCUCAAUCAAAAUAUU
AAAAGGAUAGGCUGAAUUUACAUGUUUGCCUAACCAUACAUAUCUAUGAACUUUCACUUU
CUUUAAUUGAAGAGACUGCCGGAGUAUGUAAGUGAGGCUUGGAACUUUCUUCCUUAGUUU
AAUGUGGAUUCCUUGGUUGUGUUAGUUGUUCUUGUGGUCAUUGUUUAUGACUCACUGACU
GUCGCCCUUUGUAAUGAUCAAAUCAGCUAAUGAUCCAUAAUAACCUGAAUCAUAUAAAUG
AAAAGAAGUAUCUGAAGACCCAUUUUCUUCUUGUUGAUAAACACUGUGGUUAAUGAUUCA
AGCAGCCCCAGGGAUUUUAUUUUGUUCGUGUUGGGUUUUUAGUAUGAUUGGGGUUUGAUC
CUAUGACCUUUGGGUUAAGCUAAUGGAUGGAAAGUGAAGGGACUUUGUCCCACAUAGGAU
GAAGAAAAGAAAUUUUGUUGGGUAUAACAUAAAGCAUUGGGAAGAUGGUAUGACCUCGCG
ACAUAUCUCUGUUUGGAUUGGCUCGGUGGCACACCUAAAUUCUUAACAGAUACCUCCUAU
UGGCUAUAAAGUCAGAGUUUUAUACUGAAAUUUGAAAUCCUUUAC
2. MFE structure-
(((((((..............)))))))....................................(((..((((((.....(((((((((((((...(((((((..............))))))).................(((((..((((.(((.((((((.(((((.((.(((((((((((.(((((..((((.(((
....((...((((.(((.((((((...((((((...((....((((((((((((((((((.(((.....((((((((.....))))))))........(((((....)))))((((((((((((((((((.((((((((((((((((.((((.........((...((((((......))))))...))...........
.)))).))).)))))))).((((((((((((......))))))))))))...((((...))))..(((.((((((((.(((.........))).)))))))).))).(((((..........)))))...............))))).)))))))))))))...))))).........((((((((..(((((((((...
...))))..(((((((..(((..((((((....)))))))))..)))))))((....(((.(((.((((((.(((((((((((....)))))))).))).)))))).))).)))....))..)))))..))))))))......(((((((((((((..(((((.....))))).....(((((((((((.(((.(((...
.....))))))..))))...(((((....)))))((((((.((((.....))))..))))))..(((((((((.(((((............))))).)))))))))(((((((((..((((....(((((.......)))))....))))..))))))))).......)))))))....((((((((.((((((((....
........((((........))))...((((..((((((.((((.(((((((((.((((((((..(((((((.((((((((.((.....)).))))))))((((((((........(((((.((((((.(((((((((.....((((....((((((......(((.(((..((((.((((.......(((((((((((.
.(((((((((....((((((..(((.....)))..)))))))))))))))...))))((.(((((....((.....(((((.((((........((((((...((((....)))).....))))))(((((...((((((((((.....(((....((((((..((((((((((.((((...))))..)).)))).))))
...))))))................)))....))))))))))...))))).......((((((.....))))))..(((((((..(((((((.......))).))))..)))))))..)))).)))))....))..))))).))))))))).......)))).))))..)))))).....)))))))))).)))))))))
)))((((((((((.........))))))))))..))).))))).......)))).)))))))).)))..)))))..)))))))))))).))))))))))...)))).)))))))).)))).))))......(((..(((((...........))))))))......((((((((((....)))))))))).((((((((.
....)))))))).....)))))))))))))....(((..((((....))))..)))))).))))))))))))))))))))..))))))....)))))).))).))))..))...)))..))))))))).)).....)))))))...))...)).))))).)))))).(((((((.....)))))))...(((((.....)
))))......((((.((((((((.....................((.(((((.((((......))))))))).)).((((((.((.....((..((.(((((((....))))))).))..)))))))))))).))))))))))(((((((((((((((((((((((((((..........((((((((......((((((
(((....((((.........)))).......)))))))))...))))))))))))))))..(((((.(((((((.................(((((((((((.........((((....))))(((.((((............)))).)))((((........))))....)))))))))))))))))).)))))....(
(((.......))))))))))))).))).)))))))...(((((...))))).....)))))))..)))))......))))))................))))))).((((((.........))))))((((((.(((((.....((((...))))..)))))..))))))...........((((....)))))))))).
.))).....................
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-675.19] kcal/mol.
2. The frequency of mfe structure in ensemble 1.57137e-35.
3. The ensemble diversity 613.71.
4. You may look at the dot plot containing the base pair probabilities [below]
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
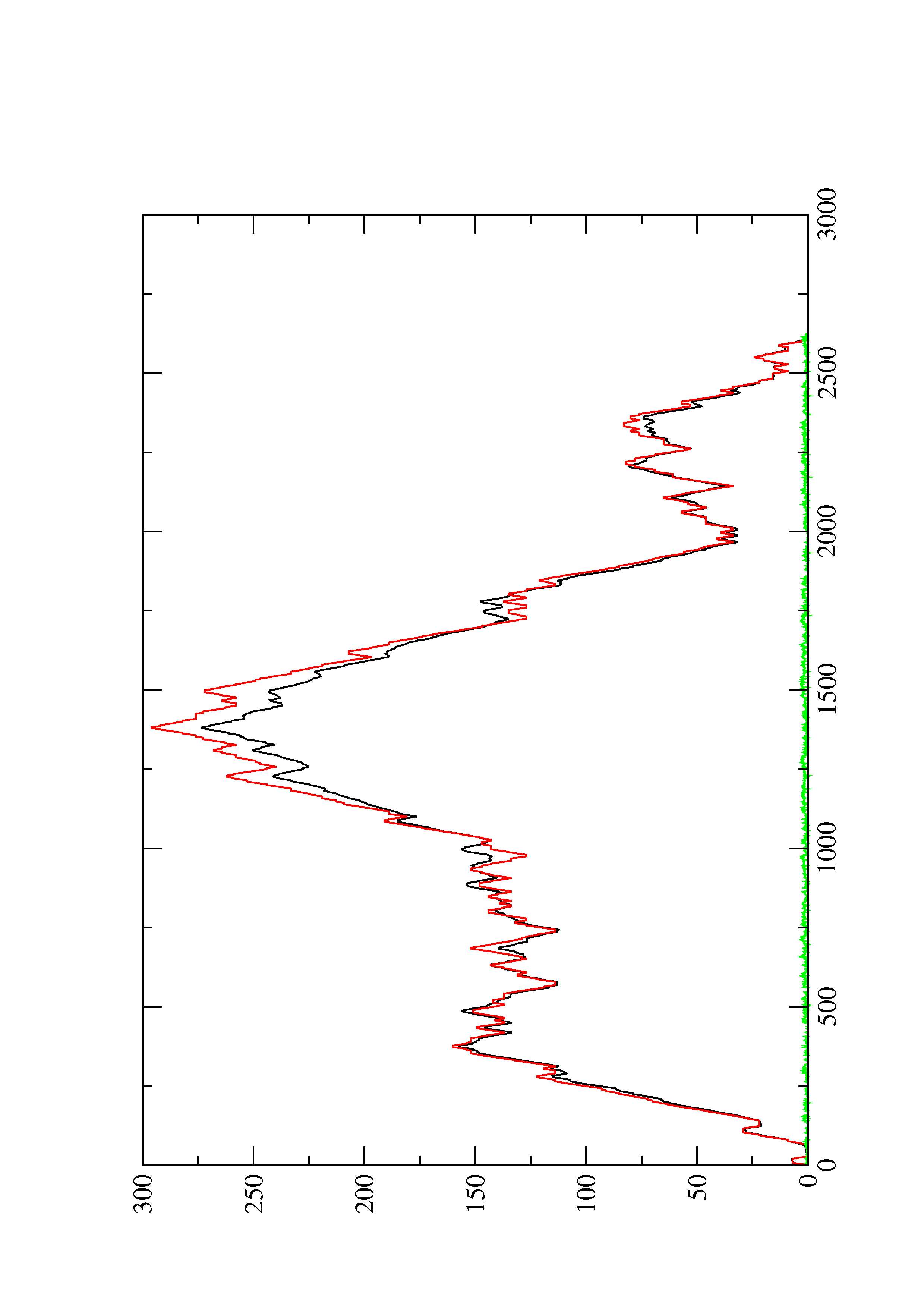
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.