lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01043443.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-1633.00) is given below.
1. Sequence-
AAAAAAUCUCAAAAACAAGUAAAAGAAGAAGAAUCUUCUAUCUUUUCUAAAUUAGAAAAG
AUGAUUAAAAUUAAAAAUCCUCAUGCUACCGAGGAAGAAUUAAUGACAGCCUAUUUGGAC
AAAAUAAAAAAUGAUUUUGUGAAAAAUUUCAAAGGCAAAAUUAAUGUCAGCCCUUAUUCG
GAAGGUUCAAGUACUUCAAUGAAGGGAGAUGAUUAUUGUCUAGAAGGUGAAUCACAACUA
GUGGAAGAACCAACUACUAAAGACCUAUUCGACAGUCUUAUUAAAGCCUUAGUGCAAAAA
AUCCAGGCAAGAAAAGAUCAUACUUCUUCUUCCAAGGGGAAAGAUAAAAUGUCUGAGUAA
GUAUUCUACUCAGAAAAAUUAUUGAAGAAGACAAAUCUGUCAUUUUCUUUUAUAAAGAAA
ACGCAGCAAGAAACAGCAAAAAGAGGAAAGCAAGACGAAACGUGCGGGAAAAAGAAAGAU
CGUUGUCAGUUUUAAUAAAGCAAUGGAUCCCAUGACUUUUGCAAAAGAAAAACUUUAAUA
AAGUUUUACUGUUUUCUUUUGAAAAAUAGAAAAAGAUGUCGGCCAUUGUAAAAGUUUAGC
CGACUAUGUAUGUCAUUUUCUUUCUUAGGCAGAAUGAUUCCACCGCCUAAGUGAGGCUAU
AUAAGGAAGCCUUGUAGUUCAGCUGUAACCAAGCUUCCAAAAAUUUCAGUUGUAAAAAUU
UUCUAUCAAUAAUAUAUUUCAGUAGUUUUCCAGUAUCCACAACACUGUUCUACAAAUAUU
GUAAGUAUUUAUUUCUUUUUUAUUUUAUUAUAAAUUUAAAUUUGUGUCUAGUAUUAUAAU
UAUGUCAGGCUAAUAGUCUGAUUGUCUAAUUUAUUCUGAGAAUUUUGGUCCUGUAUUAUU
GCAGUAUGUUAGUAAUAGCAUCACAUAAACAUUUAUAGUAAUUAUUUAAUAUGAGUAUUA
ACUAGCUAAAAUUUUUAUAUAAGGAUGAUACAGUAAUUAUAAUGGGUAUUGAAUAAUUGA
AAUAGUAACACAAUUGCCGCACAGUGCCGUUGUUAUGCCGUUUAAGCCGUUAAUGAUAUC
UAGGCGUUUAGUCAUGUUUUGGUCAUUUGUGUUGGUCUAGUUACUGUUCUUAUUAAUUUA
AUGUAAAUCCCAUUUUAUAUAAUUACUGCUAGAGCCUGAUAUAAUAAUUUUACUGGUUUA
AGUAAAUUCCCAUAUUUAAUGUAAAUGUAUUAUGUCUGGUGUUAUUACGGCUGCUCCGUG
AUGGUAUCAGGACCAAAAUUCUCAGAAUAAAUUAGACAAUCAGACUAUUAGCCUGACAUA
AUUAUAAUACUAGACACAAAUUUAAAUUUAUAAUAAAAUAAAAAAGAAAUAAAUACUUAC
AAUAUUUGUAGAACAGUGUUGUGGAUACUGGAAAACUACUGAAAUAUAUUAUUGAUAGAA
AAUUUUUACAACUGAAAUUUUUGGAAGCUUGGUUACAGCUGAACUACAAGGCUUCCUUAU
AUAGCCUCACUUAGGCGGUGGAAUCAUUCUGCCUAAGAAAGAAAAUGACAUACAUAGUCG
GCUAAACUUUUACAAUGGCCGACAUCUUUUUCUAUUUUUCAAAAGAAAACAGUAAAACUU
UAUUAAAGUUUUUCUUUUGCAAAAGUCAUGGGAUCCAUUGCUUUAUUAAAACUGACAACG
AUCUUUCUUUUUCCCGCACGUUUCGUCUUGCUUUCCUCUUUUUGCUGUUUCUUGCUGCGU
UUUCUUUAUAAAAGAAAAUGACAGAUUUGUCUUCUUCAAUAAUUUUUCUGAGUAGAAUAC
UUACUCAGACAUUUUAUCUUUCCCCUUGGAAGAAGAAGUAUGAUCUUUUCUUGCCUGGAU
UUUUUGCACUAAGGCUUUAAUAAGACUGUCGAAUAGGUCUUUAGUAGUUGGUUCUUCCAC
UAGUUGUGAUUCACCUUCUAGACAAUAAUCAUCUCCCUUCAUUGAAGUACUUGAACCUUC
CGAAUAAGGGCUGACAUUAAUUUUGCCUUUGAAAUUUUUCACAAAAUCAUUUUUUAUUUU
GUCCAAAUAGGCUGUCAUUAAUUCUUCCUCGGUAGCAUGAGGAUUUUUAAUUUUAAUCAU
CUUUUCUAAUUUAGAAAAGAUAGAAGAUUCUUCUUCUUUUACUUGUUUUUGAGAUUUUUU
2. MFE structure-
((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((
((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((
((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((
((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((
((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((((.(((.((((((((((((((((((((((((((.(((.(((..............((((((.(((((((.((((((.(((....((((((((((((
(((((((((((((((((...(((((((((......((((((((((((((....((((........(((..............))).......))))...))).)).))))).))))...)))))))))...))).)))))))).....)))))....))))))))))).))...))).)))))).)))))))))))))..
...........))).))).))))))))).))))))..))))))))))))))))))..))))))))).)))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))
))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))
))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))
))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))
))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))))
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-1641.24] kcal/mol.
2. The frequency of mfe structure in ensemble 1.56109e-06.
3. The ensemble diversity 40.57 .
4. You may look at the dot plot containing the base pair probabilities [below]

III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
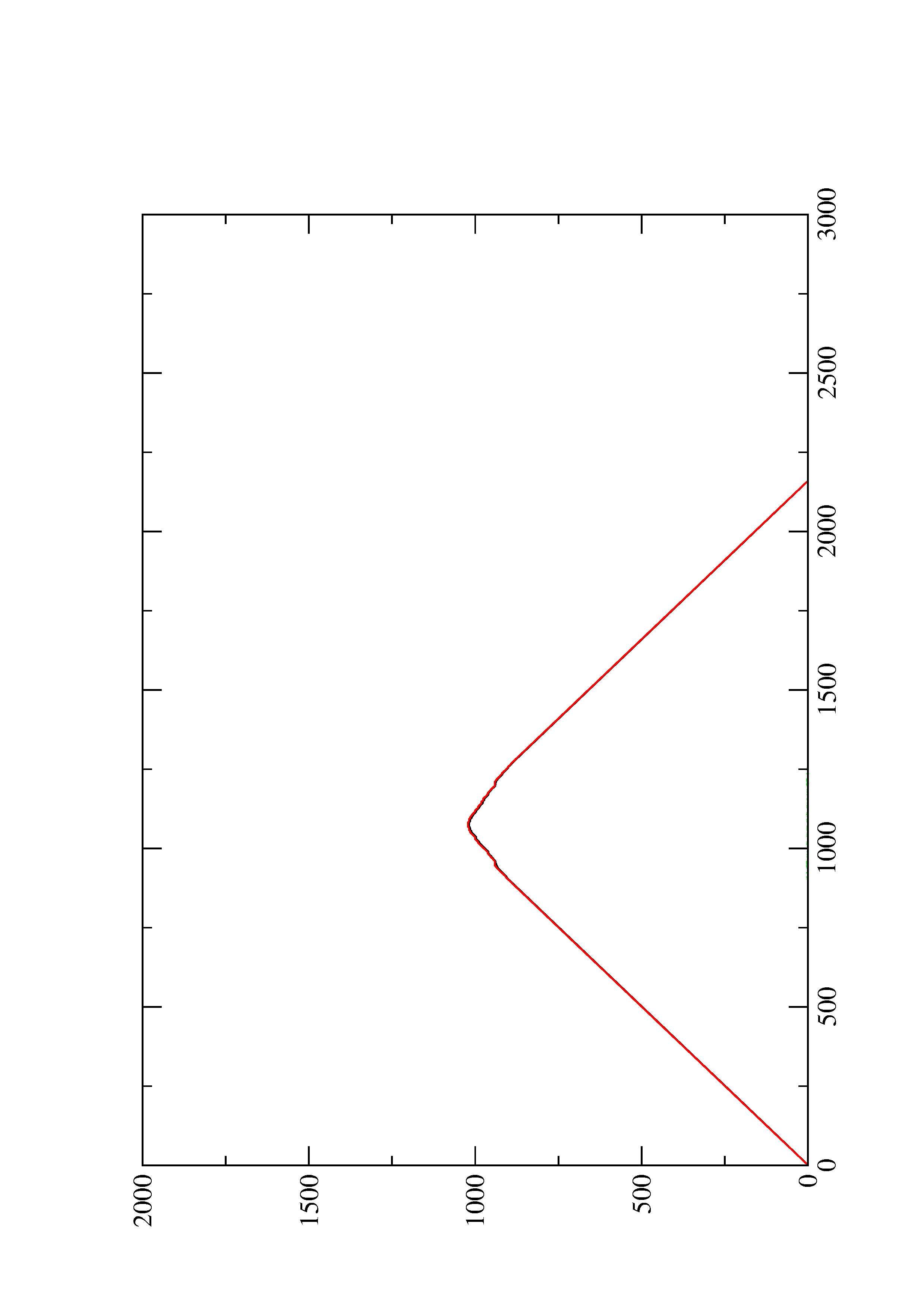
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.