lncRNA Structure Prediction
Species Name : Withania somnifera Sequence Id : GBHJ01043911.1
I. Result for minimum free energy prediction
The optimal secondary structure in dot-bracket notation with a minimum free energy of (-579.50) is given below.
1. Sequence-
CCAUUGCGUCCGGAAUGCCAUUUCCAAUGUGUUUGCAUAUUUGGUUUGUCAAAAUUGGCU
UCCGUUUGGUCCCCGAGACGGCUGGUGCGUUGGCUGAUUUUUCAGCUAAAAACAGAAAAA
ACAGCAGCCAGUUCUGUUUUAUAAGUUGCAGCUUCACUGUUUUCUCAUUUCUAAAAUCCC
CAAAUCAGUUUCUUGCCUUCCUCUCCCAAAAACAAAAAUUCCUCCAACCUUCCUUCCAAG
UUCUAAGUCAAAUUUCAACCUUCUUCCAUGUCGUUGUUGUCUAAGAUCUUGGUAAGUUCC
CUUCCUUCUUUCUUUUCUCUUAUUUUUCUCUUUCAUGAUUUCCAUCUAGGGUUGAUGGAA
UAUCAAUUGUUAGUAGUUUUCUUCAUUUAAUUCUUGUUUAUUAGCUUAGUUUUGGUUCCU
AGAGAUUGAAUCUCUUCUGAAUUACGAUUGGUUAUGGCGUAUUUAAUAAUUCUUUAAGCC
CUAUUUUUUGUUUUCUUUCCGGGCUGUUUUAUCGUUUUUUCGUUGCUCGCUUUGGGCUCA
AUUUUGGCACAUAGGUUCCUAGUUGUGUUAGGAACCUCGUGGUGAAGUUUGUUUUAGGUU
UCGGCGAGCGGGGCCCGUAAAGCCCGUUUUAGGUCCAGUACAGAAUUCGUUAUAAAUUGC
AUCAUUGGAUUCGUAUUGACGUUUGGAAUGUAUUGGUGCAUAUUUCAGAUCAUUCAGAGC
UCGUUCGACGUUAGGCAGAAUCAUAUUUCUGGUUUUUCUCAAAGCUUUGUCGUCGGACGA
CAGGUAGGUUCCUUGGGCUAUAAUCUGGGCUUAUAGCCUUAAUAAUAGUACUGAAUAGCA
UCCUCGGGUUGGGUAGCUAAUUCGUAUAGUAUCCCUGUGCCUUCUUCCAUACGCUAAUUA
UUGAUACGCUUAUGUUGGUACCUGUUUGAUACGGGACCAGGACUUAGUAUUUGUUCAGUU
GCCUGUGCAGGACAUAGGUUGUGAUAAUGGUAUGAUAUUAUGUGGUAUAUGGUUAUAUGU
UGCUUCUUUCUGUGCAUUCAUGAAUCGGAGAGACCGAGCUCAUAGCUCCGAUGAGAGUGC
AUUGGGGCAGAUUGAUUAUGAGCCUUGAAGGCUAGUGGUAUGAUUCAUGUUAUGAGCACC
AAGGGCUAAUGGUGAUUAUUGUGAUAAAAAUGAGUUAUUUAGUAAUUAAUAAUAACAUAA
GUAAUGUUUUUACUGAUUUAGUCUCGAUAUGGUAUCGAGCACCCGGUUAUAUUAUUUUUA
CUCGUUUUAGUCCAAGGAUUCAUUCUUGGCACCGGGAUUAUUCAUAUACAUGUUUUUAUA
ACUAAUUACUGGCACUUGAGUCUGGUUCUCUUCGACAGUGAGUACAGCGCUGGGACUUGA
GACCGUCGUCGUACGGCAGUGAGUACAGCUUCACAGAUAUUGACUUGUGGACCAUAGGUG
AUACUACCAGACUGAUGAUACAGAUUGAUAAGUUAUGAGAUUAUGAUUAGCGUGAUGACA
CUGUGAUAUUGUGAUGAGAUGCUACGGCACGUUGAUAUUGAAAUGAGAUGUUACGGCACG
AGGUGCCAGUUUGUACUGUAUCUGGGGAAGUGAGGUUCCUCCGACGAUCAUGUGUUUGUU
ACUUUCCGCACUUGUUUUCUUCAGUGUACCCCAGGUAUAAUGGCUCUGCCUGCGGGUGAG
CGUGAAUUUUGUGUAACGCACCAAGCGUUUAUUAGAAUUAUUUAUUUAUUUAUUUUACUG
CUUUCUAGUUAUUUAUUGUUAAAUGCUACUUCAUGAUAUGUGAGAUGAAUCAGUGCCUAG
UAUCAUUGCUUUCUCAAUUUCUCCUAUCCCUUGAGGUUACCGGUAUACCGCCUAUAUUCA
AUUCUCUAUCUUCCUCGAUUCCUAUUCUUGAUUGAGUUAUAUGUCACAUUUAUUGUUGCG
AGGAUGCCAGGUAUGUUCCUUUUUGUAACCUGUGAUCCUAGUAUGUGAUUGGCUAGCAUG
AUCUGUUAUCGUGACUUGGCUGUUUUAGGAUGCUAGCUUCCCUGAUUGUCCUAUUUACUU
UUGUGACUAUAAUUCUUGAUCUCCGUUAUUUACUAUUGUUAUUUUAGUUAUGUGCUCAGU
GGGUCCUCGAAACCUACUGAGUACAGGUUGCUUCGGCGGCUUGUACUC
2. MFE structure-
.....((.(((((((.((((...((((((((....)))).))))...........)))))))))...((....(((((.(((((((...((((((((....)))))))).....((.(((((((.(((.....((........)).....)))...))))))).)).................))))))))))))....)
).......((((((((((..............((((((((.(((.((.(((....((((.............))))))).)).))).))))).))).......................................(((((((((((.......))))))).))))...((((((((.....................)))
)))))((((((.((((((..((((((...))))))...)))))).))))))....(((...................)))....))))))))))((((.((((((.................(((((((((..((((((((.(((.((.(((((((((((((.....)))))))))).))))).))).))).....))))
).))))))))))))))).)))).((......))..((((((.(((((((................)))))))))))))..(((((((((((........)))))))))))......))))((((((((((..(((((((((((......))))))).......))))...))))))))))..((((((((....(((((.
....((((((.((((((...((((((((((.((((((((..(((((((..((((((.....((((((((((((...(((...((.((...(((((((((((.(((...(((((.(((((.(((((.....))))))))))((((((..(((((((((((((((((((.....)))))))....((((((......)))))
)(((((((((((((...............((((((..((((...((((((((((((.((((((((((((((((......)))))))))........)))))))(((((..(((((((((((((..(((((((((((..((..(((((((.(((((...(((.(((.(((((((((.....((((((.....(((((...(
((((.....)))))......(((((((((...))))))).))...)))))))))))...))))))))).))))))....))))).))))).)).))....)))))).)))))...)))).....)))))))))..)))))..))).)))))))))..)))))))))).((((..(((((...((((......))))...)
)))).))))..(((((........)))))))))))).....))))))..))))))....))))))...))))))))))))))))))))))))))).)))))...)))))))).))))(((((((.(((((..(((((((.......))))))).(((((...))))).........((((((((((((((((.((.....
..(((((........))))).....)).)))))...............)))))))))))...((((.(((....)))))))...(((((((...(((((.....)))))...)))))))........(((((((((((........(((((((((....))))))..))).........)))))))))))..))))))))
))))((((......)))).....)))))))))))))....(((....))).................(((......)))..........((((((((((((.((((((........(((.(((((..((((((((........))).)))))..))))).))).......((((((((((((.....)))))).)).)))
)....(((((((.(((.....)))..)))))))........))))))))))))))..))))...)))))))).))))))))))...)))))).))))))..)))))....))))))))(((((((((((((....)))))))))))))
You can download the minimum free energy (MFE) structure in
II. Results for thermodynamic ensemble prediction
1. The free energy of the thermodynamic ensemble is [-617.35] kcal/mol.
2. The frequency of mfe structure in ensemble 2.11723e-27.
3. The ensemble diversity 430.86.
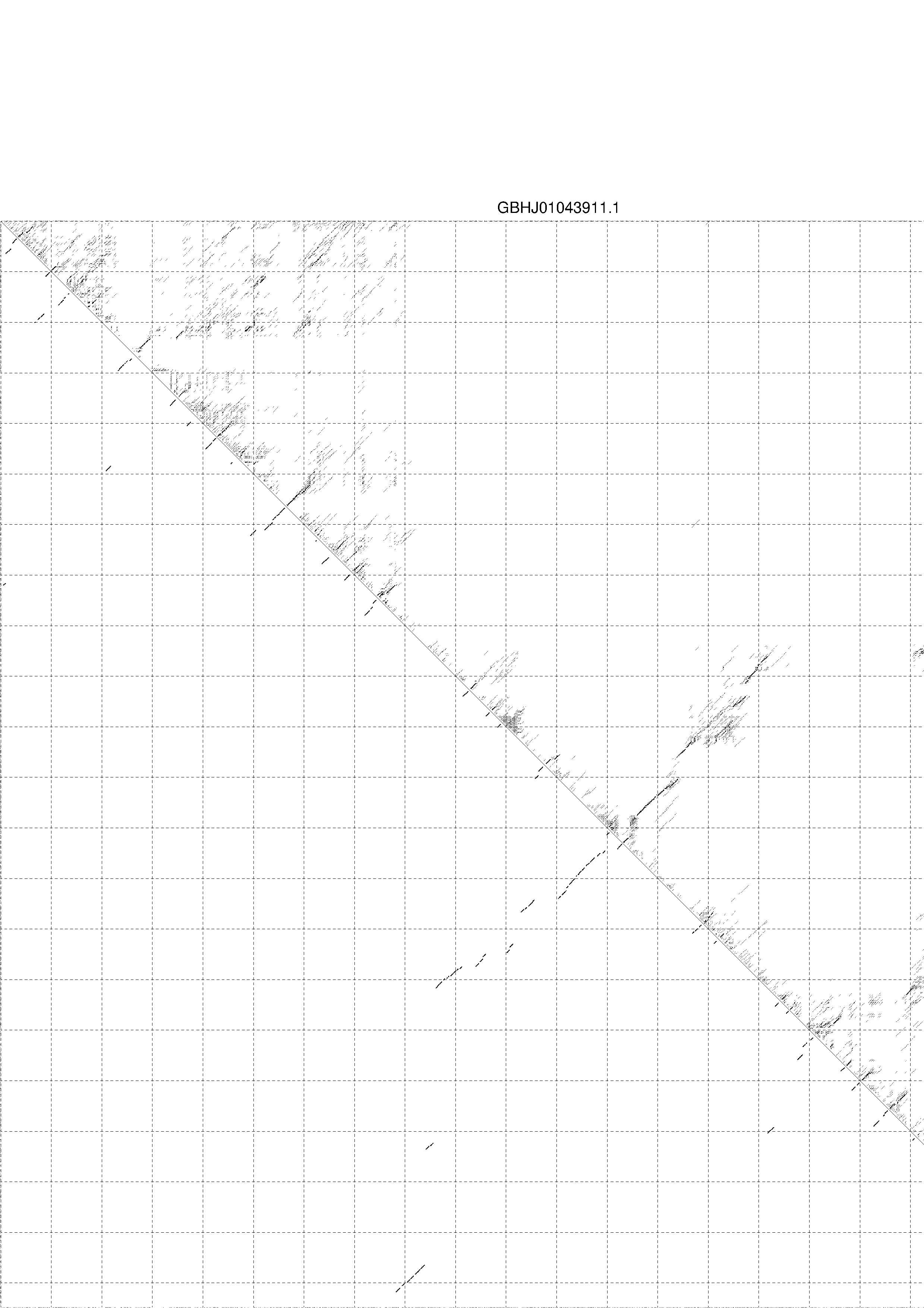
4. You may look at the dot plot containing the base pair probabilities [below]
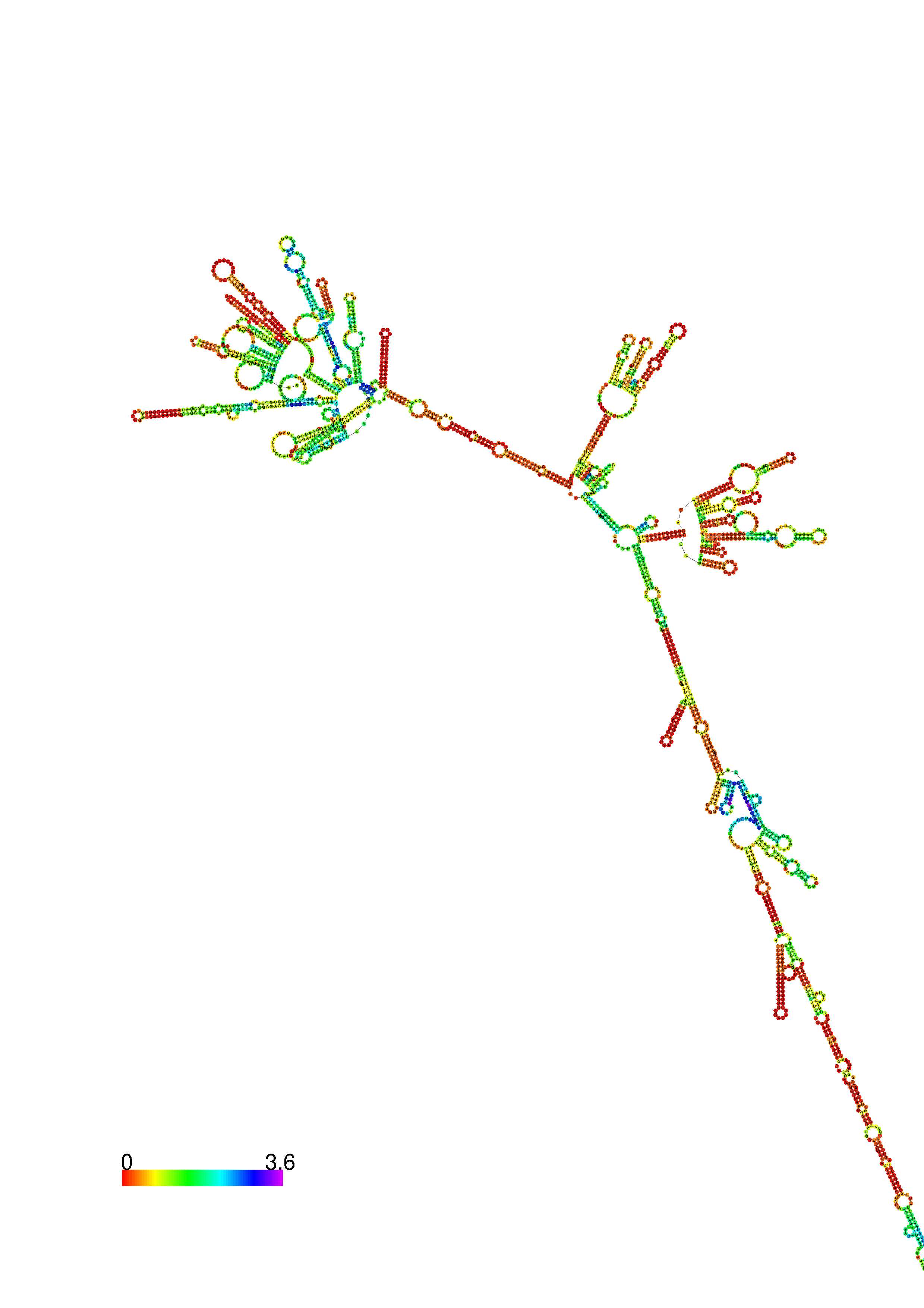
III. Graphical output
1. A note on base-pairing probabilities:--
The structure below is colored by base-pairing probabilities. For unpaired regions the color denotes the probability of being unpaired. The reliability information of RNA secondary structure
plot in the form of color annotation, Positional entropy ranging from red (low entropy, well-defined) via green to blue and
violet (high entropy, ill-defined).
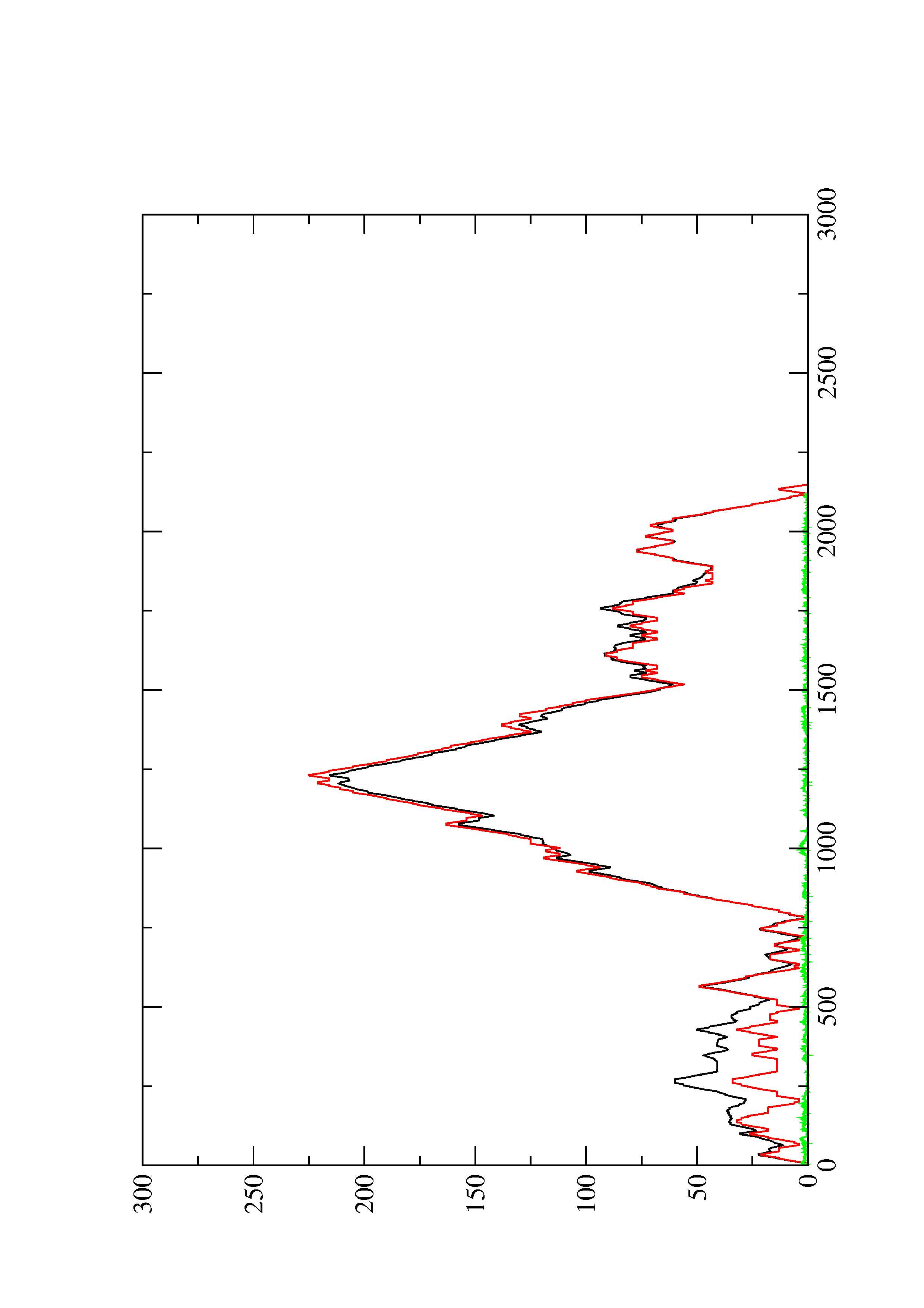
2. Here you find a mountain plot representation of the MFE structure, the thermodynamic ensemble of RNA structures, and the centroid structure. Additionally we present the positional entropy for each position. The resulting plot shows three curves, two mountain plots derived from the MFE structure (red) and the pairing probabilities (black) and a positional entropy curve (green). Well-defined regions are identified by low entropy.